آنزیم: تفاوت میان نسخهها
←جایگاه فعال: اصلاح ارقام |
|||
| خط ۱۰۲: | خط ۱۰۲: | ||
=== جایگاه فعال === |
=== جایگاه فعال === |
||
{{اصلی|جایگاه فعال}} |
{{اصلی|جایگاه فعال}} |
||
جایگاه فعال ناحیهای از آنزیمی است که در آن مولکولهای [[بستر (شیمی)|بستر]] به هم متصل می شوند و تحت [[واکنش شیمیایی]] قرار می گیرند. جایگاه فعال متشکل از باقیمانده [[اسید آمینه]] است که پیوندهای موقتی با بستر ( محل اتصال ) و باقی مانده ای ایجاد می کند که واکنش را کاتالیز می کند. <ref>{{Cite journal|last=Srinivasan|first=Bharath|date=2020-09-27|title=Words of advice: teaching enzyme kinetics|url=http://dx.doi.org/10.1111/febs.15537|journal=The FEBS Journal|doi=10.1111/febs.15537|issn=1742-464X}}</ref> اگرچه جایگاه فعال فقط ۱۰-۲۰٪ از حجم آنزیم را اشغال می کند ، {{Rp|19}} اما مهمترین بخش در ساختار یک آنزیم است زیرا مستقیماً واکنش [[واکنش شیمیایی|شیمیایی]] را کاتالیز می کند . این ماده معمولاً از سه تا چهار اسید آمینه تشکیل می شود ، در حالی که سایر اسیدهای آمینه موجود در پروتئین برای حفظ ساختار سوم آنزیم مورد نیاز است. |
|||
;[[:en:Active site]] |
|||
هر جایگاه فعال تکامل یافته است تا برای اتصال یک بستر خاص و کاتالیز یک واکنش خاص بهینه شود و در نتیجه ویژگی بالایی داشته باشد . این ویژگی با آرایش اسیدهای آمینه در جایگاه فعال و ساختار بسترها تعیین می شود. گاهی اوقات آنزیم ها نیز برای انجام عملکرد خود نیاز به اتصال با برخی از [[کوفاکتور (زیستشیمی)|کوفاکتورها]] را دارند. جایگاه فعال معمولاً یک شیار از آنزیم است که می تواند در یک تونل عمیق درون آنزیم قرار گیرد ، <ref name="Pravda">{{Cite journal|displayauthors=3|vauthors=Pravda L, Berka K, Svobodová Vařeková R, Sehnal D, Banáš P, Laskowski RA, Koča J, Otyepka M|date=2014|title=Anatomy of Enzyme Channels|journal=[[BMC Bioinformatics]]|volume=15|pages=379|doi=10.1186/s12859-014-0379-x|pmc=4245731|pmid=25403510}}</ref> یا بین رابط های [[ساختار چهارم پروتئین|آنزیم های چند مولتی]] قرار گیرد . یک جایگاه فعال می تواند یک واکنش را به طور مکرر کاتالیز کند زیرا باقیمانده ها در پایان واکنش تغییر نمی کنند (ممکن است در طول واکنش تغییر کنند ، اما تا پایان دوباره تولید می شوند). این فرآیند با کاهش [[انرژی فعالسازی|انرژی فعال سازی]] واکنش حاصل می شود ، بنابراین بسترهای بیشتری انرژی کافی برای انجام واکنش دارند. <ref>{{Cite journal|last=Srinivasan|first=Bharath|date=2020-09-27|title=Words of advice: teaching enzyme kinetics|url=http://dx.doi.org/10.1111/febs.15537|journal=The FEBS Journal|doi=10.1111/febs.15537|issn=1742-464X}}</ref> |
|||
== مکانیسم == |
== مکانیسم == |
||
نسخهٔ ۱۸ دسامبر ۲۰۲۰، ساعت ۱۹:۵۳
| بخشی از مجموعه مباحث دربارهٔ |
| بیوشیمی |
|---|
 |
زیمایهها[۱] یا آنزیمها (به فرانسوی: enzyme) پروتئینهایی هستند که به عنوان کاتالیزورهای بیولوژیکی[a] عمل میکنند. کاتالیزورها سرعت واکنشهای شیمیایی را افزایش میدهند. مولکولهایی که ممکن است آنزیمها روی آن عمل کنند سوبسترا نامیده میشوند و آنزیم بسترها را به مولکولهای مختلفی که به عنوان فراورده معروف هستند تبدیل میکند. تقریباً تمام فرایندهای متابولیک موجود در سلول نیاز به کاتالیز آنزیم دارند تا به سرعت کافی انجام شود تا زندگی ادامه یابد.[۲] : 8.1 مسیرهای متابولیک به کاتالیز مراحل فردی آنزیمها بستگی دارند. مطالعه آنزیمها که به عنوان آنزیمشناسی[b] نامیده میشود و در زمینه جدیدی به نام تجزیه شبهآنزیمی رشد یافتهاست، با درک اینکه در طول تکامل، برخی از آنزیمها توانایی انجام کاتالیز بیولوژیکی را از دست دادهاند، که غالباً در توالی اسیدهای آمینه آنها و خصوصیات غیرعادی «شبه کاتالیستی» منعکس میشود.[۳][۴]
آنزیمها در بیش از ۵۰۰۰ نوع واکنش بیوشیمیایی به عنوان کاتالیزور شناخته شدهاند.[۵] سایر بیوکاتالیستها مولکولهای RNA کاتالیزوری هستند که ریبوزیم نامیده میشوند. ویژگی آنزیمها از ساختارهای سه بعدی منحصر به فرد آنها ناشی میشود.
مانند همه کاتالیزورها، آنزیمها با کاهش انرژی فعالسازی، سرعت واکنش را افزایش میدهند. بعضی از آنزیمها میتوانند با تبدیل خود به بستر موجب تولید میلیونها بار سریعتر محصول شوند. اوروتیدین ۵-فسفات دکربوکسیلاز[c] موجب میشود واکنشی که در نبود این آنزیم میلیونها سال به طول میانجامد در چند میلیثانیه رخ دهد.[۶][۷] از نظر شیمیایی، آنزیمها مانند هر کاتالیزوری هستند و در واکنشهای شیمیایی مصرف نمیشوند و تعادل یک واکنش را تغییر نمیدهند. آنزیمها با ویژگیهای خاصی نسبت به سایر کاتالیزورهای دیگر متفاوت هستند. فعالیت آنزیم را میتوان تحت تأثیر مولکولهای دیگر قرار داد: بازدارندهها مولکولهایی هستند که باعث کاهش فعالیت آنزیم میشوند و فعالکنندهها مولکولهایی هستند که فعالیت را افزایش میدهند. بسیاری از داروهای درمانی و سموم مهارکننده یا بازدارنده آنزیم هستند. فعالیت آنزیم بهطور قابل توجهی خارج از دمای و pH مطلوب آن کاهش مییابد و بسیاری از آنزیمها هنگام قرار گرفتن در معرض گرمای بیش از حد (پایدار) واسرشته یا دناتوره میشوند و ساختار و خواص کاتالیزوری خود را از دست میدهند.
بعضی از آنزیمها بهصورت تجاری بهطور مثال در سنتز آنتیبیوتیکها استفاده میشوند. برخی از محصولات خانگی برای سرعت بخشیدن به واکنشهای شیمیایی از آنزیمها استفاده میکنند: آنزیمهای موجود در پودرهای شستشوی بیولوژیکی پروتئین، نشاسته یا لکههای چربی روی لباسها را تجزیه میکنند و آنزیمهای موجود در تردکنندههای گوشت پروتئینها را به مولکولهای کوچکتر تجزیه میکنند و باعث میشود گوشت راحت تر جویده شود.
تاریخچه

در اواخر سده ۱۷ و اوایل سده ۱۸، هضم گوشت توسط ترشحات معده[۸] و تبدیل نشاسته به قند توسط عصارههای گیاهی و بزاق شناخته شد اما مکانیسمهایی که به موجب آن اتفاق میافتد مشخص نشده بود.[۹]
شیمیدان فرانسوی، آنسلم پین اولین کسی بود که در سال ۱۸۳۳ آنزیم، دیاستاز را کشف کرد.[۱۰] چند دهه بعد، هنگام مطالعه تخمیر قند به الکل توسط مخمر، لویی پاستور نتیجه گرفت که این تخمیر توسط نیروی حیاتی موجود در سلولهای مخمر به نام «تخمیر» (ferments) ایجاد شدهاست، که تصور میشد فقط در موجودات زنده وجود دارد. وی نوشت: «تخمیر الکلی عملی است که با زندگی و سازماندهی سلولهای مخمر ارتباط دارد، نه با مرگ یا قرارگیری سلولهای مخمر.»[۱۱]
در سال ۱۸۷۷، ویلهلم کوهن (فیزیولوژیست آلمانی) برای اولین بار از اصطلاح آنزیم، که از واژه یونانی ἔνζυμον، به معنای «خمیر» یا «در خمیرمایه» است، برای توصیف این فرایند استفاده کرد.[۱۲] بعداً واژه آنزیم برای اشاره به مواد غیرزنده مانند پپسین مورد استفاده قرار گرفت و از کلمه تخمیر برای اشاره به فعالیتهای شیمیایی تولید شده توسط موجودات زنده استفاده شد.[۱۳]
ادوارد بوخنر اولین مقاله خود را در مورد مطالعه عصارههای مخمر در سال ۱۸۹۷ ارائه کرد. در یک سری آزمایشها در دانشگاه هومبولت برلین، وی دریافت که شکر توسط عصاره مخمر حتی وقتی سلول مخمر زنده در مخلوط وجود ندارد تخمیر میشود[۱۴] او آنزیمی را که تخمیر ساکارز را به وجود آورد، " زیماز " نامید.[۱۵] وی در سال ۱۹۰۷ به دلیل «کشف یک روش تخمیر بدون سلول زنده» جایزه نوبل شیمی را دریافت کرد. به دنبال نمونه بوخنر، معمولاً آنزیمها مطابق واکنشی که انجام میدهند به صورت ترکیب پسوند -از با اضافه بستر، برای نمونه، لاکتاز آنزیمی است که لاکتوز را جدا میکند یا بسته به نوع واکنش مانند، DNA پلیمراز که پلیمرهای DNA را تشکیل میدهد نامگذاری شدهاند.
هویت بیوشیمیایی آنزیمها در اوایل دهه ۱۹۰۰ ناشناخته بود. بسیاری از دانشمندان مشاهده میکردند که فعالیت آنزیمی با پروتئین همراه است، اما دیگران (مانند برنده جایزه نوبل، ریچارد ویلستر) برا این باور بودند که پروتئینها صرفاً حامل آنزیمهای واقعی هستند و پروتئینها به خودی خود قادر به کاتالیز نیستند.[۱۶] در سال ۱۹۲۶، جیمز ب. سامنر نشان داد که آنزیم اورهآز پروتئین خالص است و موجب بلوری شدن آن میشود. او همچین در سال ۱۹۳۷ برای آنزیم کاتالاز نیز همین ترتیب را انجام داد. نتیجهگیری که پروتئین خالص میتواند همان آنزیمها باشند بهطور قطعی توسط جان هاوارد نورثروب و وندل مردیت استنلی، که در سال ۱۹۳۰ روی آنزیمهای گوارشی پپسین، تریپسین و کیموتریپسین کار میکردند نشان داده شد. این سه دانشمند جایزه نوبل شیمی را در سال ۱۹۴۶ دریافت کردند.[۱۷]
این کشف که آنزیمها میتوانند متبلور شوند، درنهایت باعث شد که ساختارهای آنها با بلورشناسی پرتو ایکس حل شود و نخستین بار برای لیزوزیم، آنزیمی که در اشک، بزاق و سفیده تخممرغ یافت میشود و پوشش برخی از باکتریها را هضم میکند انجام شد. این ساختار توسط گروهی به سرپرستی دیوید چیلتون فیلیپس حل و در سال ۱۹۶۵ منتشر شد.[۱۸] ساختار با وضوح بالا ی لیزوزیم آغازگر ایجاد زمینه زیستشناسی ساختاری و تلاش برای درک نحوه عملکرد آنزیمها در سطح اتمی جزئیات بود.[۱۹]
نامگذاری
نام آنزیم اغلب از زیر لایه آن یا واکنش شیمیایی که آن را کاتالیز میکند، گرفته میشود، با واژهای که به صورت -ase در انتها تمام می شود.[۲] : 8.1.3 نمونههای آن شامل لاکتاز، الکل دهیدروژناز و DNA پلیمراز است. آنزیمهای مختلفی که همان واکنش شیمیایی را کاتالیز میکنند، ایزوزیمز[d] نامیده میشوند. : 10.3
اتحادیه بینالمللی بیوشیمی و زیستشناسی مولکولی برای نامگذاری آنزیمها، عدد گروه آنزیم (شماره EC) را ایجاد کردهاست. هر آنزیم توسط دنباله ای از چهار عدد پیش از "EC" که مخفف "کمیسیون آنزیم" است توصیف میشوند. شماره اول آنزیم را بهطور گسترده بر اساس مکانیسم آن طبقهبندی میکند.[۲۰]
طبقهبندی سطح بالا:
- EC 1، اکسیدوردوکتاز: کاتالیز کردن واکنشهای اکسایش-کاهش
- EC 2، ترانسفراز : انتقال یک گروه عاملی (به عنوان مثال یک گروه متیل یا فسفات)
- EC 3، هیدرولاز: هیدرولیز پیوندهای مختلف را کاتالیز میکند
- EC 4، لیاز: پیوندهای مختلفی را به غیر از هیدرولیز و اکسیداسیون قطع میکند.
- EC 5، ایزومرازها: کاتالیز تغییرات ایزومریزاسیون در یک مولکول واحد را انجام میدهد.
- EC 6، لیگاز: به دو مولکول با پیوندهای کووالانسی میپیوندد.
این بخشها با سایر ویژگیها از جمله بستر، محصولات و مکانیسم شیمیایی تقسیم میشوند. آنزیم با چهار نام عددی معین کاملاً مشخص میشود. به عنوان مثال، هگزوکیناز (EC 2.7.1.1) یک ترانسفراز (EC 2) است که یک گروه فسفات (EC 2.7) را به یک قند هگزوز، یک مولکول حاوی گروه الکل اضافه میکند (EC 2.7.1).[۲۱]
| گروه | واکنش کاتالیزی | واکنش شیمیایی شماتیک | نمونه |
|---|---|---|---|
| EC 1 اکسیدوردوکتازها |
برای کاتالیز واکنشهای اکسایش-کاهش; انتقال اکسیژن، هیدروژن یا الکترون از یک ماده به ماده دیگر | AH + B → A + BH (reduced) A + O → AO (oxidized) |
لاکتات دهیدروژناز، اکسیداز |
| EC 2 ترانسفرازها |
انتقال گروه عاملی از یک ماده به ماده دیگر | AB + C → A + BC | ترانسآمیناز، کیناز |
| EC 3 هیدرولازها |
تشکیل دو فراورده از یک سوبسترا به وسیله آبکافت | AB + H2O → AOH + BH | لیپاز، آمیلاز، پپتیداز |
| EC 4 لیازها |
واکنش افزایشی یا حذفی با سوبسترا از راههایی جز آبکافت و اکسیداسیون | RCOCOOH → RCOH + CO2 or [X-A-B-Y] → [A=B + X-Y] | دکربوکسیلاز، آلدولاز |
| EC 5 ایزومرازها |
نوآرایی درونمولکولی | AB → BA | ایزومراز، موتاز |
| EC 6 لیگازها |
پیوند میان دو مولکول با ایجاد پیوند کووالانسی و شکستن همزمان ATP | X + Y+ ATP → XY + ADP + Pi | سینتتاز، دیانای لیگاز |
ساختار

آنزیمها بهطور کلی پروتئینهای حلقوی هستند که به تنهایی یا در کمپلکسهای (Protein complex) بزرگتر فعالیت میکنند. دنباله اسیدهای آمینه ساختار را مشخص میکند که به نوبه خود فعالیت کاتالیزوری آنزیم را تعیین میکند.[۲۳] گرچه ساختار عملکرد را تعیین میکند، ولی فعالیت آنزیمی جدید نمیتواند به تنهایی از طریق ساختار پیشبینی شود.[۲۴] ساختارهای آنزیمی وقتی گرم میشوند یا در معرض دناتورانتهای شیمیایی قرار میگیرند آشکار میشوند که واسرشتن نام دارد و این اختلال در ساختار معمولاً باعث از بین رفتن فعالیت میشود.[۲۵]
دناتوراسیون آنزیم معمولاً با دمای بالاتر از سطح طبیعی یک گونه مرتبط است. در نتیجه، آنزیمهای موجود در باکتریهایی که در محیطهای آتشفشانی مانند چشمههای آب گرم زندگی میکنند، به دلیل توانایی عملکرد در دماهای بالا، مورد مصارف صنعتی قرار میگیرند و به این ترتیب واکنشهای آنزیمی کاتالیز شده با سرعت بسیار بالایی امکانپذیر میشوند.
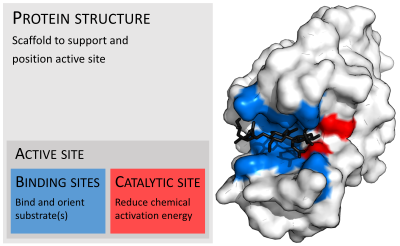
آنزیمها معمولاً بسیار بزرگتر از بسترهای آنها هستند. دامنه اندازه از ۶۲ اسید آمینه باقی مانده برای برای مونومر ۴-تاکسومراز اگزالوکروتونات(4-Oxalocrotonate tautomerase)،[۲۶] تا ۲۵۰۰ باقیمانده در سنتاز اسیدهای چرب (Fatty acid synthase) حیوانات متفاوت است.[۲۷] تنها بخش کوچکی از ساختار آنها (حدود ۲–۴ آمینو اسید) که سایت کاتالیزوری نام دارد بهطور مستقیم در کاتالیز درگیر است.[۲۸] این سایت کاتالیزوری در کنار یک یا چند سایت اتصال دهنده قرار دارد که در آن ماندهها بسترها را جهت میدهند. سایت کاتالیزوریک و سایت اتصال دهنده، سایت فعال آنزیم را تشکیل میدهند. اکثریت باقیمانده ساختار آنزیم به منظور حفظ جهتگیری دقیق و پویایی محل فعال در حضور دارد.[۲۹]
در بعضی از آنزیمها، هیچ اسید آمینه مستقیماً درگیر در کاتالیز نیست. در عوض، آنزیم حاوی سایتهایی برای اتصال و جهتیابی کوفاکتورهای کاتالیزوری است.[۲۹] ساختارهای آنزیمی همچنین ممکن است دارای مکانهای آلوستریک باشد که اتصال یک مولکول کوچک باعث ایجاد تغییر شکل میشود که باعث افزایش یا کاهش فعالیت میشود.[۳۰]
تعداد کمی از کاتالیزورهای بیولوژیکی مبتنی بر RNA به نام ریبوزیمها وجود دارند، که دوباره میتوانند به تنهایی یا در کمپلکس به همراه پروتئینها عمل کنند. رایجترین این ریبوزوم است که از پروتئین و اجزای RNA کاتالیزوری تشکیل شدهاست.[۲] : 2.2
جایگاه فعال
جایگاه فعال ناحیهای از آنزیمی است که در آن مولکولهای بستر به هم متصل می شوند و تحت واکنش شیمیایی قرار می گیرند. جایگاه فعال متشکل از باقیمانده اسید آمینه است که پیوندهای موقتی با بستر ( محل اتصال ) و باقی مانده ای ایجاد می کند که واکنش را کاتالیز می کند. [۳۱] اگرچه جایگاه فعال فقط ۱۰-۲۰٪ از حجم آنزیم را اشغال می کند ، : 19 اما مهمترین بخش در ساختار یک آنزیم است زیرا مستقیماً واکنش شیمیایی را کاتالیز می کند . این ماده معمولاً از سه تا چهار اسید آمینه تشکیل می شود ، در حالی که سایر اسیدهای آمینه موجود در پروتئین برای حفظ ساختار سوم آنزیم مورد نیاز است.
هر جایگاه فعال تکامل یافته است تا برای اتصال یک بستر خاص و کاتالیز یک واکنش خاص بهینه شود و در نتیجه ویژگی بالایی داشته باشد . این ویژگی با آرایش اسیدهای آمینه در جایگاه فعال و ساختار بسترها تعیین می شود. گاهی اوقات آنزیم ها نیز برای انجام عملکرد خود نیاز به اتصال با برخی از کوفاکتورها را دارند. جایگاه فعال معمولاً یک شیار از آنزیم است که می تواند در یک تونل عمیق درون آنزیم قرار گیرد ، [۳۲] یا بین رابط های آنزیم های چند مولتی قرار گیرد . یک جایگاه فعال می تواند یک واکنش را به طور مکرر کاتالیز کند زیرا باقیمانده ها در پایان واکنش تغییر نمی کنند (ممکن است در طول واکنش تغییر کنند ، اما تا پایان دوباره تولید می شوند). این فرآیند با کاهش انرژی فعال سازی واکنش حاصل می شود ، بنابراین بسترهای بیشتری انرژی کافی برای انجام واکنش دارند. [۳۳]
مکانیسم
اتصال بستر
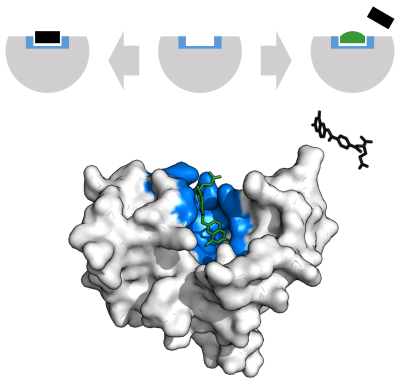
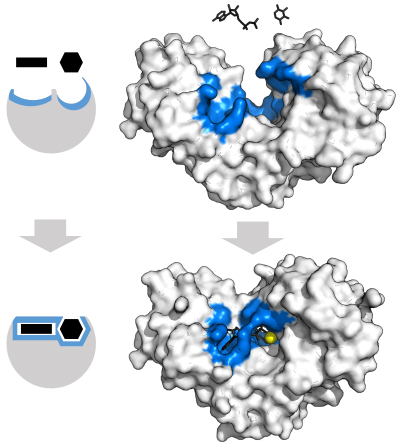
آنزیمها قبل از اینکه بتوانند هرگونه واکنش شیمیایی را کاتالیز کنند، باید بسترهای خود را متصل کنند. آنزیمها معمولاً مشخص میکنند که به چه لایههایی متصل میشوند و سپس واکنش شیمیایی کاتالیز میشود. ویژگی (en:Chemical specificity) محل اتصال (en:Binding site) با شکل مکمل، بار و ویژگیهای آب دوست / آبگریز به لایهها بدست میآید؛ بنابراین آنزیمها میتوانند بین مولکولهای سوبسترا بسیار شبیه از لحاظ شیمی گزینی، جهتگزینی و استریوسپکتیک (en:Stereospecificity) تفاوت قائل شوند.[۳۴]
برخی از آنزیمهایی که بالاترین ویژگی و دقت را نشان میدهند، در کپی و بیان ژنوم نقش دارند. برخی از این آنزیمها مکانیسمهای " نمونهخوانی " (Proofreading (biology)) دارند. در اینجا، آنزیمی مانند DNA پلیمراز در مرحله اول یک واکنش را کاتالیز میکند و سپس در مرحله دوم صحت محصول را بررسی میکند.[۳۵] این فرایند دو مرحله ای منجر به میانگین نرخ خطای کمتر از ۱ خطا در ۱۰۰ میلیون واکنش در پلیمرازهای پستانداران با راستی بالا میشود. : 5.3.1 مکانیسمهای تصحیح مشابه نیز در RNA پلی مراز،[۳۶] آمینواسیل tRNA سنتاز (Aminoacyl tRNA synthetase)[۳۷] و ریبوزوم یافت میشود.[۳۸]
برعکس، برخی از آنزیمها دارای خاصیت گسترده بی نظمی آنزیمی (Enzyme promiscuity) هستند و بر روی طیف وسیعی از لایههای مختلف مرتبط با فیزیولوژیک اثر میگذارند. بسیاری از آنزیمها دارای فعالیتهای جانبی کوچکی هستند که بهطور اتفاقی (نظریه تکامل مولکولی خنثی (Neutral theory of molecular evolution)) بوجود آمدهاند، که ممکن است نقطه شروع انتخاب تکاملی جدید باشد.[۳۹][۴۰]
مدل قفل و کلید
برای توضیح ویژگی مشاهده شده آنزیمها، در سال ۱۸۹۴ هرمان امیل فیشر، شیمیدان آلمانی و برنده جایزه نوبل شیمی پیشنهاد کرد که آنزیم و سوبسترا دارای اشکال هندسی مکمل خاصی هستند که دقیقاً در یکدیگر جای میگیرند.[۴۱] این پیشنهاد اغلب به عنوان مدل «قفل و کلید» نامیده میشود. : 8.3.2 این مدل اولیه ویژگی آنزیم را توضیح میدهد، اما قادر به توضیح ثبات حالت گذار که آنزیمها به دست میآورند نیست.
مدل تناسب القایی
در سال ۱۹۵۸، دانیل کوشلند اصلاحاتی را در مدل قفل و کلید پیشنهاد داد: از آنجا که آنزیمها ساختارهای نسبتاً انعطافپذیر هستند، در اثر فعل و انفعال بستر با آنزیم، سایت فعال بهطور مداوم با تعامل با بستر تغییر فرم میدهد.[۴۲] در نتیجه، بستر به سادگی به یک مکان فعال سفت و سخت متصل نمیشود. زنجیرههای جانبی اسید آمینه که جایگاه فعال را تشکیل میدهند در موقعیتهای دقیق قالببندی میشوند که آنزیم را قادر میسازد تا عملکرد کاتالیزوری خود را انجام دهد. در بعضی موارد مانند گلیکوزید هیدرولازها، مولکول سوبسترا نیز با ورود به محل فعال، اندکی تغییر شکل میدهد.[۴۳] سایت فعال همچنان تغییر میکند تا زمانی که بستر کاملاً بسته شود،
در آن زمان شکل نهایی و توزیع بار تعیین میشود. تناسب القایی ممکن است درستی تشخیص مولکولی را در حضوری رقابتی و نویز از طریق مکانیسم تصحیح ساختاری (Conformational proofreading) افزایش دهد.[۴۴]
کاتالیز
آنزیمها میتوانند واکنشها را از چند طریق تسریع کنند، همه این راههای گوناگون در نهایت انرژی فعال سازی را کاهش میدهند (ΔG ‡، انرژی آزاد گیبس)
- با تثبیت حالت گذار:
- ایجاد یک محیط با توزیع شارژ مکمل به حالت گذار برای کاهش انرژی آن[۴۵]
- با ارائه یک مسیر واکنش جایگزین:
- واکنش موقت با بستر، تشکیل یک واسطه کووالانسی برای ایجاد حالت انتقال انرژی پایینتر
- با بیثبات کردن حالت پایه بستر:
آنزیمها ممکن است بهطور همزمان از چندین مکانیزم استفاده کنند. به عنوان مثال، پروتئازها مانند تریپسین با استفاده از یک سهگانه کاتالیزوری، کاتالیز کووالانسی را با استفاده از یک حفره اکسیانیون (Oxyanion hole) انجام دهند و تجمع بار را در حالت گذار تثبیت میکنند و با استفاده از یک بستر آب گرا، هیدرولیز کامل را ایجاد میکنند.[۴۸]
پویاییشناسی
آنزیمها ساختارهای ساکن و سختی نیستند. در عوض آنها دارای حرکتهای دینامیکی پیچیده داخلی شامل حرکات قسمتهایی از ساختار آنزیم مانند باقیمانده اسیدهای آمینه جداگانه، گروههای باقیمانده که یک حلقه پروتئین یا واحد ساختار ثانویه تشکیل میدهند، یا حتی یک کل حوزه پروتئین هستند. این حرکات منجر به ایجاد یک مجموعه ساختاری از ساختارهای کمی متفاوت میشود که در تعادل با یکدیگر تعامل برقرار میکنند. حالات مختلف درون این مجموعه ممکن است با جنبههای مختلف عملکرد آنزیم مرتبط باشد. به عنوان مثال، ترکیبات مختلف آنزیم دی هیدروفولات ردوکتاز مطابق با تئوری تشدید کاتالیزوری (Catalytic resonance theory) با اتصال بستر، تجزیه، آزاد سازی کوفاکتور و مراحل آزاد سازی محصول از چرخه کاتالیزوری مرتبط است.[۴۹]
ارائه بستر
ارائه بستر (Substrate presentation) فرایندی است که در آن آنزیم از بستر خود جدا میشود. آنزیمها را میتوان به غشا پلاسما به دور از یک بستر در هسته یا سیتوزول جدا کرد. یا درون غشا، یک آنزیم را میتوان به صورت تقسیم لیپید دور از بستر خود در ناحیه مختل شده جدا کرد. هنگامی که آنزیم آزاد میشود با بستر آن مخلوط میشود. متناوباً، میتوان آنزیم را در نزدیکی بستر آن جداسازی کرد تا آنزیم فعال شود. به عنوان مثال، آنزیم میتواند محلول باشد و هنگام فعال شدن به یک لیپید در غشای پلاسما متصل شود و سپس بر روی مولکولهای غشای پلاسما عمل کند.
مدولاسیون آلوستریک
مکانهای آلوستریک یا دگرریختار محلهایی روی آنزیم هستند که از جایگاه فعال متمایز هستند و به مولکولهای محیط سلولی متصل میشوند. این مولکولها سپس باعث تغییر در ساختار یا دینامیک آنزیمی میشوند که به جایگاه فعال منتقل میشود و بنابراین بر سرعت واکنش آنزیم تأثیر میگذارد.[۵۰] به این ترتیب، برهمکنش دگرریختار میتواند آنزیمها را مهار یا فعال کند. برهمکنش دگرریختار با متابولیتهای بالادست یا پایین دست در مسیر متابولیسم آنزیم باعث تنظیم بازخورد میشود و فعالیت آنزیم را با توجه به شار (Flux (metabolism)) از طریق بقیه مسیر تغییر میدهد.[۵۱]
کوفاکتورها

برخی از آنزیمها برای نشان دادن فعالیت کامل نیازی به مؤلفههای اضافی ندارند. برخی دیگر برای محدود کردن فعالیت به مولکولهای غیر پروتئینی به نام کوفاکتور نیاز دارند.[۵۲] کوفاکتورها میتوانند به صورت معدنی (مانند یونهای فلزی و خوشههای گوگرد آهن (Iron–sulfur cluster)) یا ترکیبات آلی (به عنوان مثال، فلاوین (Flavin group) و هم) باشند. این کوفاکتورها اهداف بسیاری را ارائه میدهند. به عنوان مثال، یونهای فلزی میتوانند در تثبیت گونههای هسته ای در محل فعال کمک کنند.[۵۳] کوفاکتورهای آلی میتوانند یا کوآنزیمهایی باشند که در طی واکنش از محل فعال آنزیم آزاد میشوند، یا گروههای پروتز که محکم به آنزیم وصل میشوند. گروههای پروتزهای آلی میتوانند به صورت کووالانسی محدود شوند (به عنوان مثال، بیوتین در آنزیمهایی مانند پیروات کربوکسیلاز).[۵۴]
کربنیک آنهیدراز نمونه ای از آنزیمیست که حاوی کوفاکتور است، که از یک کوفاکتور روی که به عنوان بخشی از سایت فعال آن وجود دارد استفاده میکند.[۵۵] این یونها یا مولکولهای معمولاً در محل فعال یافت میشوند و درگیر در کاتالیز هستند.[۲] : 8.1.1 به عنوان مثال، فلاوین و سازندههای هم اغلب در واکنشهای ردوکس نقش دارند. : 17
به آنزیمهایی که به کوفاکتور احتیاج دارند اما محدودیتی ندارند، آنزیم بنزیم یا آپوپروتئین گفته میشود. یک آنزیم همراه با کوفاکتور مورد نیاز برای فعالیت یک هولو آنزیم[e] یا هالوآنزیم[f] نامیده میشود. اصطلاح هولوزیم نیز میتواند برای آنزیمهایی از جمله DNA پلیمرازها که حاوی زیر واحدهای پروتئینی متعددی هستند استفاده شود. در اینجا هولوزانیم یک کمپلکس کامل است که شامل تمام زیر واحدهای مورد نیاز برای فعالیت است.[۲] : 8.1.1
کوآنزیمها
کوآنزیمها مولکولهای آلی کوچکی هستند که میتوانند آزادانه یا محکم به آنزیم متصل شوند. کوآنزیمها گروههای شیمیایی را از یک آنزیم به دیگری منتقل میکنند.[۵۶] نمونههای آن شامل نیکوتینآمید آدنین دینوکلئوتید فسفات (NADPH)، نیکوتینآمید آدنین دینوکلئوتید (NADH) و آدنوزین تریفسفات (ATP) است. برخی کوآنزیمها، مانند فلاوین مونونوکلئوتید (FMN)، فلاوین آدنین دینوکلئوتید (FAD)، تیامین پیروفسفات (TPP) و تترا هیدروفولات (THF) از ویتامینها مشتق میشوند. این کوآنزیمها نمیتوانند توسط بدن de novo سنتز (De novo synthesis) شوند و ترکیبات (ویتامینها) که از نزدیک مرتبط هستند باید از رژیم غذایی حاصل شود. گروههای شیمیایی حمل شده شامل موارد زیر هستند:
- یون هیدرید (H -)، که توسط NAD یا NADP + حمل شدهاست.
- گروه فسفات، توسط آدنوزین تریفسفات حمل شدهاست.
- گروه استیل، توسط کوآنزیم آ حمل شدهاست.
- گروههای فرمیل، متیل یا متیل، توسط اسید فولیک حمل شدهاست؛ و
- گروه متیل، توسط S-آدنوزیل متیونین حمل شدهاست.[۵۶]
از آنجا که کوآنزیمها به عنوان یک نتیجه از عمل آنزیم شیمیایی تغییر مییابند، در نظر گرفتن کوآنزیمها به عنوان یک کلاس خاص از سوبستراها، یا سوبستراهای دوم مفید است که برای بسیاری از آنزیمهای مختلف مشترک است. به عنوان مثال، حدود ۱۰۰۰ آنزیم شناخته شدهاست که از کوآنزیم NADH استفاده میکنند.[۵۷]
کوآنزیمها معمولاً بهطور مداوم بازسازی میشوند و غلظت آنها در سطح ثابت داخل سلول حفظ میشود. به عنوان مثال، NADPH از طریق مسیر فسفات پنتوز و S-آدنوزیل متیونین توسط متیونین آدنوزیل ترانسفراز ( Methionine adenosyltransferase) بازسازی میشود. این بازسازی مداوم یعنی مقادیر کمی از کوآنزیمها میتوانند بسیار فشرده استفاده شوند. به عنوان مثال، بدن انسان هر روز وزن خود را در ATP تغییر میدهد.[۵۸]
ترمودینامیک
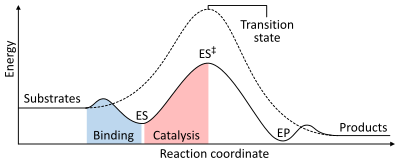
مانند همه کاتالیزورها، آنزیمها موقعیت تعادل شیمیایی واکنش را تغییر نمیدهند. در حضور آنزیم، واکنش در همان جهت که آنزیم نیز حضور ندارد انجام میشود فقط حضور آنزیم موجب میشود که با سرعت بیشتری انجام شود.[۲] : 8.2.3 به عنوان مثال، انیدراز کربنیک بسته به غلظت واکنش دهندههای آن، واکنش آن را از هر جهت تغییر میدهد:[۵۹]
-
(in tissues; high CO2 concentration)
()
-
(in lungs; low CO2 concentration)
()
میزان واکنش بستگی به انرژی فعالسازی مورد نیاز برای تشکیل حالت گذار دارد که پس از آن در محصولات فروپاشی میشود. آنزیمها با کاهش انرژی حالت گذار، سرعت واکنش را افزایش میدهند. اول، اتصال یک مجموعه پیچیده آنزیم-سوبسترا کم انرژی (ES) را تشکیل میدهد. دوم، آنزیم وضعیت انتقال را به گونهای تثبیت میکند که در مقایسه با واکنش غیرقابل تجزیه (ES ‡) به انرژی کمتری برای رسیدن به آن نیاز دارد. سرانجام، کمپلکس آنزیم-محصول (EP) برای انتشار محصولات جدا میشود.[۲] : 8.3
آنزیمها میتوانند دو یا چند واکنش ایجاد کنند، به طوری که میتوان از یک واکنش ترمودینامیکی مطلوب برای «رانش» ترمودینامیکی نامطلوب استفاده کرد به طوری که انرژی ترکیبی محصولات پایینتر از سوبستراها باشد. به عنوان مثال، هیدرولیز ATP اغلب برای هدایت سایر واکنشهای شیمیایی استفاده میشود.[۶۰]
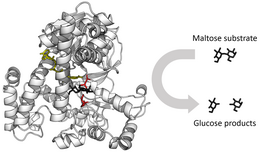
سینتیک
سینتیک آنزیم بررسی چگونگی اتصال آنزیمها به سوبستراها و تبدیل آنها به محصولات است.[۶۱] دادههای نرخ استفاده شده در آنالیزهای سینتیکی معمولاً از سنجش آنزیم به دست میآیند. در سال ۱۹۱۳ لیونور میکائلیس و ماد منتن نظریه کمی از سینتیک آنزیم را مطرح کردند که از آن به عنوان سینتیک میکائلیس–منتن یاد میشود.[۶۲] سهم عمده میکائلیس و منتن در فکر کردن به واکنشهای آنزیمی در دو مرحله بود. در حالت اول، سوبسترا برگشتپذیر به آنزیم متصل میشود، و کمپلکس آنزیم-سوبسترا را تشکیل میدهد که به افتخار آنها مجموعه کمپلکس میکائلیس و منتن نامیده میشود. سپس آنزیم مرحله شیمیایی واکنش را کاتالیز میکند و محصول را آزاد میکند. این کار توسط جورج ادوارد بریگز (George Edward Briggs) و جان هالدین توسعه یافت. آنها معادلات سینتیک را بدست آوردند که امروزه هنوز هم کاربرد گستردهای دارند.[۶۳]
میزان آنزیم به شرایط محلول و غلظت سوبسترا بستگی دارد. برای پیدا کردن حداکثر سرعت یک واکنش آنزیمی، غلظت سوبسترا افزایش مییابد تا اینکه یک مقدار ثابت در شکلگیری محصول مشاهده شود که در منحنی اشباع مقابل نشان داده شدهاست. با افزایش غلظت سوبسترا، بیشتر و بیشتر آنزیم آزاد به کمپلکس substrate-bound ES ES تبدیل میشود و اشباع رخ میدهد. در ماکزیمم سرعت واکنش (V Max) آنزیم، تمام سایتهای فعال آنزیم به سوبسترا متصل شده و مقدار کمپلکس ES همان مقدار کل آنزیم است.[۲] : 8.4
V max تنها یکی از چند پارامتر مهم سینتیکی است. مقدار بستر مورد نیاز برای دستیابی به میزان واکنش خاص نیز مهم است که توسط ثابت سینتیک میکائلیس–منتن (Km) که غلظت بستر مورد نیاز یک آنزیم برای رسیدن به نیمی از سرعت واکنش حداکثر (V max) آن است، داده شدهاست. بهطور کلی، هر آنزیم دارای یک KM ویژه برای یک بستر مشخص است. یکی دیگر از ثابتهای مفید، kcat است که به آن عدد گردش نیز گفته میشود و تعداد مولکولهای بستر است که توسط یک سایت فعال در هر ثانیه اداره میشود.[۲] : 8.4
سینتیک میکائلیس–منتن به قانون فعالیت جرمی (Law of mass action) متکی است، که از مفروضات واپخش آزاد و برخورد تصادفی از ترمودینامیک ناشی میشود. بسیاری از فرایندهای بیوشیمیایی یا سلولی به دلیل ازدحام ماکرومولکولی (Macromolecular crowding) و حرکت مولکولی محدود، از این شرایط بهطور قابل توجهی منحرف میشوند.[۶۴] اخیراً، با گسترش نمونه کمپلکس سعی در اصلاح این اثرات دارند.[۶۵]
بازداری
میزان سرعت واکنش آنزیم به وسیله انواع مختلفی از مهارکنندههای آنزیم قابل کاهش است. بازدارندههای آنزیمی عوامل مولکولی هستند که با کاتالیزوز تداخل پیدا کرده و واکنشهای آنزیمی را آهسته یا متوقف میکنند. بازدارندهها به صورت برگشتپذیر یا برگشتناپذیر هستند. بازدارندههای برگشتپذیر عبارتند از:[۶۷] : 73–74
برگشتپذیر
رقابتی
یک بازدارندهٔ رقابتی به خاطر تشابه در هندسه مولکولی یا سوبسترا برای جایگاه فعال آنزیم رقابت میکند با اشغال جایگاه فعال توسط بازدارنده از اتصال سوبسترا با آنزیم ممانعت میکند. مهارکننده (Competitive inhibition) و سوبسترا نمیتوانند همزمان با آنزیم متصل شوند.[۶۸] اغلب مهار کنندههای رقابتی کاملاً شبیه سوبسترا واقعی آنزیم هستند. به عنوان مثال، داروی متوترکسات یک مهار کننده رقابتی آنزیم دی هیدروفولات ردوکتاز است که باعث کاهش دی هیدروفولات به تتراهیدروفولات میشود.[۶۶] شباهت بین ساختارهای دی هیدروفولات و این دارو در شکل نشان داده شدهاست. با غلظت زیاد سوبسترا میتوان بر این نوع مهارکننده غلبه کرد. در برخی موارد، مهارکننده میتواند به سایتی غیر از محل اتصال سوبسترا معمولی وصل شود و یک اثر آلوستریک برای تغییر شکل محل اتصال معمولی اعمال کند.[۶۹]
غیر رقابتی
مهارکننده غیر رقابتی (Non-competitive inhibition) به مکانی غیر از محل اتصال سوبسترا متصل میشود. سوبسترا هنوز هم با تمایل معمول آن متصل میشود و از این رو Km یکسان باقی میماند. با این حال بازدارنده بازده کاتالیزوری آنزیم را کاهش میدهد به طوری که V max کاهش مییابد. بر خلاف مهار رقابتی، نمی توان با غلظت زیاد سوبسترا بر مهار غیر رقابتی سوبسترا غلبه کرد. مهار آنزیم انولاز در مسیر گلیکولیز توسط یون فلوئور نوعی از مهار غیر رقابتی است.[۶۷] : 76–78
بدون رقابت
مهارکننده بدون رقابت (Uncompetitive inhibitor) نمیتواند تنها به مجموعه آنزیم سوبسترا وصل شود، از این رو، این نوع مهارکنندهها در غلظت بالای سوبسترا مؤثر هستند. در حضور مهارکننده، مجموعه آنزیم-سوبسترا غیرفعال است.[۶۷] : 78 این نوع مهار نادر است. یک بازدارندهٔ نارقابتی در مکانی غیر از سوبسترا به آنزیم متصل شده و بر خلاف مهارکنندهٔ رقابتی تنها به کمپلکس ES متصل میشود.[۷۰]
مخلوط
یک مهار کننده مخلوط (Mixed inhibition) به یک سایت آلوستریک متصل میشود و اتصال سوبسترا و بازدارنده روی یکدیگر تأثیر میگذارند. عملکرد آنزیم هنگام اتصال به بازدارنده کاهش مییابد اما از بین نمیرود. این نوع بازدارندهها از معادله میکائلیس–منتن پیروی نمیکنند.[۶۷]: 76–78
برگشتناپذیر
یک بازدارنده برگشتناپذیر، آنزیم را معمولاً با ایجاد پیوند کووالانسی به پروتئین بطور دائمی غیرفعال میکند.[۷۱] پنیسیلین[۷۲] و آسپرین داروهای رایجی هستند که به این روش عمل میکنند.[۷۳]
عملکرد
در بسیاری از ارگانیسمها، بازدارندهها ممکن است به عنوان بخشی از یک مکانیسم بازخورد عمل کنند. اگر یک آنزیم بیش از حد یک ماده در ارگانیسم تولید کند، ممکن است آن ماده به عنوان یک بازدارنده برای آنزیم در ابتدای مسیر تولیدکننده آن عمل کند و باعث کند شدن تولید ماده در صورت وجود مقدار کافی شود که نوعی بازخورد منفی است. مسیرهای متابولیکی عمده مانند چرخه اسید سیتریک از این مکانیسم استفاده میکنند.[۲]: 17.2.2
از آنجا که بازدارندهها عملکرد آنزیمها را تعدیل میکنند، اغلب به عنوان دارو استفاده میشوند. بسیاری از این داروها بازدارندههای قابل برگشت رقابتی هستند که شبیه سوبسترا بومی آنزیم هستند، مشابه متوترکسات که در بالا اشاره شد، سایر نمونههای معروف شامل استاتینهایی است که برای درمان کلسترول بالا استفاده میشود،[۷۴] و بازدارندههای پروتئاز که برای درمان عفونتهای ویروسی مانند ویروس HIV استفاده میشود.[۷۵] یک نمونه معمول از یک بازدارنده برگشتناپذیر که به عنوان دارو استفاده میشود، آسپرین یا استیلسالیسیلیک اسید است که آنزیمهای COX-1 و COX-2 را که پروستاگلاندین پیام رسان التهاب را مهار میکنند.[۷۳] دیگر بازدارندههای آنزیم سموم هستند. به عنوان مثال، سیانور یک مهارکننده آنزیم برگشتناپذیر است که با مس و آهن در محل فعال آنزیم سیتوکروم اکسیداز سی ترکیب شده و از تنفس سلولی جلوگیری میکند.[۷۶]
تثبیت آنزیم
اکثر آنزیمها نسبتاً ناپایدار هستند و هزینههای تولید و جداسازی بالایی دارند و این یک نقطه ضعف را نشان میدهد که بازیابی آنزیمهای فعال در مخلوط واکنش پس از استفاده از نظر فنی بسیار دشوار است.[۷۷] آنزیمهای بی حرکت مورد توجه بسیاری قرار گرفته که مایلند از فناوری بیحرکتی آنزیمی برای اهداف خاص در بخشهای پزشکی و صنعتی استفاده کنند.[۷۸] اصطلاح «آنزیمهای بی حرکت» به آنزیمهایی گفته میشود که از نظر فیزیکی به تکیه گاههای جامد خاص متصل شده و بنابراین محدود شدهاند و میتوانند بطور مکرر و مداوم با حفظ فعالیتهای کاتالیزوری خود مورد استفاده قرار گیرند.[۷۹]
در سالهای اخیر، بهطور موازی با درک مکانیسمهای بیوسنتز آنزیمی، بهرهوری آنزیمی از طریق بهبود فناوری مهندسی ژنتیک، فناوری کشت میکروبی در حال رشد است. استفاده آنزیم بی حرکت در بیوتکنولوژی دارای مزایایی است.[۸۰] معرفی کاتالیزورهای آنزیمی بی حرکت، عملکرد فنی فرایندهای صنعتی را بسیار بهبود بخشیده و در نتیجه راندمان و بهرهوری اقتصادی را افزایش دادهاست. آنزیمهای بی حرکت معمولاً پایدارتر از آنزیمهای متحرک هستند، میتوان با حذف آنزیم از محلول واکنش، سرعت واکنش را کنترل کرد. یک مزیت اضافی جداسازی آسان آنزیم از محصول است تا بتوان از آلودگی جلوگیری کرد. همچنین، استفاده از آنزیم بی حرکت امکان ایجاد یک سیستم واکنش چند آنزیمی را فراهم میکند. طی دهههای گذشته، مطالعات بیوشیمیایی و بیوفیزیکی به منظور افزایش پایداری و فعالیت آنزیمها از طریق بیحرکتی آنزیمها، بهطور فعال انجام شدهاست.[۸۱]
- Enzyme-Based Electrodes
عوامل مؤثر بر فعالیت آنزیم
از آنجا که آنزیمها از پروتئین تشکیل شدهاند، عملکرد آنها نسبت به تغییر در بسیاری از عوامل شیمیایی فیزیکی مانند pH، دما، غلظت بستر و غیره حساس است.
جدول زیر pH مناسب را برای آنزیمهای مختلف نشان میدهد.[۸۲]
| آنزیم | pH بهینه | توضیحات pH |
|---|---|---|
| پپسین | ۱٫۵–۱٫۶ | بسیار اسیدی است |
| اینوراز | ۴٫۵ | اسیدی |
| لیپاز (معده) | ۴٫۰–۵٫۰ | اسیدی |
| لیپاز (روغن کرچک) | ۴٫۷ | اسیدی |
| لیپاز (لوزالمعده) | ۸٫۰ | قلیایی |
| آمیلاز (مالت) | ۴٫۶–۵٫۲ | اسیدی |
| آمیلاز (لوزالمعده) | ۶٫۷–۷٫۰ | اسیدی-خنثی |
| سلوبیاز | ۵٫۰ | اسیدی |
| مالتاز | ۶٫۱–۶٫۸ | اسیدی |
| سوکراز | ۶٫۲ | اسیدی |
| کاتالاز | ۷٫۰ | خنثی |
| اوره | ۷٫۰ | خنثی |
| کولین استراز | ۷٫۰ | خنثی |
| ریبونوکلئاز | ۷٫۰–۷٫۵ | خنثی |
| فوماراز | ۷٫۸ | قلیایی |
| تریپسین | ۷٫۸–۸٫۷ | قلیایی |
| آدنوزین تری فسفات | ۹٫۰ | قلیایی |
| آرژیناز | ۱۰٫۰ | بسیار قلیایی است |
کارکرد زیستی
آنزیمها کاربردهای گستردهای در اندامهای زنده دارند. آنها برای ترارسانی پیام و تنظیم فعالیتهای سلول ضروری اند؛ از جمله مهمترین آنزیمها در تنظیم سلول میتوان به کیناز و فسفاتاز اشاره کرد.[۸۳] آنزیمها در ایجاد حرکت در ماهیچهها هم موثرند آنها با کمک میوزین و آبکافت ایتیپیایز در ماهیچهها کشش ایجاد میکنند. علاوه بر این آنزیمها در انتقال مواد در پیرامون سلول و جزئی از اسکلت سلولی اهمیت دارند.[۸۴]
فرگشت
مشابه پروتئینهای دیگر، آنزیمها با گذشت زمان از طریق جهشها و واگرایی توالی تغییر میکنند. با توجه به نقش اصلی آنها در متابولیسم ، تکامل آنزیم نقش اساسی در سازگاری دارد؛ بنابراین پرسشی اساسی مطرح است که چگونه آنزیمها میتوانند فعالیت آنزیمی خود را در کنار هم تغییر دهند. بهطور کلی پذیرفته شدهاست که بسیاری از فعالیتهای جدید آنزیم از طریق تکثیر ژن و جهش نسخههای تکراری تکامل یافتهاند اگرچه تکامل نیز میتواند بدون تکثیر انجام شود. یک نمونه از آنزیمی که فعالیت خود را تغییر دادهاست اجداد متیونیل آمینو پپتیداز (Methionyl aminopeptidase) (MAP) و کراتین آمیدینوهیدرولاز (کراتیناز) (Creatinase) است که به وضوح همولوگ هستند اما واکنشهای بسیار متفاوت را کاتالیز میکنند (MAP موجب حذف متیونین آمینو ترمینال در پروتئینهای جدید میشود، در حالی که کراتین را به سمت سارکوزین و اوره هیدرولیز میکند). افزون بر این، MAP وابسته به یون فلزی است در حالی که کراتیناز نیست، از این رو این ویژگی نیز با گذشت زمان از بین رفتهاست.[۸۵] تغییرات کوچک فعالیت آنزیمی در بین آنزیمها بسیار متداول است. بهطور خاص، ویژگی اتصال سوبسترا به راحتی و به سرعت با تغییر اسیدهای آمینه تک در پاکتهای اتصال دهنده سوبسترا خود تغییر میکند که اغلب در کلاسهای اصلی آنزیم مانند کینازها مشاهده میشود.[۸۶]
امروزه تکامل مصنوعی (in vitro) برای اصلاح فعالیت آنزیم یا ویژگی کاربردهای صنعتی مورد استفاده قرار میگیرد.
کاربردهای صنعتی
در صورت نیاز به کاتالیزورهای بسیار خاص، آنزیمها در صنایع شیمیایی و سایر کاربردهای صنعتی مورد استفاده قرار میگیرند. بهطور کلی آنزیمها در تعداد واکنشی که برای کاتالیز کردن و همچنین عدم ثبات آنها در حلالهای آلی و در دماهای بالا وجود دارد، محدود هستند. به عنوان یک نتیجه، مهندسی پروتئین یک زمینه فعال از تحقیقات است و شامل تلاش برای ایجاد آنزیمهای جدید با خواص جدید، چه از طریق طراحی منطقی و چه از طریق تکامل هدایت شده یا آزمایشگاهی است.[۸۷] این تلاشها با موفقیت شروع شدهاست و اکنون چند آنزیم برای کاتالیز واکنشهایی که در طبیعت وجود ندارد، طراحی شدهاست.[۸۸]
| کاربرد | آنزیمهای مورد استفاده | استفاده میکند |
|---|---|---|
| صنعت سوختهای زیستی | سلولز | سلولز را درون قندهایی که تخمیر میشوند برای تولید اتانول سلولزی تخمیر کنید.[۸۹] |
| لیگنینازها | پیش درمانی زیست توده برای تولید سوختهای زیستی. | |
| مواد شوینده بیولوژیکی | پروتئینها، آمیلازها، لیپازها | لکههای پروتئین، نشاسته و چربی یا روغن را از لباسهای شسته شده و ظرفها جدا کنید.[۹۰] |
| مانوزیداز(Beta-mannosidase) | لکههای مواد غذایی را از صمغ گوار افزودنی مواد غذایی حذف کنید. | |
| صنعت آبجو | آمیلاز، گلوکاناز(Glucanase)، پروتئازها | پلی ساکاریدها و پروتئینها را در مالت تقسیم کنید.[۹۱] : 150–9 |
| بتاگلوکاناز(Betaglucanases) | ویژگیهای تصفیه مخمر آبجو (Wort) و آبجو را بهبود بخشید. : 545 | |
| آمیلاز و پولولاناز | آبجو کم کالری تهیه کرده و تخمیر را تنظیم کنید. : 575 | |
| دکربوکسیلاز استولکتات (ALDC)(Acetolactate decarboxylase) | با کاهش تشکیل دیاستیل راندمان تخمیر را افزایش دهید.[۹۲] | |
| کاربردهای آشپزی | پاپائین | ترد گوشت برای پختوپز.[۹۳] |
| صنایع لبنی | رنین rennin (Chymosin) | پروتئین هیدرولیز در ساخت پنیر.[۹۴] |
| لیپازها | پنیر کاممبرت و پنیرهای آبی مانند روکفوررا تولید کنید.[۹۵] | |
| فرآوری مواد غذایی | آمیلاز | از نشاسته، قندهایی مانند تهیه شربت ذرت با فروکتوز بالا تولید کنید.[۹۶] |
| پروتئینها | سطح پروتئین آرد را مانند ساخت بیسکویت پایین بیاورید. | |
| تریپسین | غذاهای کودک هیپوآلرژیک تولید کنید.[۹۷] | |
| سلولاز(Cellulase)، پکتیناز(Pectinase) | آب میوهها را روشن کنید.[۹۸] | |
| زیستشناسی مولکولی | هستههای هسته ای، DNA لیگاز و پلیمراز(Polymerase) | برای ایجاد DNA نوترکیب از هضم محدود کننده و واکنش زنجیره ای پلیمراز استفاده کنید.[۲] : 6.2 |
| صنعت کاغذ | زایلاناز(Xylanase)، همیسلولاز( hemicellulases) و لیگنین پراکسیداز(Lignin peroxidase) | لیگنینرا از خمیر کرافت بردارید.[۹۹] |
| مراقبت شخصی | پروتئینها | پروتئینها را روی لنزهای تماسی جدا کنید تا از عفونت جلوگیری شود.[۱۰۰] |
| صنعت نشاسته | آمیلاز | نشاسته را به گلوکز و شربتهای مختلف تبدیل کنید.[۱۰۱] |
جستارهای وابسته
- فهرست آنزیمها
- آنزیمهای صنعتی
- بانکهای اطلاعاتی آنزیم
- BRENDA
- EXPASy
- IntEnz
- دانش نامه ژن و ژنوم کیوتو
- MetaCyc
واژنامه
منابع
- ↑ «زیمایه، آنزیم» [شیمی] همارزِ «آنزیم» (enzyme)؛ منبع: گروه واژهگزینی. جواد میرشکاری، ویراستار. دفتر اول. فرهنگ واژههای مصوب فرهنگستان. تهران: انتشارات فرهنگستان زبان و ادب فارسی. شابک ۹۶۴-۷۵۳۱-۳۱-۱ (ذیل سرواژهٔ زیمایه)
- ↑ ۲٫۰۰ ۲٫۰۱ ۲٫۰۲ ۲٫۰۳ ۲٫۰۴ ۲٫۰۵ ۲٫۰۶ ۲٫۰۷ ۲٫۰۸ ۲٫۰۹ ۲٫۱۰ Biochemistry (5th ed.). San Francisco: W.H. Freeman. 2002. ISBN 0-7167-4955-6.
- ↑ Murphy JM, Farhan H, Eyers PA (2017). "Bio-Zombie: the rise of pseudoenzymes in biology". Biochem Soc Trans. 45 (2): 537–544. doi:10.1042/bst20160400. PMID 28408493.
- ↑ Murphy JM, et al. (2014). "A robust methodology to subclassify pseudokinases based on their nucleotide-binding properties". Biochemical Journal. 457 (2): 323–334. doi:10.1042/BJ20131174. PMC 5679212. PMID 24107129.
- ↑ Schomburg I, Chang A, Placzek S, Söhngen C, Rother M, Lang M, Munaretto C, Ulas S, Stelzer M, Grote A, Scheer M, Schomburg D (January 2013). "BRENDA in 2013: integrated reactions, kinetic data, enzyme function data, improved disease classification: new options and contents in BRENDA". Nucleic Acids Research. 41 (Database issue): D764–72. doi:10.1093/nar/gks1049. PMC 3531171. PMID 23203881.
- ↑ Radzicka A, Wolfenden R (January 1995). "A proficient enzyme". Science. 267 (5194): 90–931. Bibcode:1995Sci...267...90R. doi:10.1126/science.7809611. PMID 7809611.
- ↑ Callahan BP, Miller BG (December 2007). "OMP decarboxylase—An enigma persists". Bioorganic Chemistry. 35 (6): 465–9. doi:10.1016/j.bioorg.2007.07.004. PMID 17889251.
- ↑ de Réaumur RA (1752). "Observations sur la digestion des oiseaux". Histoire de l'Academie Royale des Sciences. 1752: 266, 461.
- ↑ Williams, Henry Smith (1904). A History of Science: in Five Volumes. Volume IV: Modern Development of the Chemical and Biological Sciences. Harper and Brothers.
- ↑ Payen A, Persoz JF (1833). "Mémoire sur la diastase, les principaux produits de ses réactions et leurs applications aux arts industriels" [Memoir on diastase, the principal products of its reactions, and their applications to the industrial arts]. Annales de chimie et de physique. 2nd (به فرانسوی). 53: 73–92.
- ↑ Manchester KL (December 1995). "Louis Pasteur (1822–1895)–chance and the prepared mind". Trends in Biotechnology. 13 (12): 511–5. doi:10.1016/S0167-7799(00)89014-9. PMID 8595136.
- ↑ Kühne coined the word "enzyme" in: Kühne W (1877). "Über das Verhalten verschiedener organisirter und sog. ungeformter Fermente" [On the behavior of various organized and so-called unformed ferments]. Verhandlungen des Naturhistorisch-medicinischen Vereins zu Heidelberg. new series (به آلمانی). 1 (3): 190–193. Relevant passage on page 190: "Um Missverständnissen vorzubeugen und lästige Umschreibungen zu vermeiden schlägt Vortragender vor, die ungeformten oder nicht organisirten Fermente, deren Wirkung ohne Anwesenheit von Organismen und ausserhalb derselben erfolgen kann, als Enzyme zu bezeichnen." (Translation: In order to obviate misunderstandings and avoid cumbersome periphrases, [the author, a university lecturer] suggests designating as "enzymes" the unformed or not organized ferments, whose action can occur without the presence of organisms and outside of the same.)
- ↑ Holmes, Frederic Lawrence (2003). "Enzymes". In Heilbron, John L. (ed.). The Oxford Companion to the History of Modern Science. Oxford: Oxford University Press. p. 270. ISBN 978-0-19-974376-6.
- ↑ "Eduard Buchner". Nobel Laureate Biography. Nobelprize.org. Retrieved 23 February 2015.
- ↑ "Eduard Buchner – Nobel Lecture: Cell-Free Fermentation". Nobelprize.org. 1907. Retrieved 23 February 2015.
- ↑ Willstätter R (1927). "Faraday lecture. Problems and methods in enzyme research". Journal of the Chemical Society (Resumed): 1359–1381. doi:10.1039/JR9270001359. quoted in Blow D (April 2000). "So do we understand how enzymes work?" (PDF). Structure. 8 (4): R77–R81. doi:10.1016/S0969-2126(00)00125-8. PMID 10801479. Archived from the original (PDF) on 4 March 2016. Retrieved 16 February 2012.
- ↑ "Nobel Prizes and Laureates: The Nobel Prize in Chemistry 1946". Nobelprize.org. Retrieved 23 February 2015.
- ↑ Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR (May 1965). "Structure of hen egg-white lysozyme. A three-dimensional Fourier synthesis at 2 Ångström resolution". Nature. 206 (4986): 757–61. Bibcode:1965Natur.206..757B. doi:10.1038/206757a0. PMID 5891407.
- ↑ Johnson LN, Petsko GA (1999). "David Phillips and the origin of structural enzymology". Trends Biochem. Sci. 24 (7): 287–9. doi:10.1016/S0968-0004(99)01423-1. PMID 10390620.
- ↑ Nomenclature Committee. "Classification and Nomenclature of Enzymes by the Reactions they Catalyse". International Union of Biochemistry and Molecular Biology (NC-IUBMB). School of Biological and Chemical Sciences, Queen Mary, University of London. Archived from the original on 17 March 2015. Retrieved 6 March 2015.
- ↑ Nomenclature Committee. "EC 2.7.1.1". International Union of Biochemistry and Molecular Biology (NC-IUBMB). School of Biological and Chemical Sciences, Queen Mary, University of London. Archived from the original on 1 December 2014. Retrieved 6 March 2015.
- ↑ Moss GP. "Recommendations of the Nomenclature Committee". International Union of Biochemistry and Molecular Biology on the Nomenclature and Classification of Enzymes by the Reactions they Catalyse. Retrieved 2006-03-14.
- ↑ Anfinsen CB (July 1973). "Principles that govern the folding of protein chains". Science. 181 (4096): 223–30. Bibcode:1973Sci...181..223A. doi:10.1126/science.181.4096.223. PMID 4124164.
- ↑ Dunaway-Mariano D (November 2008). "Enzyme function discovery". Structure. 16 (11): 1599–600. doi:10.1016/j.str.2008.10.001. PMID 19000810.
- ↑ Petsko, Gregory A.; Ringe, Dagmar (2003). "Chapter 1: From sequence to structure". Protein structure and function. London: New Science. p. 27. ISBN 978-1-4051-1922-1.
- ↑ Chen LH, Kenyon GL, Curtin F, Harayama S, Bembenek ME, Hajipour G, Whitman CP (September 1992). "4-Oxalocrotonate tautomerase, an enzyme composed of 62 amino acid residues per monomer". The Journal of Biological Chemistry. 267 (25): 17716–21. PMID 1339435.
- ↑ Smith S (December 1994). "The animal fatty acid synthase: one gene, one polypeptide, seven enzymes". FASEB Journal. 8 (15): 1248–59. doi:10.1096/fasebj.8.15.8001737. PMID 8001737.
- ↑ "The Catalytic Site Atlas". The European Bioinformatics Institute. Retrieved 4 April 2007.
- ↑ ۲۹٫۰ ۲۹٫۱ Suzuki H (2015). "Chapter 7: Active Site Structure". How Enzymes Work: From Structure to Function. Boca Raton, FL: CRC Press. pp. 117–140. ISBN 978-981-4463-92-8.
- ↑ Krauss G (2003). "The Regulations of Enzyme Activity". Biochemistry of Signal Transduction and Regulation (3rd ed.). Weinheim: Wiley-VCH. pp. 89–114. ISBN 978-3-527-60576-7.
- ↑ Srinivasan, Bharath (2020-09-27). "Words of advice: teaching enzyme kinetics". The FEBS Journal. doi:10.1111/febs.15537. ISSN 1742-464X.
- ↑ Pravda L, Berka K, Svobodová Vařeková R, Sehnal D, Banáš P, Laskowski RA, Koča J, Otyepka M (2014). "Anatomy of Enzyme Channels". BMC Bioinformatics. 15: 379. doi:10.1186/s12859-014-0379-x. PMC 4245731. PMID 25403510.
{{cite journal}}: Unknown parameter|displayauthors=ignored (|display-authors=suggested) (help) - ↑ Srinivasan, Bharath (2020-09-27). "Words of advice: teaching enzyme kinetics". The FEBS Journal. doi:10.1111/febs.15537. ISSN 1742-464X.
- ↑ Jaeger KE, Eggert T (August 2004). "Enantioselective biocatalysis optimized by directed evolution". Current Opinion in Biotechnology. 15 (4): 305–13. doi:10.1016/j.copbio.2004.06.007. PMID 15358000.
- ↑ Shevelev IV, Hübscher U (May 2002). "The 3' 5' exonucleases". Nature Reviews Molecular Cell Biology. 3 (5): 364–76. doi:10.1038/nrm804. PMID 11988770.
- ↑ Zenkin N, Yuzenkova Y, Severinov K (July 2006). "Transcript-assisted transcriptional proofreading". Science. 313 (5786): 518–20. Bibcode:2006Sci...313..518Z. doi:10.1126/science.1127422. PMID 16873663.
- ↑ Ibba M, Soll D (2000). "Aminoacyl-tRNA synthesis". Annual Review of Biochemistry. 69: 617–50. doi:10.1146/annurev.biochem.69.1.617. PMID 10966471.
- ↑ Rodnina MV, Wintermeyer W (2001). "Fidelity of aminoacyl-tRNA selection on the ribosome: kinetic and structural mechanisms". Annual Review of Biochemistry. 70: 415–35. doi:10.1146/annurev.biochem.70.1.415. PMID 11395413.
- ↑ Khersonsky O, Tawfik DS (2010). "Enzyme promiscuity: a mechanistic and evolutionary perspective". Annual Review of Biochemistry. 79: 471–505. doi:10.1146/annurev-biochem-030409-143718. PMID 20235827.
- ↑ O'Brien PJ, Herschlag D (April 1999). "Catalytic promiscuity and the evolution of new enzymatic activities". Chemistry & Biology. 6 (4): R91–R105. doi:10.1016/S1074-5521(99)80033-7. PMID 10099128.
- ↑ Fischer E (1894). "Einfluss der Configuration auf die Wirkung der Enzyme" [Influence of configuration on the action of enzymes]. Berichte der Deutschen Chemischen Gesellschaft zu Berlin (به آلمانی). 27 (3): 2985–93. doi:10.1002/cber.18940270364. From page 2992: "Um ein Bild zu gebrauchen, will ich sagen, dass Enzym und Glucosid wie Schloss und Schlüssel zu einander passen müssen, um eine chemische Wirkung auf einander ausüben zu können." (To use an image, I will say that an enzyme and a glucoside [i.e., glucose derivative] must fit like a lock and key, in order to be able to exert a chemical effect on each other.)
- ↑ Koshland DE (February 1958). "Application of a Theory of Enzyme Specificity to Protein Synthesis". Proceedings of the National Academy of Sciences of the United States of America. 44 (2): 98–104. Bibcode:1958PNAS...44...98K. doi:10.1073/pnas.44.2.98. PMC 335371. PMID 16590179.
- ↑ Vasella A, Davies GJ, Böhm M (October 2002). "Glycosidase mechanisms". Current Opinion in Chemical Biology. 6 (5): 619–29. doi:10.1016/S1367-5931(02)00380-0. PMID 12413546.
- ↑ Savir Y, Tlusty T (2007). Scalas E (ed.). "Conformational proofreading: the impact of conformational changes on the specificity of molecular recognition" (PDF). PLOS ONE. 2 (5): e468. Bibcode:2007PLoSO...2..468S. doi:10.1371/journal.pone.0000468. PMC 1868595. PMID 17520027. Archived from the original (PDF) on 14 May 2011. Retrieved 22 August 2010.
- ↑ Warshel A, Sharma PK, Kato M, Xiang Y, Liu H, Olsson MH (August 2006). "Electrostatic basis for enzyme catalysis". Chemical Reviews. 106 (8): 3210–35. doi:10.1021/cr0503106. PMID 16895325.
- ↑ Benkovic SJ, Hammes-Schiffer S (August 2003). "A perspective on enzyme catalysis". Science. 301 (5637): 1196–202. Bibcode:2003Sci...301.1196B. doi:10.1126/science.1085515. PMID 12947189.
- ↑ Villa J, Strajbl M, Glennon TM, Sham YY, Chu ZT, Warshel A (October 2000). "How important are entropic contributions to enzyme catalysis?". Proceedings of the National Academy of Sciences of the United States of America. 97 (22): 11899–904. Bibcode:2000PNAS...9711899V. doi:10.1073/pnas.97.22.11899. PMC 17266. PMID 11050223.
- ↑ Polgár, L. (2005-07-07). "The catalytic triad of serine peptidases". Cellular and Molecular Life Sciences (به انگلیسی). 62 (19–20): 2161–2172. doi:10.1007/s00018-005-5160-x. ISSN 1420-682X. PMID 16003488.
- ↑ Ramanathan A, Savol A, Burger V, Chennubhotla CS, Agarwal PK (2014). "Protein conformational populations and functionally relevant substates". Acc. Chem. Res. 47 (1): 149–56. doi:10.1021/ar400084s. OSTI 1565147. PMID 23988159.
- ↑ Tsai CJ, Del Sol A, Nussinov R (2009). "Protein allostery, signal transmission and dynamics: a classification scheme of allosteric mechanisms" (PDF). Mol Biosyst. 5 (3): 207–16. doi:10.1039/b819720b. PMC 2898650. PMID 19225609.
- ↑ Changeux JP, Edelstein SJ (June 2005). "Allosteric mechanisms of signal transduction". Science. 308 (5727): 1424–8. Bibcode:2005Sci...308.1424C. doi:10.1126/science.1108595. PMID 15933191.
- ↑ de Bolster, M.W.G. (1997). "Glossary of Terms Used in Bioinorganic Chemistry: Cofactor". International Union of Pure and Applied Chemistry. Archived from the original on 21 January 2017. Retrieved 30 October 2007.
- ↑ Voet, Donald; Voet, Judith; Pratt, Charlotte (2016). Fundamentals of Biochemistry. Hoboken, New Jersey: John Wiley & Sons, Inc. p. 336. ISBN 978-1-118-91840-1.
- ↑ Chapman-Smith A, Cronan JE (1999). "The enzymatic biotinylation of proteins: a post-translational modification of exceptional specificity". Trends Biochem. Sci. 24 (9): 359–63. doi:10.1016/s0968-0004(99)01438-3. PMID 10470036.
- ↑ Fisher Z, Hernandez Prada JA, Tu C, Duda D, Yoshioka C, An H, Govindasamy L, Silverman DN, McKenna R (February 2005). "Structural and kinetic characterization of active-site histidine as a proton shuttle in catalysis by human carbonic anhydrase II". Biochemistry. 44 (4): 1097–115. doi:10.1021/bi0480279. PMID 15667203.
- ↑ ۵۶٫۰ ۵۶٫۱ Wagner AL (1975). Vitamins and Coenzymes. Krieger Pub Co. ISBN 0-88275-258-8.
- ↑ "BRENDA The Comprehensive Enzyme Information System". Technische Universität Braunschweig. Archived from the original on 6 May 2015. Retrieved 23 February 2015.
- ↑ Törnroth-Horsefield S, Neutze R (December 2008). "Opening and closing the metabolite gate". Proceedings of the National Academy of Sciences of the United States of America. 105 (50): 19565–6. Bibcode:2008PNAS..10519565T. doi:10.1073/pnas.0810654106. PMC 2604989. PMID 19073922.
- ↑ "Chapter 9: The Pulmonary System and Exercise". Essentials of Exercise Physiology (3rd ed.). Baltimore, Maryland: Lippincott Williams & Wilkins. 2006. pp. 312–3. ISBN 978-0-7817-4991-6.
- ↑ Bioenergetics 3 (3rd ed.). San Diego: Academic. 2002. ISBN 0-12-518121-3.
- ↑ Hans, Bisswanger. Enzyme kinetics: principles and methods (Third, enlarged and improved ed.). Weinheim, Germany. ISBN 978-3-527-80646-1. OCLC 992976641.
- ↑ Michaelis L, Menten M (1913). "Die Kinetik der Invertinwirkung" [The Kinetics of Invertase Action]. Biochem. Z. (به آلمانی). 49: 333–369.; Michaelis L, Menten ML, Johnson KA, Goody RS (2011). "The original Michaelis constant: translation of the 1913 Michaelis–Menten paper". Biochemistry. 50 (39): 8264–9. doi:10.1021/bi201284u. PMC 3381512. PMID 21888353.
- ↑ Briggs GE, Haldane JB (1925). "A Note on the Kinetics of Enzyme Action". The Biochemical Journal. 19 (2): 338–9. doi:10.1042/bj0190338. PMC 1259181. PMID 16743508.
- ↑ Ellis RJ (October 2001). "Macromolecular crowding: obvious but underappreciated". Trends in Biochemical Sciences. 26 (10): 597–604. doi:10.1016/S0968-0004(01)01938-7. PMID 11590012.
- ↑ Kopelman R (September 1988). "Fractal reaction kinetics". Science. 241 (4873): 1620–26. Bibcode:1988Sci...241.1620K. doi:10.1126/science.241.4873.1620. PMID 17820893.
- ↑ ۶۶٫۰ ۶۶٫۱ Goodsell, David S. (1999-08-01). "The Molecular Perspective: Methotrexate". The Oncologist (به انگلیسی). 4 (4): 340–341. doi:10.1634/theoncologist.4-4-340. ISSN 1083-7159. PMID 10476546.
- ↑ ۶۷٫۰ ۶۷٫۱ ۶۷٫۲ ۶۷٫۳ Cornish-Bowden A (2004). Fundamentals of Enzyme Kinetics (3 ed.). London: Portland Press. ISBN 1-85578-158-1.
- ↑ Price NC (1979). "What is meant by 'competitive inhibition'?". Trends in Biochemical Sciences. 4 (11): N272–N273. doi:10.1016/0968-0004(79)90205-6.
- ↑ Wu P, Clausen MH, Nielsen TE (December 2015). "Allosteric small-molecule kinase inhibitors" (PDF). Pharmacology & Therapeutics. 156: 59–68. doi:10.1016/j.pharmthera.2015.10.002. PMID 26478442.
- ↑ Cornish-Bowden A (July 1986). "Why is uncompetitive inhibition so rare? A possible explanation, with implications for the design of drugs and pesticides". FEBS Letters. 203 (1): 3–6. doi:10.1016/0014-5793(86)81424-7. PMID 3720956.
- ↑ Strelow, John M. (2017-01-01). "A Perspective on the Kinetics of Covalent and Irreversible Inhibition". SLAS DISCOVERY: Advancing Life Sciences R&D (به انگلیسی). 22 (1): 3–20. doi:10.1177/1087057116671509. ISSN 2472-5552. PMID 27703080.
- ↑ Fisher JF, Meroueh SO, Mobashery S (February 2005). "Bacterial resistance to beta-lactam antibiotics: compelling opportunism, compelling opportunity". Chemical Reviews. 105 (2): 395–424. doi:10.1021/cr030102i. PMID 15700950.
- ↑ ۷۳٫۰ ۷۳٫۱ Johnson DS, Weerapana E, Cravatt BF (June 2010). "Strategies for discovering and derisking covalent, irreversible enzyme inhibitors". Future Medicinal Chemistry. 2 (6): 949–64. doi:10.4155/fmc.10.21. PMC 2904065. PMID 20640225.
- ↑ Endo A (1 November 1992). "The discovery and development of HMG-CoA reductase inhibitors". J. Lipid Res. 33 (11): 1569–82. PMID 1464741.
- ↑ Wlodawer A, Vondrasek J (1998). "Inhibitors of HIV-1 protease: a major success of structure-assisted drug design". Annual Review of Biophysics and Biomolecular Structure. 27: 249–84. doi:10.1146/annurev.biophys.27.1.249. PMID 9646869.
- ↑ Yoshikawa S, Caughey WS (May 1990). "Infrared evidence of cyanide binding to iron and copper sites in bovine heart cytochrome c oxidase. Implications regarding oxygen reduction". The Journal of Biological Chemistry. 265 (14): 7945–58. PMID 2159465.
- ↑ Gurung, Neelam; Ray, Sumanta; Bose, Sutapa; Rai, Vivek (2013-09-11). "A Broader View: Microbial Enzymes and Their Relevance in Industries, Medicine, and Beyond". BioMed Research International (به انگلیسی). 2013. doi:doi.org/10.1155/2013/329121. ISSN 2314-6133. Retrieved 2020-12-02.
{{cite journal}}: Check|doi=value (help) - ↑ DiCosimo, Robert; McAuliffe, Joseph; Poulose, Ayrookaran J.; Bohlmann, Gregory (2013-07-08). "Industrial use of immobilized enzymes". Chemical Society Reviews (به انگلیسی). 42 (15): 6437–6474. doi:10.1039/C3CS35506C. ISSN 1460-4744. Retrieved 2020-12-02.
- ↑ "Immobilized enzymes - learning from past successes and failures". Trends in Biotechnology (به انگلیسی). 11 (11): 471–478. 1993-11-01. doi:10.1016/0167-7799(93)90080-S. ISSN 0167-7799. Retrieved 2020-12-02.
- ↑ Rai, R. (2015). Advances in Food Biotechnology (به انگلیسی). Wiley. p. 145. Retrieved 2020-12-02.
- ↑ Zhang, Yifei; Ge, Jun; Liu, Zheng (2015-06-25). "Enhanced Activity of Immobilized or Chemically Modified Enzymes". ACS Catalysis (به انگلیسی). American Chemical Society (ACS). 5 (8). doi:10.1021/acscatal.5b00996. ISSN 2155-5435.
- ↑ Jain, J. L. (May 1999). Fundamentals of biochemistry. New Delhi: S. Chand and Co. ISBN 8121903432. OCLC 818809626.
- ↑ Hunter T (January 1995). "Protein kinases and phosphatases: the yin and yang of protein phosphorylation and signaling". Cell. 80 (2): 225–36. doi:10.1016/0092-8674(95)90405-0. PMID 7834742. S2CID 13999125.
- ↑ Berg JS, Powell BC, Cheney RE (April 2001). "A millennial myosin census". Molecular Biology of the Cell. 12 (4): 780–94. doi:10.1091/mbc.12.4.780. PMC 32266. PMID 11294886.
- ↑ Murzin, A. G. "Can homologous proteins evolve different enzymatic activities?". Trends in Biochemical Sciences. 18 (11): 403–405. doi:10.1016/0968-0004(93)90132-7. ISSN 0968-0004. PMID 8291080.
- ↑ Ochoa, David; Bradley, David; Beltrao, Pedro. "Evolution, dynamics and dysregulation of kinase signalling". Current Opinion in Structural Biology. 48: 133–140. doi:10.1016/j.sbi.2017.12.008. ISSN 1879-033X. PMID 29316484.
- ↑ Renugopalakrishnan V, Garduño-Juárez R, Narasimhan G, Verma CS, Wei X, Li P (November 2005). "Rational design of thermally stable proteins: relevance to bionanotechnology". Journal of Nanoscience and Nanotechnology. 5 (11): 1759–1767. doi:10.1166/jnn.2005.441. PMID 16433409.
- ↑ Jiang L, Althoff EA, Clemente FR, Doyle L, Röthlisberger D, Zanghellini A, Gallaher JL, Betker JL, Tanaka F, Barbas CF, Hilvert D, Houk KN, Stoddard BL, Baker D (March 2008). "De novo computational design of retro-aldol enzymes". Science. 319 (5868): 1387–91. Bibcode:2008Sci...319.1387J. doi:10.1126/science.1152692. PMC 3431203. PMID 18323453.
- ↑ Sun Y, Cheng J (May 2002). "Hydrolysis of lignocellulosic materials for ethanol production: a review". Bioresource Technology. 83 (1): 1–11. doi:10.1016/S0960-8524(01)00212-7. PMID 12058826.
- ↑ Kirk O, Borchert TV, Fuglsang CC (August 2002). "Industrial enzyme applications". Current Opinion in Biotechnology. 13 (4): 345–351. doi:10.1016/S0958-1669(02)00328-2. PMID 12323357.
- ↑ Briggs, Dennis E. (1998). Malts and Malting (1st ed.). London: Blackie Academic. ISBN 978-0-412-29800-4.
- ↑ Dulieu C, Moll M, Boudrant J, Poncelet D (2000). "Improved performances and control of beer fermentation using encapsulated alpha-acetolactate decarboxylase and modeling". Biotechnology Progress. 16 (6): 958–65. doi:10.1021/bp000128k. PMID 11101321.
- ↑ Tarté, Rodrigo (2008). Ingredients in Meat Products Properties, Functionality and Applications. New York: Springer. pp. 177. ISBN 978-0-387-71327-4.
- ↑ "Chymosin – GMO Database". GMO Compass. European Union. 10 July 2010. Archived from the original on 26 March 2015. Retrieved 1 March 2015.
- ↑ Molimard P, Spinnler HE (February 1996). "Review: Compounds Involved in the Flavor of Surface Mold-Ripened Cheeses: Origins and Properties". Journal of Dairy Science. 79 (2): 169–184. doi:10.3168/jds.S0022-0302(96)76348-8.
- ↑ Guzmán-Maldonado H, Paredes-López O (September 1995). "Amylolytic enzymes and products derived from starch: a review". Critical Reviews in Food Science and Nutrition. 35 (5): 373–403. doi:10.1080/10408399509527706. PMID 8573280.
- ↑ "Protease – GMO Database". GMO Compass. European Union. 10 July 2010. Archived from the original on 24 February 2015. Retrieved 28 February 2015.
- ↑ Alkorta I, Garbisu C, Llama MJ, Serra JL (January 1998). "Industrial applications of pectic enzymes: a review". Process Biochemistry. 33 (1): 21–28. doi:10.1016/S0032-9592(97)00046-0.
- ↑ Bajpai P (March 1999). "Application of enzymes in the pulp and paper industry". Biotechnology Progress. 15 (2): 147–157. doi:10.1021/bp990013k. PMID 10194388.
- ↑ Begley CG, Paragina S, Sporn A (March 1990). "An analysis of contact lens enzyme cleaners". Journal of the American Optometric Association. 61 (3): 190–4. PMID 2186082.
- ↑ Farris, Paul L. (2009). "Economic Growth and Organization of the U.S. Starch Industry". In BeMiller, James N.; Whistler, Roy L. (eds.). Starch Chemistry and Technology (3rd ed.). London: Academic. ISBN 978-0-08-092655-1.
خواندن بیشتر
|
عمومی
تاریخچه و نامگذاری
|
ساختار و مکانیسم آنزیم
سینتیک و بازداری
|

![{\displaystyle {\ce {CO2{}+H2O->[{\text{Carbonic anhydrase}}]H2CO3}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/cb4c8837b26e96fe552c17d863f93e0618cd998b)
![{\displaystyle {\ce {CO2{}+H2O<-[{\text{Carbonic anhydrase}}]H2CO3}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/618e95485aa1c3c44a29c557ac448ae5b544ff07)