پیشنویس:سوختوساز

بیوشیمی کلیدی[ویرایش]

بسیاری از ساختارهای تشکیلدهندۀ جانوران، گیاهان و میکروبها از چهار دستهٔ اساسی مولکولها ساخته شدهاند: آمینو اسیدها، کربوهیدراتها، نوکلئیک اسیدها و لیپیدها (اغلب چربی نامیده میشوند). این مولکولها که برای زندگی بسیار مهم هستند در واکنشهای سوختوسازی هنگام تولید یاختهها و بافتهای جدید ساخته میشوند یا به وسیله گوارش شکسته میشوند تا به عنوان منبع انرژی استفاده شوند. این مواد بیوشیمیایی میتوانند با پیوستن به هم پلیمرهایی مانند دیانای و پروتئینها را بسازند که از درشتمولکولهای ضروری زندگی هستند.[۱]
| مولکول | نام شکل مونومری | نام شکل پلیمری | نمونه پلیمر |
|---|---|---|---|
| آمینو اسید | آمینو اسید | پروتئینها (ساختهشده از پلیپپتید) | پروتئینهای فیبری و پروتئینهای کروی |
| کربوهیدرات | مونوساکارید | پلیساکارید | نشاسته، گلیکوژن و سلولز |
| نوکلئیک اسید | نوکلئوتید | پلینوکلئوتید | دیانای و آرانای |
آمینو اسیدها و پروتئینها[ویرایش]
پروتئینها را آمینو اسیدهایی میسازند که با پیوند پپتیدی در یک زنجیرۀ خطی به هم پیوستهاند. بسیاری از پروتئینها آنزیمهایی هستند که واکنشهای شیمیایی سوختوساز را کاتالیز میکنند. دیگر پروتئینها عملکردهای ساختاری یا مکانیکی دارند، مانند آنهایی که اسکلت یاختهای (سامانهای از داربستها که شکل یاخته را حفظ میکند) را شکل میدهند.[۲] پروتئینها همچنین در پیامرسانی یاختهای، پاسخهای ایمنی، چسبندگی یاختهای، انتقال فعال در غشاها و چرخهٔ یاختهای تاثیرگذار هستند. آمینو اسیدها با فراهم کردن منبع کربن برای ورود به چرخه سیتریک اسید (چرخه کربس)،[۳] کمک بسیاری به سوختوساز انرژی در یاخته میکنند، به ویژه زمانی که منبع اولیه تامین انرژی، مانند گلوکز، کمیاب باشد و یا زمانی که یاختهها تحت فشار سوختوسازی قرار میگیرند.[۴]
لیپیدها[ویرایش]
لیپیدها گوناگونترین گروه مواد بیوشیمیایی هستند. کاربردهای اصلی آنها بهعنوان بخشی از ساختار غشاهای زیستی داخلی یا خارجی، مانند غشای یاختهای، و آزادسازی انرژی شیمیایی اکسیژن است.[۵] لیپیدها پلیمرهای اسیدهای چرب هستند که یک زنجیره هیدروکربنی بلند و غیرقطبی و یک ناحیه قطبی کوچک دارای اکسیژن دارد. لیپیدها معمولاً به عنوان مولکولهای زیستی آبگریز یا دوگانهدوست تعریف میشوند که در حلالهای آلی مانند الکل، بنزن یا کلروفرم حل میگردند.[۶] چربیها گروه بزرگی از ترکیبهای دارای اسیدهای چرب و گلیسرول هستند. یک مولکول گلیسرول که توسط پیوندهای استری به سه اسید چرب متصل است، تری گلیسرید نامیده میشود.[۷] در برخی مواقع نیز ممکن است ساختار پایهای لیپیدها با ترکیبات دیگری، مانند اسفنگوزین در اسفنگومیلین و گروههای آبدوست مانند فسفات در فسفولیپیدها همراه شود. استروئیدها مانند استرول یک گروه بزرگ دیگر از لیپیدها هستند.
کربوهیدراتها[ویرایش]

کربوهیدراتها، آلدهیدها یا کتونهایی هستند که گروههای هیدروکسیل زیادی دارند و میتوانند به صورت راستزنجیر یا حلقه وجود داشته باشند. کربوهیدراتها فراوانترین مولکولهای زیستی هستند و نقشهای پرشماری مانند ذخیره و انتقال انرژی (نشاسته، گلیکوژن) و تشکیل اجزای ساختاری (سلولز در گیاهان، کیتین در جانوران) را بر عهده دارند. واحدهای پایهای کربوهیدرات مونوساکارید نام دارند که شامل گالاکتوز، فروکتوز و مهمتر از همه گلوکز میشوند. مونوساکاریدها را میتوان به روشهای تقریباً نامحدودی به یکدیگر پیوند داد تا پلیساکاریدها را تشکیل دهند.[۸]
نوکلئوتیدها[ویرایش]
نوکلئیک اسیدهایی مانند دیانای و آرانای، پلیمرهای نوکلئوتیدی هستند. هر نوکلئوتید از یک قند ریبوز یا دئوکسی ریبوز تشکیل شده که به یک باز نیتروژندار و یک فسفات متصل است. نوکلئیک اسیدها برای ذخیره و استفاده از اطلاعات ژنتیکی و تفسیر آنها از طریق فرآیندهای رونویسی و بیوسنتز پروتئین حیاتی هستند. این اطلاعات توسط فرایندهای بازسازی دیانای محافظت و از طریق همانندسازی دیانای تکثیر میشوند. بسیاری از ویروسها، مانند اچآیوی، دارای ژنوم آرانای هستند که از رونویسی معکوس برای ایجاد الگوی دیانای از ژنوم آرانای ویروسی خود استفاده میکند.[۹] آرانای موجود در ریبوزیمها نقشی مشابه پیرایشگرها و آنزیمها دارند و میتواند واکنشهای شیمیایی را کاتالیز کند. نوکلئوزیدهای منفرد با اتصال یک نوکلئوباز به یک قند ریبوز ساخته میشوند. این بازها ترکیبهای ناجورحلقهٔ دارای نیتروژن هستند که به عنوان پورین یا پیریمیدین طبقهبندی میشوند. نوکلئوتیدها همچنین به عنوان کوآنزیم در سوختوساز واکنشهای انتقال گروهی عمل میکنند.[۱۰]
کوآنزیمها[ویرایش]

سوختوساز گسترهٔ بزرگی از واکنشهای شیمیایی را دربر میگیرد اما بیشتر آنها تحت چند نوع واکنش اساسی هستند که شامل انتقال گروههای عاملی اتمها و پیوندهای آنها در داخل مولکولها میشود.[۱۱] این شیمی رایج به یاختهها اجازه میدهد تا از مجموعه کوچکی از واسطههای سوختوسازی برای جابهجایی گروههای شیمیایی بین واکنشهای گوناگون استفاده کنند.[۱۰] این واسطههای انتقال گروه، کوآنزیم نام دارند. هر دسته از واکنشهای انتقال گروه توسط کوآنزیم ویژهای انجام میشود که بستری است برای مجموعهای از آنزیمهایی که آن را تولید و مجموعهای از آنزیمهایی که آن را مصرف میکنند؛ بنابراین این کوآنزیمها بهطور مداوم ساخته، مصرف و سپس بازیافت میشوند.[۱۲]
یکی از کوآنزیمهای مرکزی آدنوزین تریفسفات است که واحد انرژی سراسری یاختهها است. این نوکلئوتید برای انتقال انرژی شیمیایی بین واکنشهای شیمیایی گوناگون بهکار میرود. تنها مقدار کمی ATP در یاختهها وجود دارد، اما از آنجایی که بهطور مداوم بازسازی میشود، بدن انسان میتواند تقریباً وزن خود را در روز از ATP استفاده کند.[۱۲] ATP به عنوان پلی بین کاتابولیسم و آنابولیسم عمل میکند. کاتابولیسم مولکولها را تجزیه میکند و آنابولیسم آنها را در کنار هم قرار میدهد. واکنشهای کاتابولیک ATP تولید میکنند و واکنشهای آنابولیک آن را مصرف میکنند. همچنین در واکنشهای فسفوریلاسیون، گروههای فسفات را جابهجا میکند.[۱۳]
ویتامین یک ترکیب آلی مورد نیاز در مقادیر کم است که در یاختهها ساخته نمیشود. در تغذیه انسان، بیشتر ویتامینها پس از اصلاح به عنوان کوآنزیم عمل میکنند. برای نمونه، تمام ویتامینهای محلول در آب فسفریله میشوند یا هنگامی که در یاختهها استفاده میشوند با نوکلئوتیدها جفت میشوند.[۱۴] نیکوتینآمید آدنین دینوکلئوتید (+NAD) که از ویتامین ب۳ (نیاسین) مشتق شدهاست، یک کوآنزیم مهم و پذیرندهٔ هیدروژن است. صدها گونه جداگانه از دهیدروژنازها الکترونها را از بسترهای خود جدا میکنند و +NAD را به NADH کاهش میدهند. سپس شکل کاهشیافته کوآنزیم، بستری برای ردوکتازهای درون یاخته است که نیاز به انتقال اتمهای هیدروژن به بسترهای خود دارند.[۱۵] نیکوتینآمید آدنین دینوکلئوتید به دو شکل مرتبط در یاخته وجود دارد، NADH و NADPH. شکل NAD+/NADH در واکنشهای کاتابولیک مهمتر است، در حالی که NADP+/NADPH در واکنشهای آنابولیک استفاده میشود.

مواد معدنی و کوفاکتورها[ویرایش]
عناصر معدنی نقش مهمی در سوختوساز دارند. برخی از آنها فراوان هستند (مانند سدیم و پتاسیم) در حالی که برخی دیگر در غلظتهای معین کار میکنند. حدود ۹۹ درصد وزن بدن انسان از عناصر کربن، نیتروژن، کلسیم، سدیم، کلر، پتاسیم، هیدروژن، فسفر، اکسیژن و گوگرد تشکیل شدهاست. ترکیبهای آلی (پروتئینها، لیپیدها و کربوهیدراتها) دارای مقدار زیادی کربن و نیتروژن هستند. اکسیژن و هیدروژن در بدن بیشتر بهشکل آب هستند.[۱۶]
عناصر معدنی فراوان نقش الکترولیت را ایفا میکنند. مهمترین یونهای معدنی سدیم، پتاسیم، کلسیم، منیزیم، کلرید، فسفات هستند و مهمترین یون آلی بیکربنات است. حفظ شیب یونی دقیق در سراسر غشای یاختهای، فشار اسمزی و پیاچ را حفظ میکند. یونها برای کارکرد درست عصب و ماهیچه بسیار مهم هستند، زیرا پتانسیل عمل در این بافتها با تبادل الکترولیتها بین مایع برونیاختهای و مایع درونیاختهای سیتوزول تولید میشود. الکترولیتها از طریق پروتئینهایی در غشای یاختهای به نام کانالهای یونی وارد و خارج یاختهها میشوند. برای نمونه، انقباض ماهیچه به جابهجایی کلسیم، سدیم و پتاسیم از طریق کانالهای یونی در غشای یاختهای و لولههای T بستگی دارد.[۱۷]
فلزهای واسطه در جانداران معمولاً به عنوان عناصر کممقدار وجود دارند که روی و آهن فراوانترین آنها هستند. کوفاکتورهای فلزی محکم به مکانهای ویژهای در پروتئینها متصل میشوند. اگرچه کوفاکتورهای آنزیمی را میتوان در طول کاتالیز اصلاح کرد، اما همیشه در پایان واکنش کاتالیزشده به حالت اولیه خود بازمیگردند. ریزمغذیهای فلزی توسط ناقلهای ویژه وارد جانداران میشوند و هنگامی که استفاده نمیشوند به پروتئینهای ذخیرهسازی مانند فریتین یا متالوتیونئین میپیوندند.[۱۸][۱۹]
کاتابولیسم[ویرایش]
کاتابولیسم مجموعهای از فرآیندهای سوختوسازی است که مولکولهای بزرگ را تجزیه میکند. از جمله این موارد میتوان به شکستن و اکسیدکردن مولکولهای غذایی اشاره کرد. هدف از واکنشهای کاتابولیک تأمین انرژی و اجزای مورد نیاز واکنشهای آنابولیکی است که مولکولها را میسازند. ماهیت دقیق واکنشهای کاتابولیک از جانداری به جاندار دیگر متفاوت است و جانداران را میتوان بر اساس منابع انرژی، هیدروژن و کربن (گروههای تغذیهای ابتدایی آنها) طبقهبندی کرد، همانطور که در جدول زیر نشان داده شدهاست. ارگانوتروفها از مولکولهای آلی به عنوان منبعی از اتمهای هیدروژن یا الکترونهای استفاده میکنند، در حالی که لیتوتروفها از بسترهای معدنی استفاده میکنند. فتوتروفها نور خورشید را به انرژی شیمیایی تبدیل میکنند،[۲۰] درحالیکه کموتروفها به واکنشهای اکسایش-کاهش وابستهاند که در آنها الکترونها از مولکولهای اکسنده مانند مولکولهای آلی، هیدروژن، هیدروژن سولفید یا یونهای آهن به مولکولهای کاهندهٔ غنی از انرژی مانند اکسیژن،[۵] نیترات یا سولفات انتقال مییابند. در جانوران، این واکنشها شامل مولکولهای آلی پیچیدهای هستند که به مولکولهای سادهتر مانند کربن دیاکسید و آب تجزیه میشوند. جانداران فتوسنتزی، مانند گیاهان و سیانوباکترها، از واکنشهای مشابه انتقال الکترون برای ذخیره انرژی جذبشده از نور خورشید استفاده میکنند.[۲۱]
| منبع انرژی | نور خورشید | نور- | -تروف | ||
| مولکولهایپیشساخته | کمو- | ||||
| دهنده هیدروژن یا الکترون | ترکیب آلی | ارگانو- | |||
| ترکیب معدنی | لیتو- | ||||
| منبع کربن | ترکیب آلی | هترو- | |||
| ترکیب معدنی | اتو- | ||||
رایجترین مجموعه واکنشهای کاتابولیک در جانوران را میتوان به سه مرحله اصلی تقسیم کرد. در مرحلهٔ یکم مولکولهای آلی بزرگ مانند پروتئینها، پلیساکاریدها یا لیپیدها در خارج از یاختهها به اجزای کوچکتر خود گوارش میشوند. سپس، این مولکولهای کوچکتر توسط یاختهها جذب و به مولکولهای کوچکتر، معمولاً استیل کوآنزیم آ (acetyl-CoA) تبدیل میشوند که مقداری انرژی آزاد میکند. در نهایت، گروه استیل این مولکول در چرخه سیتریک اسید و زنجیره انتقال الکترون به آب و کربن دیاکسید اکسایش مییابد و انرژی O2 را آزاد میکند،[۵] در حالی که کوآنزیم +NAD را به NADH کاهش میدهد.
گوارش[ویرایش]
درشتمولکولها را نمیتوان مستقیم توسط یاختهها پردازش کرد. درشتمولکولها باید قبل از استفاده در سوختوساز یاختهای به واحدهای کوچکتر تقسیم شوند. دستههای گوناگونی از آنزیمها برای گوارش این پلیمرها استفاده میشوند. این آنزیمهای گوارشی شامل پروتئازهایی هستند که پروتئینها را به آمینو اسیدها تجزیه میکنند و همچنین گلیکوزید هیدرولازهایی که پلیساکاریدها را به قندهای سادهای به نام مونوساکارید میشکنند.
میکروبها به سادگی آنزیمهای گوارشی را به محیط اطراف خود ترشح میکنند،[۲۲][۲۳] در حالی که جانوران این آنزیمها را فقط از یاختههای ویژهای در لولهٔ گوارش خود، از جمله معده و پانکراس و غدد بزاقی ترشح میکنند.[۲۴] آمینو اسید یا قندهای آزادشده توسط این آنزیمهای برونیاختهای توسط پروتئینهای انتقال فعال به یاختهها پمپ میشوند.[۲۵][۲۶]

انرژی دستامد از ترکیبهای آلی[ویرایش]
کاتابولیسم کربوهیدرات شامل تجزیه کربوهیدراتها به واحدهای کوچکتر است. کربوهیدراتها معمولاً پس از گوارش به مونوساکاریدها وارد یاختهها میشوند.[۲۷] پس از ورود، گلیکولیز مسیر اصلی تجزیه است، جایی که قندهایی مانند گلوکز و فروکتوز به پیروات تبدیلشده و مقداری ATP تولید میشود.[۲۸] پیرووات واسطهٔ چندین مسیر سوختوسازی است، اما بیشتر آن از طریق گلیکولیز هوازی (با اکسیژن) به استیل کوآ تبدیلشده و به چرخه سیتریک اسید وارد میشود. گرچه که بیشتر ATP در چرخه سیتریک اسید تولید میشود، با این وجود مهمترین محصول، NADH است که با اکسیدشدن استیل-CoA از +NAD تولید میشود. کربن دیاکسید در این اکسیداسیون به عنوان یک محصول زائد آزاد میشود. در شرایط بیهوازی، گلیکولیز لاکتات تولید میکند، از طریق آنزیم لاکتات دهیدروژناز که NADH را برای استفاده مجدد در گلیکولیز دوباره به +NAD اکسایش میکند.[۲۹] یک مسیر جایگزین برای تجزیه گلوکز، مسیر پنتوز فسفات است که کوآنزیم NADPH را کاهش میدهد و قندهای پنتوز مانند ریبوز را تولید میکند.
چربیها با هیدرولیز به اسیدهای چرب آزاد و گلیسرول کاتابولیز میشوند. گلیسرول وارد گلیکولیز میشود و اسیدهای چرب توسط اکسیداسیون بتا تجزیه میشوند تا استیل-کوآ آزاد شود که سپس به چرخه سیتریک اسید وارد میشود. اسیدهای چرب در هنگام اکسیداسیون انرژی بیشتری نسبت به کربوهیدراتها آزاد میکنند زیرا کربوهیدراتها دارای اکسیژن بیشتری در ساختار خود هستند.[۳۰] استروئیدها نیز توسط برخی باکتریها در فرآیندی شبیه به اکسیداسیون بتا تجزیه میشوند که شامل آزادشدن مقادیر قابل توجهی استیل کوآ، پروپیونیل کوآ و پیروات است که همگی میتوانند توسط یاخته برای انرژی استفاده شوند. همچنین مایکوباکتریوم توبرکلوزیس میتواند روی کلسترول به عنوان تنها منبع کربن رشد کند و ژنهای دخیل در مسیر (های) استفاده از کلسترول در مراحل مختلف چرخه زندگی عفونت مایکوباکتریوم توبرکلوزیس معتبر هستند.[۳۱]
آمینو اسیدها یا برای ساخت پروتئینها و دیگر مولکولهای زیستی استفاده میشوند یا برای تولید انرژی به اوره و کربن دیاکسید اکسایش مییابند.[۳۲] مسیر اکسیداسیون با حذف گروه آمین توسط یک ترانسآمیناز آغاز میشود. این گروه آمین وارد چرخه اوره میشود و یک اسکلت کربنی دآمینشده به شکل یک کتو اسید باقی میماند. تعدادی از این کتو اسیدها واسطههای چرخه سیتریک اسید هستند، برای نمونه آلفا-کتوگلوتارات که از دآمیناسیون گلوتامات تشکیل میشود.[۳۳] آمینو اسیدهای گلوکوژنیک نیز میتوانند از طریق گلوکونئوژنز به گلوکز تبدیل شوند (در زیر بحث شدهاست).[۳۴]
تحولات انرژی[ویرایش]
فسفرگیری اکسایشی[ویرایش]
در فسفرگیری اکسایشی، الکترونهای حذفشده از مولکولهای آلی در مناطقی مانند چرخه سیتریک اسید به اکسیژن منتقل میشود و انرژی آزاد شده برای ساخت ATP استفاده میشود. این کار در یوکاریوتها توسط یک سری پروتئین در غشای میتوکندری به نام زنجیرهٔ انتقال الکترون انجام میشود. در پروکاریوتها، این پروتئینها در غشای داخلی یاخته یافت میشوند.[۳۵] این پروتئینها از انرژی آزادشده توسط اکسیژن[۵] استفاده میکنند، زیرا الکترونها را از مولکولهای کاهشیافته مانند NADH دریافت میکنند تا پروتونها را در سراسر غشا پمپاژ کنند.[۳۶]

پمپاژ پروتونها از میتوکندری باعث ایجاد اختلاف غلظت پروتون در سراسر غشا میشود و یک گرادیان الکتروشیمیایی ایجاد میکند.[۳۷] این نیرو، پروتونها را از طریق یک پایهٔ آنزیمی به نام ایتیپی سنتاز به داخل میتوکندری برمیگرداند. جریان پروتونها باعث چرخش زیرواحد ساقه میشود و باعث میشود جایگاه فعال حوزه سنتاز تغییر شکل دهد و آدنوزین دیفسفات فسفریله شود و به ایتیپی تبدیل شود.[۱۲]
انرژی دستامد از ترکیبات معدنی[ویرایش]
کمولیتوتروفی نوعی سوختوساز است که در پروکاریوتها یافت میشود که در آن انرژی از اکسیداسیون ترکیبهای معدنی به دست میآید. این جانداران میتوانند از هیدروژن،[۳۸] ترکیبهای گوگرد کاهشیافته (مانند سولفید، هیدروژن سولفید و تیوسولفات)، اکسید آهن (II)[۳۹] یا آمونیاک[۴۰] به عنوان منابع قدرت کاهنده استفاده کنند و از اکسیداسیون این ترکیبها با گیرندههای الکترون پرانرژی مانند اکسیژن[۵] یا نیترات انرژی دریافت کنند.[۴۱] این فرآیندهای میکروبی در چرخه بیوژئوشیمی جهانی مانند استوژنز، نیتریفیکاسیون و دنیتریفیکاسیون و حاصلخیزی خاک مهم هستند.[۴۲][۴۳]
انرژی دستامد از نور[ویرایش]
انرژی نور خورشید توسط گیاهان، سیانوباکترها، باکتریهای بنفش، باکتریهای گوگردی سبز و برخی از آغازیان جذب میشود. این فرایند بیشتر با تبدیل کربن دیاکسید به ترکیبات آلی، به عنوان بخشی از فتوسنتز، که در زیر مورد بحث قرار میگیرد، همراه است. با این حال، سیستمهای جذب انرژی و تثبیت کربن میتوانند بهطور جداگانه در پروکاریوتها عمل کنند، زیرا باکتریهای بنفش و باکتریهای گوگردی سبز میتوانند از نور خورشید به عنوان منبع انرژی استفاده کنند، در حالی که بین تثبیت کربن و تخمیر ترکیبهای آلی جابهجا میشوند.[۴۴][۴۵]
در بسیاری از جانداران، جذب انرژی خورشید در اصل مشابه فسفرگیری اکسایشی است، زیرا شامل ذخیره انرژی به عنوان یک گرادیان غلظت پروتون است. سپس این نیروی محرکه پروتون باعث ساخت ایتیپی میشود الکترونهای مورد نیاز برای هدایت این زنجیره انتقال الکترون از پروتئینهای جمعآوری نور به نام مراکز واکنش فتوسنتزی میآیند. مراکز واکنش بسته به ماهیت رنگدانه فتوسنتزی موجود به دو نوع طبقهبندی میشوند، بیشتر باکتریهای فتوسنتزی فقط یک نوع مرکز واکنش دارند، در حالی که گیاهان و سیانوباکتریها هر دو نوع مرکز واکنش را دارند.[۴۶]
در گیاهان، جلبکها و سیانوباکتریها، فتوسیستم II از انرژی نور برای حذف الکترونها از آب استفاده میکند و اکسیژن را به عنوان یک محصول زائد آزاد میکند. سپس الکترونها به سمت کمپلکس سیتوکروم b6f جریان مییابند که از انرژی آنها برای پمپاژ پروتونها در سراسر غشای تیلاکوئید در کلروپلاست استفاده میکند.[۲۱] این پروتونها همانطور که قبلاً ایتیپی سنتاز را هدایت میکنند از طریق غشا به عقب بر میگردند. سپس الکترونها از طریق فتوسیستم I جریان مییابند و سپس میتوان از آنها برای کاهش کوآنزیم NADP+ استفاده میکند.[۴۷] این کوآنزیم میتواند وارد چرخه کالوین شود که در زیر به آن پرداخته شدهاست یا برای تولید بیشتر ATP بازیافت شود.
آنابولیسم[ویرایش]
آنابولیسم مجموعهای از فرآیندهای سوختوسازی سازنده است که در آن انرژی آزادشده توسط کاتابولیسم برای ساخت مولکولهای پیچیده استفاده میشود. بهطور کلی، مولکولهای پیچیدهای که ساختارهای یاختهای را تشکیل میدهند، گامبهگام از پیشسازهای کوچکتر و سادهتر ساخته میشوند. آنابولیسم شامل سه مرحله اساسی است. یکم تولید پیشسازهایی مانند آمینو اسیدها، مونوساکاریدها، ایزوپرنوئیدها و نوکلئوتیدها، دوم فعالشدن آنها به اشکال واکنشی با استفاده از انرژی ATP و سوم، تبدیل این پیشسازها به مولکولهای پیچیده مانند پروتئینها، پلیساکاریدها، لیپیدها و نوکلئیک اسیدها.[۴۸]
آنابولیسم در جانداران میتواند با توجه به منبع مولکولهای ساختهشده در یاختههای آنها متفاوت باشد. اتوتروفهایی مانند گیاهان میتوانند مولکولهای آلی پیچیده در یاختههای خود مانند پلیساکاریدها و پروتئینها را از مولکولهای ساده مانند کربن دیاکسید و آب بسازند. از طرف دیگر هتروتروفها برای تولید این مولکولهای پیچیده به منبعی از مواد پیچیدهتری مانند مونوساکاریدها و آمینو اسیدها نیاز دارند. جانداران را میتوان برپایهٔ منبع نهایی انرژی خود طبقهبندی کرد: فتواتوتروفها و فتوهتروتروفها انرژی را از نور دریافت میکنند، در حالی که کمواتوتروفها و کموهتروتروفها انرژی را از واکنشهای اکسیداسیون دریافت میکنند.[۴۸]
تثبیت کربن[ویرایش]

فتوسنتز ساخت کربوهیدرات از نور خورشید و کربن دیاکسید است. در گیاهان، سیانوباکترها و جلبکها، فتوسنتز اکسیژنی، آب را میشکافد و اکسیژن بهعنوان یک محصول زائد تولید میشود. این فرایند از ATP و NADPH تولید شده توسط مراکز واکنش فتوسنتز، برای تبدیل CO2 به گلیسرات ۳-فسفات استفاده میکند که پس از آن میتواند به گلوکز تبدیل شود. این واکنش تثبیت کربن توسط آنزیم RuBisCO به عنوان بخشی از چرخه کالوین–بنسون انجام میشود.[۴۹] سه نوع فتوسنتز در گیاهان رخ میدهد، تثبیت کربن C3، تثبیت کربن C4 و فتوسنتز CAM. اینها برپایهٔ مسیری که کربن دیاکسید به چرخه کالوین طی میکند با هم متفاوت هستند، به گونهای که گیاهان C3 کربن دیاکسید را مستقیم تثبیت میکنند، در حالی که گیاهان C4 و CAM کربن دیاکسید را ابتدا در ترکیبهای دیگر تثبیت میکند. این تثبیت کربن دو مرحلهای یک نوع سازگاری برای مقابله با نور شدید خورشید و شرایط خشک است.[۵۰]
در پروکاریوتهای فتوسنتزکننده تثبیت کربن متنوعتر است. در اینجا، کربن دیاکسید را میتوان توسط چرخه کالوین–بنسون، چرخه سیتریک اسید معکوس،[۵۱] یا کربوکسیلاسیون استیل-کوآ تثبیت کرد.[۵۲][۵۳] کمواتوتروفهای پروکاریوتی نیز CO2 را از طریق چرخه کالوین-بنسون تثبیت میکنند، اما از انرژی حاصل از ترکیبهای معدنی برای هدایت واکنش استفاده میکنند.[۵۴]
کربوهیدراتها و گلیکانها[ویرایش]
در آنابولیسم کربوهیدرات، اسیدهای آلی ساده را میتوان به مونوساکاریدهایی مانند گلوکز تبدیل کرد و سپس برای ساختپلیساکاریدهایی مانند نشاسته استفاده کرد. تولید گلوکز از ترکیبهایی مانند پیروات، لاکتات، گلیسرول، گلیسرات ۳-فسفات و آمینو اسید را گلوکونئوژنز میگویند. گلوکونئوژنز، پیرووات را از طریق مجموعهای از مواد واسطه که بسیاری از آنها با گلیکولیز مشترک هستند، به گلوکز-۶-فسفات تبدیل میکند.[۲۸] با این حال، این مسیر به سادگی گلیکولیز به صورت معکوس انجام نمیشود، زیرا چندین مرحله توسط آنزیمهای غیر گلیکولیتیک کاتالیز میشوند. این مهم است زیرا اجازه میدهد تا تشکیل و تجزیه گلوکز بهطور جداگانه تنظیم شود و از حرکت همزمان هر دو مسیر در یک چرخه بیهوده جلوگیری میکند.[۵۵][۵۶]
اگرچه چربی یک روش متداول برای ذخیره انرژی است، اما در مهرهدارانی مانند انسان، اسیدهای چرب موجود در این ذخایر نمیتوانند از طریق گلوکونئوژنز به گلوکز تبدیل شوند، زیرا این جانداران نمیتوانند استیل-کوآ را به پیروات تبدیل کنند، اما گیاهان ماشینآلات آنزیمی لازم برای این کار را دارند.[۵۷] در نتیجه، پس از گرسنگی درازمدت، مهرهداران نیاز به تولید اجسام کتونی از اسیدهای چرب دارند تا جایگزین گلوکز در بافتهایی مانند مغز شوند که نمیتوانند اسیدهای چرب را متابولیزه کنند.[۵۸] در جانداران دیگر مانند گیاهان و باکتریها، این مشکل سوختوسازی با استفاده از چرخهٔ گلیاگزالات حل میشود، که مرحله دکربوکسیلاسیون را در چرخه سیتریک اسید دور میزند و امکان تبدیل استیل-کوآ به اگزالواستات را فراهم میکند، جایی که میتوان از آن برای تولید گلوکز استفاده کرد.[۵۷][۵۹] به غیر از چربی، گلیکوژنز، گلوکز را در بیشتر بافتها بهعنوان یک منبع انرژی موجود در بافت ذخیره میکند که معمولاً برای حفظ سطح گلوکز در خون استفاده میشود.[۶۰]
پلیساکاریدها و گلیکانها با افزودن پیدرپی مونوساکاریدها توسط گلیکوزیلترانسفراز از یک اهداکننده قند-فسفات فعال مانند اوریدین دیفسفات گلوکز به یک گروه پذیرندهٔ هیدروکسیلی روی پلیساکارید در حال رشد ساخته میشوند. از آنجایی که هر یک از گروههای هیدروکسیل روی حلقه بستر میتوانند پذیرنده باشند، پلیساکاریدهای تولیدشده میتوانند ساختار راست یا شاخهای داشته باشند. پلیساکاریدهای تولیدشده میتوانند خود عملکردهای ساختاری یا سوختوسازی داشته باشند یا توسط آنزیمهایی به نام الیگوساکاریلترانسفراز به لیپیدها و پروتئینها منتقل شوند.[۶۱][۶۲]
اسیدهای چرب، ایزوپرنوئیدها و استرول[ویرایش]
اسیدهای چرب توسط سنتازهای اسید چرب ساخته میشوند که واحدهای استیل کوآ را پلیمریزه و سپس کاهش میدهند. زنجیرههای آسیل در اسیدهای چرب با چرخهای از واکنشها گسترش مییابند که گروه آسیل را اضافه میکند، آن را به الکل کاهش میدهد، با گرفتن آب، آن را به یک گروه آلکن تبدیل میکند و سپس دوباره آن را به یک گروه آلکان کاهش میدهد. آنزیمهای بیوسنتز اسیدهای چرب به دو گروه تقسیم میشوند: در جانوارن و قارچها، تمام این واکنشهای سنتاز اسیدهای چرب توسط یک پروتئین چند عملکردی نوع I انجام میشود،[۶۳] در حالی که در پلاستیدهای گیاهی و باکتریها آنزیمهای نوع II جداگانه هر مرحله را در مسیر انجام میدهند.[۶۴][۶۵]
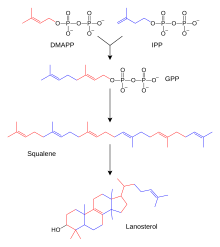
ترپنها و ایزوپرنوئیدها دسته بزرگی از لیپیدها هستند که شامل کاروتنوئیدها هستند و بزرگترین دسته از فراوردههای طبیعی گیاهی را تشکیل میدهند.[۶۶] این ترکیبها با مونتاژ و اصلاح واحدهای ایزوپرن اهدایی از پیشسازهای واکنشی ایزوپنتنیل پیروفسفات و دیمتیلآلیل پیروفسفات ساخته میشوند.[۶۷] این پیشسازها میتوانند به روشهای گوناگونی ساخته شوند. در جانوران و آرکیها، مسیر موالونات این ترکیبها را از استیل کوآ تولید میکند؛[۶۸] در حالی که در گیاهان و باکتریها، مسیر غیر موالونات از پیروات و گلیسرآلدئید-۳-فسفات به عنوان بستر استفاده میکند.[۶۷][۶۹] یکی از واکنشهای مهمی که از این اهداکنندگان ایزوپرن فعال استفاده میکند، بیوسنتز استرول است. در اینجا، واحدهای ایزوپرن به هم متصل میشوند تا اسکوالن بسازند و سپس تا میشوند و به صورت مجموعهای از حلقهها در میآیند تا لانوسترول ساختهشود.[۷۰] سپس لانوسترول میتواند به دیگر استرولها مانند کلسترول و ارگوسترول تبدیل شود.[۷۰][۷۱]
پروتئینها[ویرایش]
توانایی جانداران در ساخت ۲۰ آمینو اسید رایج متفاوت است. بیشتر باکتریها و گیاهان میتوانند هر بیست آمینو اسید را بسازند، اما پستانداران فقط میتوانند ۱۱ آمینو اسید غیر ضروری را بسازند، بنابراین ۹ آمینو اسید ضروری را باید از غذا دریافت کنند. برخی از انگلهای ساده، مانند باکتری مایکوپلاسما پنومونیه، نمیتوانند هیچیک از آمینو اسیدها را بسازند و آمینو اسیدهای خود را مستقیماً از میزبان خود میگیرند.[۷۲] تمام آمینو اسیدها از واسطههای گلیکولیز، چرخه سیتریک اسید یا مسیر پنتوز فسفات ساخته میشوند. نیتروژن توسط گلوتامات و گلوتامین تأمین میشود. سنتز آمینو اسید غیر ضروری به تشکیل آلفا-کتو اسید مناسب بستگی دارد، که سپس برای تشکیل یک آمینو اسید ترانسآمینه میشود.
آمینو اسیدها با اتصال در زنجیرهای از پیوندهای پپتیدی به پروتئین تبدیل میشوند. هر پروتئین متفاوت دنبالهای منحصر به فرد از باقیمانده آمینو اسیدها دارد: این دنباله، ساختار اولیه پروتئین است. همانگونه که حروف الفبا را میتوان با یکدیگر ترکیب کرد تا تنوع تقریباً بیپایانی از واژگان ایجاد شود، آمینو اسیدها نیز میتوانند در دنبالههای گوناگونی به هم بپیوندند و تنوع بزرگی از پروتئینها را تشکیل دهند. پروتئینها از آمینو اسیدهایی ساخته میشوند که با اتصال به آرانای حامل از طریق پیوند استری فعال شدهاند. این پیش ساز آمینواسیل-تیآرانای در یک واکنش وابسته به ATP که توسط یک آمینواسیل تیآرانای سنتتاز انجام میشود تولید میشود.[۷۳] سپس این آمینواسیل-تیآرانای بستری برای ریبوزوم است، که با استفاده از اطلاعات دنبالهٔ آرانای پیامرسان، آمینو اسید را به زنجیره پروتئینی طویلشده پیوند میدهد.[۷۴]
سنتز و نجات نوکلئوتید[ویرایش]
نوکلئوتیدها از آمینو اسیدها، کربن دیاکسید و فرمیک اسید در مسیرهایی ساخته میشوند که به مقادیر زیادی انرژی سوختوسازی نیاز دارند.[۷۵] در نتیجه، بیشتر جانداران دارای سیستمهای کارآمدی برای بازیافت نوکلئوتیدهای از پیشساخته هستند.[۷۵][۷۶] پورینها بهصورت نوکلئوزیدها (بازهای متصل به ریبوز) ساخته میشوند.[۷۷] آدنین و گوانین هر دو از پیشساز نوکلئوزید اینوزین مونوفسفات ساخته میشوند که با استفاده از اتمهای آمینو اسید گلیسین، گلوتامین و آسپارتیک اسید و همچنین فرمات انتقالیافته از کوآنزیم تتراهیدروفولات ساخته میشود. پیریمیدینها، از باز اوروتات که از گلوتامین و آسپارتات تشکیل میشود، ساخته میشوند.[۷۸]
ژنوبیوتیک و سوختوساز اکسایش و کاهش[ویرایش]
همه جانداران پیوسته در معرض ترکیباتی هستند که نمیتوانند از آنها به عنوان غذا استفاده کنند و اگر در یاختهها تجمع کنند، مضر خواهند بود، زیرا عملکرد سوختوسازی ندارند. این ترکیبات بالقوه آسیبرسان بیگانهزیست نام دارند.[۷۹] بیگانهزیستها مانند داروهای مصنوعی، سموم طبیعی و آنتیبیوتیکها توسط مجموعهای از آنزیمهای متابولیزهکننده بیگانهزیست، سمزدایی میشوند. در انسان، اینها شامل سیتوکروم پی ۴۵۰ اکسیدازها،[۸۰] یودیپی-گلوکورونوزیلترانسفراز،[۸۱] و گلوتاتیون اس-ترانسفرازها است.[۸۲] این سیستم از آنزیمها در سه مرحله عمل میکند تا ابتدا بیگانهزیست را اکسید کرده (فاز I) و سپس گروههای محلول در آب را با مولکول درهم میآمیزد (فاز II). بیگانهزیست اصلاحشده محلول در آب میتواند از یاختهها خارج شود و در جانداران چندیاختهای ممکن است پیش از دفع بیشتر متابولیزه شود (فاز III). در اکولوژی، این واکنشها بهویژه در تخریب زیستی میکروبی آلایندهها و پاکسازی زیستی زمینهای آلوده و نشت نفت اهمیت دارند.[۸۳] بسیاری از این واکنشهای میکروبی با جانداران چندیاختهای مشترک است، اما به دلیل تنوع باورنکردنی انواع میکروبها، این جانداران میتوانند با گستره بسیار بزرگی از بیگانهزیستها نسبت به جانداران چندیاختهای مقابله کنند و میتوانند حتی آلایندههای آلی پایدار مانند ترکیبهای آلی کلر را تجزیه کنند.[۸۴]
یک مشکل مرتبط برای جانداران هوازی استرس اکسیداتیو است.[۸۵] در اینجا، فرآیندهایی از جمله فسفرگیری اکسایشی و تشکیل پیوندهای دیسولفیدی در طی تاخوردگی پروتئین، گونههای فعال اکسیژن مانند هیدروژن پراکسید را میسازند.[۸۶] این اکسیدانهای مضر توسط متابولیتهای آنتیاکسیدانی مانند گلوتاتیون و آنزیمهایی مانند کاتالازها و پراکسیدازها حذف میشوند.[۸۷][۸۸]
ترمودینامیک جانداران زنده[ویرایش]
جانداران زنده باید از قوانین ترمودینامیک پیروی کنند که انتقال گرما و کار را توصیف میکند. قانون دوم ترمودینامیک بیان میکند که در هر سیستم ایزوله، مقدار آنتروپی (آشفتگی) نمیتواند کاهش یابد. اگرچه به نظر میرسد پیچیدگی شگفتانگیز جانداران زنده با این قانون در تناقض است، اما زندگی ممکن است زیرا همه جانداران سامانههای باز هستند که ماده و انرژی را با محیط پیرامون خود مبادله میکنند. سیستمهای زنده در تعادل نیستند، اما در عوض سیستمهای اتلافی هستند که با ایجاد افزایش بیشتر در آنتروپی محیطشان، وضعیت پیچیدگی بالایی خود را حفظ میکنند.[۸۹] سوختوساز یک یاخته با جفت کردن فرآیندهای خودبهخودی کاتابولیسم با فرآیندهای غیر خودبهخودی آنابولیسم به این امر دست مییابد. در اصطلاح ترمودینامیکی، سوختوساز با ایجاد بینظمی نظم را حفظ میکند.[۹۰]
تنظیم و کنترل[ویرایش]
با توجه به اینکه محیط بیشتر جانداران پیوسته در دگرگونی است، واکنشهای سوختوسازی باید به خوبی تنظیم شود تا مجموعهای از شرایط ثابت در یاختهها حفظ شود، وضعیتی که همایستایی نام دارد.[۹۱][۹۲] تنظیم سوختوسازی همچنین به جانداران اجازه میدهد تا به سیگنالها پاسخ دهند و بهطور فعال با محیط خود تعامل داشته باشند.[۹۳] دو مفهوم نزدیک به هم برای درک چگونگی کنترل مسیرهای سوختوسازی مهم هستند. یکم، تنظیم یک آنزیم در یک مسیر به این است که چگونه فعالیت آن در پاسخ به سیگنالها افزایش و کاهش مییابد. دوم، کنترل اعمال شده توسط این آنزیم تأثیری است که این تغییرات در فعالیت آن بر سرعت کلی مسیر (شار از طریق مسیر) میگذارد.[۹۴] برای نمونه، یک آنزیم ممکن است تغییرات زیادی در فعالیت نشان دهد (یعنی به شدت تنظیم شدهاست) اما اگر این تغییرات تأثیر کمی بر شار یک مسیر سوختوسازی داشته باشد، این آنزیم در کنترل مسیر نقش ندارد.[۹۵]

سطوح مختلفی از تنظیم سوختوسازی وجود دارد. در تنظیم ذاتی، مسیر سوختوسازی خود تنظیم میشود تا به تغییرات در سطوح بسترها یا فراوردهها پاسخ دهد. برای نمونه، کاهش در مقدار محصول میتواند شار را از طریق مسیر برای جبران افزایش دهد.[۹۴] این نوع تنظیم بیشتر شامل تنظیم آلوستریک فعالیت آنزیمهای پرشمار در مسیر است.[۹۶] کنترل بیرونی شامل یک یاخته در یک جاندار چندیاختهای است که سوختوساز خود را در پاسخ به سیگنالهای یاختههای دیگر تغییر میدهد. این سیگنالها معمولاً به شکل پیامرسانهای محلول در آب مانند هورمونها و فاکتورهای رشد هستند و توسط گیرندههای خاصی در سطح یاخته شناسایی میشوند.[۹۷] سپس این سیگنالها توسط سیستمهای پیامرسان دوم که اغلب درگیر فسفریلاسیون پروتئینها هستند، در داخل یاخته منتقل میشوند.[۹۸]
یک مثال کاملاً درکشده از کنترل بیرونی، تنظیم سوختوساز گلوکز توسط هورمون انسولین است.[۹۹] انسولین در پاسخ به افزایش سطح گلوکز خون تولید میشود. اتصال این هورمون به گیرندههای انسولین روی یاختهها، آبشاری از پروتئین کینازها را فعال میکند که باعث میشود یاختهها گلوکز را جذب کرده و آن را به مولکولهای ذخیرهسازی مانند اسیدهای چرب و گلیکوژن تبدیل کنند.[۱۰۰] سوختوساز گلیکوژن توسط فعالیت فسفریلاز، آنزیمی که گلیکوژن را تجزیه میکند و گلیکوژن سنتاز، آنزیمی که آن را میسازد، کنترل میشود. این آنزیمها به روشی متقابل تنظیم میشوند، با فسفوریلاسیون، گلیکوژن سنتاز را مهار میکند، اما فسفوریلاز را فعال میکند. انسولین با فعال کردن پروتئین فسفاتازها و کاهش فسفوریلاسیون این آنزیمها باعث ساخت گلیکوژن میشود.[۱۰۱]
تکامل[ویرایش]

مسیرهای مرکزی سوختوساز که در بالا توضیح دادهشد، مانند گلیکولیز و چرخه سیتریک اسید، در هر سه حوزه جانداران زنده وجود دارند و در آخرین نیای مشترک جهانی وجود داشتند.[۱۰۲][۱۰۳] این یاخته اجدادی جهانی پروکاریوتی و احتمالاً متانوژنی بود که دارای سوختوساز آمینو اسید، نوکلئوتید، کربوهیدرات و لیپید گسترده بود.[۱۰۴] حفظ این مسیرهای باستانی در طول تکامل بعدی ممکن است نتیجه این باشد که این واکنشها راه حلی بهینه برای مشکلات سوختوسازی ویژهٔ آنها بودهاست، با مسیرهایی مانند گلیکولیز و چرخه سیتریک اسید که فراوردههای نهایی خود را بسیار کارآمد و در حداقل تعداد مراحل تولید میکند.[۱۰۵][۱۰۶] اولین مسیرهای سوختوساز مبتنی بر آنزیم ممکن است بخشی از سوختوساز نوکلئوتید پورین باشد، در حالی که مسیرهای سوختوسازی قبلی بخشی از دنیای آرانای باستانی بودهاست.[۱۰۷]
مدلهای بسیاری برای توصیف سازوکارهایی پیشنهاد شدهاند که به کمک آن مسیرهای سوختوسازی جدید تکامل مییابند. از جمله این موارد میتوان به اضافهشدن پیدرپی آنزیمهای جدید به یک مسیر کوتاه اجدادی، تکثیر و سپس واگرایی همهٔ مسیرها و همچنین بهکارگیری آنزیمهای از پیش موجود و مونتاژ آنها در یک مسیر واکنش جدید اشاره کرد.[۱۰۸] اهمیت نسبی این سازوکارها نامشخص است، اما پژوهشهای ژنومی نشان دادهاند که آنزیمهای موجود در یک مسیر به احتمال زیاد نسب مشترکی دارند و پیشنهاد میکنند که بسیاری از مسیرها در یک روش گامبهگام با کارکردهای نو از مراحل پیشین در مسیر تکامل یافتهاند.[۱۰۹] یک مدل جایگزین از مطالعاتی که تکامل ساختار پروتئینها را در شبکههای سوختوسازی ردیابی میکند، نشان میدهد که آنزیمها بهطور فراگیر و برای انجام عملکردهای مشابه در مسیرهای سوختوساز گوناگون (که در پایگاه داده MANET مشهود است) بهکار گرفته میشوند.[۱۱۰] این فرآیندهای بهکارگیری منجر به یک موزاییک آنزیمی تکاملی میشود.[۱۱۱] احتمال سوم این است که برخی از بخشهای سوختوساز ممکن است «ماژولهایی» باشند که میتوانند در مسیرهای گوناگون مورد استفاده دولاره قرار گیرند و عملکردهای مشابهی را روی مولکولهای گوناگون انجام دهند.[۱۱۲]
تکامل افزون بر ساخت مسیرهای سوختوسازی جدید، میتواند باعث از دست دادن عملکردهای سوختوسازی نیز شود. برای نمونه، در برخی از انگلها فرآیندهای سوختوسازی که برای بقا ضروری نیستند از میان میروند و آمینو اسیدهای از پیشساختهشده، نوکلئوتیدها و کربوهیدراتها ممکن است از میزبان گرفته شوند.[۱۱۳] کاهش تواناییهای سوختوسازی مشابهی در جانداران درونهمزیستی دیده میشود.[۱۱۴]
تحقیق و دستکاری[ویرایش]

بهطور کلاسیک، سوختوساز با یک رویکرد تقلیلگرایانه که بر یک مسیر سوختوسازی واحد متمرکز است، مطالعه میشود. استفاده از ردیابهای رادیواکتیو در سطح کل جاندار، بافت و یاخته بسیار ارزشمند است که با شناسایی واسطهها و فراوردهها دارای برچسب رادیواکتیو، مسیرهای پیشسازها تا فراوردههای نهایی را مشخص میکند.[۱۱۵] آنزیمهایی که این واکنشهای شیمیایی را کاتالیز میکنند، میتوانند خالص شوند و سینتیک و پاسخ آنها به بازدارندهها بررسی شود. یک رویکرد موازی، شناسایی مولکولهای کوچک در یک یاخته یا بافت است. مجموعه کامل این مولکولها متابولوم نامیده میشود. بهطور کلی، این مطالعات دید خوبی از ساختار و عملکرد مسیرهای سوختوسازی ساده ارائه میدهند، اما زمانی که برای سیستمهای پیچیدهتر مانند سوختوساز یک یاخته کامل اعمال شوند، کافی نیستند.[۱۱۶]
تصوری از پیچیدگی شبکههای سوختوسازی در یاختههایی که دارای هزاران آنزیم گوناگون هستند با شکلی که برهمکنشهای بین ۴۳ پروتئین و ۴۰ متابولیت را در سمت راست نشان میدهد ارائه میشود: توالیهای ژنوم فهرستهایی دارای هر چیزی تا ۲۶٬۵۰۰ ژن را ارائه میدهند.[۱۱۷] با این حال، اکنون میتوان از این دادههای ژنومی برای بازسازی شبکههای کامل واکنشهای بیوشیمیایی و تولید مدلهای ریاضی جامعتر استفاده کرد که ممکن است رفتار آنها را توضیح و پیشبینی کند.[۱۱۸] این مدلها به ویژه زمانی قدرتمند هستند که برای ادغام مسیر و دادههای متابولیت بهدستآمده از روشهای کلاسیک با دادههای مربوط به بیان ژن از مطالعات پروتئومی و ریزآرایههای دیانای استفاده شوند.[۱۱۹] با استفاده از این تکنیکها، اکنون مدلی از سوختوساز انسان در دست داریم که کشف دارو و پژوهشهای بیوشیمیایی آینده را هدایت خواهد کرد.[۱۲۰] این مدلها اکنون در تجزیه و تحلیل شبکه استفاده میشوند تا بیماریهای انسانی را به گروههایی طبقهبندی کنند که پروتئینها یا متابولیتهای مشترک دارند.[۱۲۱][۱۲۲]
شبکههای سوختوسازی باکتریایی نمونهای بارز از سازماندهی پاپیون[۱۲۳][۱۲۴][۱۲۵] هستند، معماری که قادر است گسترهٔ بزرگی از مواد مغذی را وارد و گسترهٔ بزرگی از فراوردهها و درشتمولکولهای پیچیده را با استفاده از تعداد نسبتاً معدودی از ارزهای رایج متوسط تولید کند.
کاربرد فناورانه عمدهٔ این اطلاعات، مهندسی سوختوساز است. در اینجا، جاندارانی مانند مخمر، گیاهان یا باکتریها اصلاح ژنتیکی میشوند تا آنها را در زیستفناوری مفیدتر کنند و به تولید داروهایی مانند آنتیبیوتیکها یا مواد شیمیایی صنعتی مانند ۳٬۱- پروپاندیول و اسید شیکیمیک کمک کنند.[۱۲۶][۱۲۷][۱۲۸] این تغییرات ژنتیکی معمولاً با هدف کاهش میزان انرژی مصرفی برای تولید محصول، افزایش بازده و کاهش تولید پسماند انجام میشود.[۱۲۹]
پیشینه[ویرایش]
اصطلاح متابولیسم از فرانسوی «métabolisme» یا یونانی باستان «metabolή - Metabole» برای «تغییر» گرفته شدهاست که از «μεταβάλλ - Metaballein» به معنای «تغییر کردن» آمدهاست.[۱۳۰]

فلسفه یونانی[ویرایش]
اعضای جانوران ارسطو جزئیاتی کافی از دیدگاههای او دربارهٔ سوختوساز را برای ایجاد یک مدل جریان باز بیان میکند. او میپنداشت که در هر مرحله از فرایند، مواد غذایی تبدیل میشوند و گرما به عنوان عنصر کلاسیک آتش آزاد میشود و مواد باقیمانده بهصورت ادرار، صفرا یا مدفوع دفع میشوند.
طب اسلامی[ویرایش]
ابن النفیس سوختوساز را در اثر خود در سال ۱۲۶۰ میلادی با عنوان الرساله الکامیلیه فی سیره النبویه شرح دادهاست که شامل عبارت زیر است: «بدن و اعضای آن در یک حالت پیوسته از انحلال و تغذیه هستند، بنابراین آنها ناگزیر دستخوش تغییر دائمی میشوند.»
کاربرد روش علمی[ویرایش]
پیشینه مطالعه علمی سوختوساز چندین قرن را در بر میگیرد و از بررسی کامل جانوران در مطالعات اولیه به بررسی واکنشهای سوختوسازی فردی در بیوشیمی نوین حرکت کردهاست. نخستین آزمایش کنترلشده دربارهٔ سوختوساز بدن انسان را سانتوریو سانتوریو در ۱۶۱۴ در کتاب خود Ars de statica medicina منتشر کرد.[۱۳۱] او توضیح داد که چگونه قبل و بعد از خوردن غذا، خواب، کار، رابطه جنسی، روزه، نوشیدن و دفع خود را وزن کردهاست. او متوجه شد که بیشتر غذایی که میخورد از طریق چیزی که او آن را «تعریق نامحسوس» مینامید از بین میرفت.

در این پژوهشهای اولیه، مکانیسمهای این فرآیندهای سوختوسازی شناسایی نشده بودند و تصور میشد که نیرویی حیاتی برای زندهکردن بافت زنده وجود دارد. در قرن نوزدهم، هنگام مطالعه تخمیر شکر به الکل توسط مخمر، لویی پاستور به این نتیجه رسید که تخمیر توسط مواد درون یاختههای مخمر کاتالیز میشود که او آن را تخمیر مینامید. او نوشت که «تخمیر الکلی عملی است که با زندگی و سازماندهی یاختههای مخمر مرتبط است، نه با مرگ یا پوسیدگی یاختهها.»[۱۳۲] این کشف، همراه با انتشار مقالهای توسط فردریش ولر در سال ۱۸۲۸ در مورد ساخت شیمیایی اوره،[۱۳۳] و به دلیل اینکه اولین ترکیب آلی تهیهشده از پیشسازهای کاملاً معدنی است، قابل توجه است. این ثابت کرد که ترکیبهای آلی و واکنشهای شیمیایی موجود در یاختهها در اصل تفاوتی با دیگر بخشهای شیمی ندارند.
کشف آنزیمها در آغاز قرن بیستم توسط ادوارد بوشنر بود که مطالعه واکنشهای شیمیایی سوختوساز را از مطالعه زیستشناسی یاختهها جدا کرد و آغاز بیوشیمی را رقم زد.[۱۳۴] انبوه دانش بیوشیمیایی در اوایل قرن بیستم به سرعت رشد کرد. یکی از پرکارترین این بیوشیمیدانان نوین، هانس کربس بود که کمک زیادی به مطالعه سوختوساز کرد.[۱۳۵] او چرخه اوره را کشف کرد و بعدها با همکاری هانس کورنبرگ، چرخه سیتریک اسید و چرخه گلیاگزالات را کشف کرد.[۱۳۶][۱۳۷][۵۹] توسعه تکنیکهای جدید مانند کروماتوگرافی، پراش اشعه ایکس، طیفسنجی NMR، نشانگذاری رادیوایزوتوپی، میکروسکوپ الکترونی و شبیهسازی دینامیک مولکولی به پژوهشهای بیوشیمیایی نوین کمک زیادی کردهاست. این تکنیکها امکان کشف و تجزیه و تحلیل دقیق بسیاری از مولکولها و مسیرهای سوختوسازی در یاختهها را فراهم کردهاند.
جستارهای وابسته[ویرایش]
- آنتی متابولیتها
- کالریسنجی – اندازهگیری حالت ترمودینامیکی
- خطاهای مادرزادی متابولیسم – دستهای از بیماریهای ژنتیکی
- اختلال متابولیک
- گروههای تغذیهای ابتدایی
- دانش نامه ژن و ژنوم کیوتو – مجموعه پایگاههای بیوانفورماتیک
واژهنامه[ویرایش]
پانویس[ویرایش]
منابع[ویرایش]
- ↑ Cooper, Geoffrey M. (2000). "The Molecular Composition of Cells". The Cell: A Molecular Approach. 2nd Edition (به انگلیسی).
- ↑ Michie KA, Löwe J (2006). "Dynamic filaments of the bacterial cytoskeleton". Annual Review of Biochemistry. 75: 467–92. doi:10.1146/annurev.biochem.75.103004.142452. PMID 16756499.
- ↑ Kelleher JK, Bryan BM, Mallet RT, Holleran AL, Murphy AN, Fiskum G (September 1987). "Analysis of tricarboxylic acid-cycle metabolism of hepatoma cells by comparison of 14CO2 ratios". The Biochemical Journal. 246 (3): 633–9. doi:10.1042/bj2460633. PMC 1148327. PMID 3120698.
- ↑ Hothersall JS, Ahmed A (2013). "Metabolic fate of the increased yeast amino Acid uptake subsequent to catabolite derepression". Journal of Amino Acids. 2013: 461901. doi:10.1155/2013/461901. PMC 3575661. PMID 23431419.
- ↑ ۵٫۰ ۵٫۱ ۵٫۲ ۵٫۳ ۵٫۴ Schmidt-Rohr, K. (2020). "Oxygen Is the High-Energy Molecule Powering Complex Multicellular Life: Fundamental Corrections to Traditional Bioenergetics". ACS Omega 5: 2221-2233. http://dx.doi.org/10.1021/acsomega.9b03352.
- ↑ Fahy E, Subramaniam S, Brown HA, Glass CK, Merrill AH, Murphy RC, et al. (May 2005). "A comprehensive classification system for lipids". Journal of Lipid Research. 46 (5): 839–61. doi:10.1194/jlr.E400004-JLR200. PMID 15722563.
- ↑ "Lipid nomenclature Lip-1 & Lip-2". www.qmul.ac.uk. Retrieved 2020-06-06.
- ↑ Raman R, Raguram S, Venkataraman G, Paulson JC, Sasisekharan R (November 2005). "Glycomics: an integrated systems approach to structure-function relationships of glycans". Nature Methods. 2 (11): 817–24. doi:10.1038/nmeth807. PMID 16278650.
- ↑ Sierra S, Kupfer B, Kaiser R (December 2005). "Basics of the virology of HIV-1 and its replication". Journal of Clinical Virology. 34 (4): 233–44. doi:10.1016/j.jcv.2005.09.004. PMID 16198625.
- ↑ ۱۰٫۰ ۱۰٫۱ Wimmer MJ, Rose IA (1978). "Mechanisms of enzyme-catalyzed group transfer reactions". Annual Review of Biochemistry. 47: 1031–78. doi:10.1146/annurev.bi.47.070178.005123. PMID 354490.
- ↑ Mitchell P (March 1979). "The Ninth Sir Hans Krebs Lecture. Compartmentation and communication in living systems. Ligand conduction: a general catalytic principle in chemical, osmotic and chemiosmotic reaction systems". European Journal of Biochemistry. 95 (1): 1–20. doi:10.1111/j.1432-1033.1979.tb12934.x. PMID 378655.
- ↑ ۱۲٫۰ ۱۲٫۱ ۱۲٫۲ Dimroth P, von Ballmoos C, Meier T (March 2006). "Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series". EMBO Reports. 7 (3): 276–82. doi:10.1038/sj.embor.7400646. PMC 1456893. PMID 16607397.
- ↑ Bonora M, Patergnani S, Rimessi A, De Marchi E, Suski JM, Bononi A, et al. (September 2012). "ATP synthesis and storage". Purinergic Signalling. 8 (3): 343–57. doi:10.1007/s11302-012-9305-8. PMC 3360099. PMID 22528680.
- ↑ Berg, Jeremy M.; Tymoczko, John L.; Stryer, Lubert (2002). "Vitamins Are Often Precursors to Coenzymes". Biochemistry. 5th Edition (به انگلیسی).
- ↑ Pollak N, Dölle C, Ziegler M (March 2007). "The power to reduce: pyridine nucleotides--small molecules with a multitude of functions". The Biochemical Journal. 402 (2): 205–18. doi:10.1042/BJ20061638. PMC 1798440. PMID 17295611.
- ↑ Heymsfield SB, Waki M, Kehayias J, Lichtman S, Dilmanian FA, Kamen Y, et al. (August 1991). "Chemical and elemental analysis of humans in vivo using improved body composition models". The American Journal of Physiology. 261 (2 Pt 1): E190-8. doi:10.1152/ajpendo.1991.261.2.E190. PMID 1872381.
- ↑ Dulhunty AF (September 2006). "Excitation-contraction coupling from the 1950s into the new millennium". Clinical and Experimental Pharmacology & Physiology. 33 (9): 763–72. doi:10.1111/j.1440-1681.2006.04441.x. PMID 16922804.
- ↑ Cousins RJ, Liuzzi JP, Lichten LA (August 2006). "Mammalian zinc transport, trafficking, and signals". The Journal of Biological Chemistry. 281 (34): 24085–9. doi:10.1074/jbc.R600011200. PMID 16793761.
- ↑ Dunn LL, Suryo Rahmanto Y, Richardson DR (February 2007). "Iron uptake and metabolism in the new millennium". Trends in Cell Biology. 17 (2): 93–100. doi:10.1016/j.tcb.2006.12.003. PMID 17194590.
- ↑ Raven, Ja (2009-09-03). "Contributions of anoxygenic and oxygenic phototrophy and chemolithotrophy to carbon and oxygen fluxes in aquatic environments". Aquatic Microbial Ecology (به انگلیسی). 56: 177–192. doi:10.3354/ame01315. ISSN 0948-3055.
- ↑ ۲۱٫۰ ۲۱٫۱ Nelson N, Ben-Shem A (December 2004). "The complex architecture of oxygenic photosynthesis". Nature Reviews. Molecular Cell Biology. 5 (12): 971–82. doi:10.1038/nrm1525. PMID 15573135.
- ↑ Häse CC, Finkelstein RA (December 1993). "Bacterial extracellular zinc-containing metalloproteases". Microbiological Reviews. 57 (4): 823–37. doi:10.1128/MMBR.57.4.823-837.1993. PMC 372940. PMID 8302217.
- ↑ Gupta R, Gupta N, Rathi P (June 2004). "Bacterial lipases: an overview of production, purification and biochemical properties". Applied Microbiology and Biotechnology. 64 (6): 763–81. doi:10.1007/s00253-004-1568-8. PMID 14966663.
- ↑ Hoyle T (1997). "The digestive system: linking theory and practice". British Journal of Nursing. 6 (22): 1285–91. doi:10.12968/bjon.1997.6.22.1285. PMID 9470654.
- ↑ Souba WW, Pacitti AJ (1992). "How amino acids get into cells: mechanisms, models, menus, and mediators". Journal of Parenteral and Enteral Nutrition. 16 (6): 569–78. doi:10.1177/0148607192016006569. PMID 1494216.
- ↑ Barrett MP, Walmsley AR, Gould GW (August 1999). "Structure and function of facilitative sugar transporters". Current Opinion in Cell Biology. 11 (4): 496–502. doi:10.1016/S0955-0674(99)80072-6. PMID 10449337.
- ↑ Bell GI, Burant CF, Takeda J, Gould GW (September 1993). "Structure and function of mammalian facilitative sugar transporters". The Journal of Biological Chemistry. 268 (26): 19161–4. doi:10.1016/S0021-9258(19)36489-0. PMID 8366068.
- ↑ ۲۸٫۰ ۲۸٫۱ Bouché C, Serdy S, Kahn CR, Goldfine AB (October 2004). "The cellular fate of glucose and its relevance in type 2 diabetes". Endocrine Reviews. 25 (5): 807–30. doi:10.1210/er.2003-0026. PMID 15466941.
- ↑ Alfarouk KO, Verduzco D, Rauch C, Muddathir AK, Adil HH, Elhassan GO, et al. (18 December 2014). "Glycolysis, tumor metabolism, cancer growth and dissemination. A new pH-based etiopathogenic perspective and therapeutic approach to an old cancer question". Oncoscience. 1 (12): 777–802. doi:10.18632/oncoscience.109. PMC 4303887. PMID 25621294.
- ↑ Schmidt-Rohr, K. (2015). "Why Combustions Are Always Exothermic, Yielding About 418 kJ per Mole of O2", J. Chem. Educ. 92: 2094-2099. http://dx.doi.org/10.1021/acs.jchemed.5b00333.
- ↑ Wipperman MF, Sampson NS, Thomas ST (2014). "Pathogen roid rage: cholesterol utilization by Mycobacterium tuberculosis". Critical Reviews in Biochemistry and Molecular Biology. 49 (4): 269–93. doi:10.3109/10409238.2014.895700. PMC 4255906. PMID 24611808.
- ↑ Sakami W, Harrington H (1963). "Amino Acid Metabolism". Annual Review of Biochemistry. 32: 355–98. doi:10.1146/annurev.bi.32.070163.002035. PMID 14144484.
- ↑ Brosnan JT (April 2000). "Glutamate, at the interface between amino acid and carbohydrate metabolism". The Journal of Nutrition. 130 (4S Suppl): 988S–90S. doi:10.1093/jn/130.4.988S. PMID 10736367.
- ↑ Young VR, Ajami AM (September 2001). "Glutamine: the emperor or his clothes?". The Journal of Nutrition. 131 (9 Suppl): 2449S–59S, discussion 2486S–7S. doi:10.1093/jn/131.9.2449S. PMID 11533293.
- ↑ Hosler JP, Ferguson-Miller S, Mills DA (2006). "Energy transduction: proton transfer through the respiratory complexes". Annual Review of Biochemistry. 75: 165–87. doi:10.1146/annurev.biochem.75.062003.101730. PMC 2659341. PMID 16756489.
- ↑ Schultz BE, Chan SI (2001). "Structures and proton-pumping strategies of mitochondrial respiratory enzymes" (PDF). Annual Review of Biophysics and Biomolecular Structure. 30: 23–65. doi:10.1146/annurev.biophys.30.1.23. PMID 11340051.
- ↑ Capaldi RA, Aggeler R (March 2002). "Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor". Trends in Biochemical Sciences. 27 (3): 154–60. doi:10.1016/S0968-0004(01)02051-5. PMID 11893513.
- ↑ Friedrich B, Schwartz E (1993). "Molecular biology of hydrogen utilization in aerobic chemolithotrophs". Annual Review of Microbiology. 47: 351–83. doi:10.1146/annurev.mi.47.100193.002031. PMID 8257102.
- ↑ Weber KA, Achenbach LA, Coates JD (October 2006). "Microorganisms pumping iron: anaerobic microbial iron oxidation and reduction". Nature Reviews. Microbiology. 4 (10): 752–64. doi:10.1038/nrmicro1490. PMID 16980937.
- ↑ Jetten MS, Strous M, van de Pas-Schoonen KT, Schalk J, van Dongen UG, van de Graaf AA, et al. (December 1998). "The anaerobic oxidation of ammonium". FEMS Microbiology Reviews. 22 (5): 421–37. doi:10.1111/j.1574-6976.1998.tb00379.x. PMID 9990725.
- ↑ Simon J (August 2002). "Enzymology and bioenergetics of respiratory nitrite ammonification". FEMS Microbiology Reviews. 26 (3): 285–309. doi:10.1111/j.1574-6976.2002.tb00616.x. PMID 12165429.
- ↑ Conrad R (December 1996). "Soil microorganisms as controllers of atmospheric trace gases (H2, CO, CH4, OCS, N2O, and NO)". Microbiological Reviews. 60 (4): 609–40. doi:10.1128/MMBR.60.4.609-640.1996. PMC 239458. PMID 8987358.
- ↑ Barea JM, Pozo MJ, Azcón R, Azcón-Aguilar C (July 2005). "Microbial co-operation in the rhizosphere". Journal of Experimental Botany. 56 (417): 1761–78. doi:10.1093/jxb/eri197. PMID 15911555.
- ↑ van der Meer MT, Schouten S, Bateson MM, Nübel U, Wieland A, Kühl M, et al. (July 2005). "Diel variations in carbon metabolism by green nonsulfur-like bacteria in alkaline siliceous hot spring microbial mats from Yellowstone National Park". Applied and Environmental Microbiology. 71 (7): 3978–86. Bibcode:2005ApEnM..71.3978V. doi:10.1128/AEM.71.7.3978-3986.2005. PMC 1168979. PMID 16000812.
- ↑ Tichi MA, Tabita FR (November 2001). "Interactive control of Rhodobacter capsulatus redox-balancing systems during phototrophic metabolism". Journal of Bacteriology. 183 (21): 6344–54. doi:10.1128/JB.183.21.6344-6354.2001. PMC 100130. PMID 11591679.
- ↑ Allen JP, Williams JC (October 1998). "Photosynthetic reaction centers". FEBS Letters. 438 (1–2): 5–9. doi:10.1016/S0014-5793(98)01245-9. PMID 9821949.
- ↑ Munekage Y, Hashimoto M, Miyake C, Tomizawa K, Endo T, Tasaka M, Shikanai T (June 2004). "Cyclic electron flow around photosystem I is essential for photosynthesis". Nature. 429 (6991): 579–82. Bibcode:2004Natur.429..579M. doi:10.1038/nature02598. PMID 15175756.
- ↑ ۴۸٫۰ ۴۸٫۱ Mandal, Ananya (2009-11-26). "What is Anabolism?". News-Medical.net (به انگلیسی). Retrieved 2020-07-04.
- ↑ Miziorko HM, Lorimer GH (1983). "Ribulose-1,5-bisphosphate carboxylase-oxygenase". Annual Review of Biochemistry. 52: 507–35. doi:10.1146/annurev.bi.52.070183.002451. PMID 6351728.
- ↑ Dodd AN, Borland AM, Haslam RP, Griffiths H, Maxwell K (April 2002). "Crassulacean acid metabolism: plastic, fantastic". Journal of Experimental Botany. 53 (369): 569–80. doi:10.1093/jexbot/53.369.569. PMID 11886877.
- ↑ Hügler M, Wirsen CO, Fuchs G, Taylor CD, Sievert SM (May 2005). "Evidence for autotrophic CO2 fixation via the reductive tricarboxylic acid cycle by members of the epsilon subdivision of proteobacteria". Journal of Bacteriology. 187 (9): 3020–7. doi:10.1128/JB.187.9.3020-3027.2005. PMC 1082812. PMID 15838028.
- ↑ Strauss G, Fuchs G (August 1993). "Enzymes of a novel autotrophic CO2 fixation pathway in the phototrophic bacterium Chloroflexus aurantiacus, the 3-hydroxypropionate cycle". European Journal of Biochemistry. 215 (3): 633–43. doi:10.1111/j.1432-1033.1993.tb18074.x. PMID 8354269.
- ↑ Wood HG (February 1991). "Life with CO or CO2 and H2 as a source of carbon and energy". FASEB Journal. 5 (2): 156–63. doi:10.1096/fasebj.5.2.1900793. PMID 1900793.
- ↑ Shively JM, van Keulen G, Meijer WG (1998). "Something from almost nothing: carbon dioxide fixation in chemoautotrophs". Annual Review of Microbiology. 52: 191–230. doi:10.1146/annurev.micro.52.1.191. PMID 9891798.
- ↑ Boiteux A, Hess B (June 1981). "Design of glycolysis". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 293 (1063): 5–22. Bibcode:1981RSPTB.293....5B. doi:10.1098/rstb.1981.0056. PMID 6115423.
- ↑ Pilkis SJ, el-Maghrabi MR, Claus TH (June 1990). "Fructose-2,6-bisphosphate in control of hepatic gluconeogenesis. From metabolites to molecular genetics". Diabetes Care. 13 (6): 582–99. doi:10.2337/diacare.13.6.582. PMID 2162755.
- ↑ ۵۷٫۰ ۵۷٫۱ Ensign SA (July 2006). "Revisiting the glyoxylate cycle: alternate pathways for microbial acetate assimilation". Molecular Microbiology. 61 (2): 274–6. doi:10.1111/j.1365-2958.2006.05247.x. PMID 16856935.
- ↑ Finn PF, Dice JF (2006). "Proteolytic and lipolytic responses to starvation". Nutrition. 22 (7–8): 830–44. doi:10.1016/j.nut.2006.04.008. PMID 16815497.
- ↑ ۵۹٫۰ ۵۹٫۱ Kornberg HL, Krebs HA (May 1957). "Synthesis of cell constituents from C2-units by a modified tricarboxylic acid cycle". Nature. 179 (4568): 988–91. Bibcode:1957Natur.179..988K. doi:10.1038/179988a0. PMID 13430766.
- ↑ Evans, Rhys D.; Heather, Lisa C. (June 2016). "Metabolic pathways and abnormalities". Surgery (Oxford). 34 (6): 266–272. doi:10.1016/j.mpsur.2016.03.010. ISSN 0263-9319.
- ↑ Opdenakker G, Rudd PM, Ponting CP, Dwek RA (November 1993). "Concepts and principles of glycobiology". FASEB Journal. 7 (14): 1330–7. doi:10.1096/fasebj.7.14.8224606. PMID 8224606.
- ↑ McConville MJ, Menon AK (2000). "Recent developments in the cell biology and biochemistry of glycosylphosphatidylinositol lipids (review)". Molecular Membrane Biology. 17 (1): 1–16. doi:10.1080/096876800294443. PMID 10824734.
- ↑ Chirala SS, Wakil SJ (November 2004). "Structure and function of animal fatty acid synthase". Lipids. 39 (11): 1045–53. doi:10.1007/s11745-004-1329-9. PMID 15726818.
- ↑ White SW, Zheng J, Zhang YM (2005). "The structural biology of type II fatty acid biosynthesis". Annual Review of Biochemistry. 74: 791–831. doi:10.1146/annurev.biochem.74.082803.133524. PMID 15952903.
- ↑ Ohlrogge JB, Jaworski JG (June 1997). "Regulation of Fatty Acid Synthesis". Annual Review of Plant Physiology and Plant Molecular Biology. 48: 109–136. doi:10.1146/annurev.arplant.48.1.109. PMID 15012259.
- ↑ Dubey VS, Bhalla R, Luthra R (September 2003). "An overview of the non-mevalonate pathway for terpenoid biosynthesis in plants" (PDF). Journal of Biosciences. 28 (5): 637–46. doi:10.1007/BF02703339. PMID 14517367. Archived from the original (PDF) on 15 April 2007.
- ↑ ۶۷٫۰ ۶۷٫۱ Kuzuyama T, Seto H (April 2003). "Diversity of the biosynthesis of the isoprene units". Natural Product Reports. 20 (2): 171–83. doi:10.1039/b109860h. PMID 12735695.
- ↑ Grochowski LL, Xu H, White RH (May 2006). "Methanocaldococcus jannaschii uses a modified mevalonate pathway for biosynthesis of isopentenyl diphosphate". Journal of Bacteriology. 188 (9): 3192–8. doi:10.1128/JB.188.9.3192-3198.2006. PMC 1447442. PMID 16621811.
- ↑ Lichtenthaler HK (June 1999). "The 1-Deoxy-D-Xylulose-5-Phosphate Pathway of Isoprenoid Biosynthesis in Plants". Annual Review of Plant Physiology and Plant Molecular Biology. 50: 47–65. doi:10.1146/annurev.arplant.50.1.47. PMID 15012203.
- ↑ ۷۰٫۰ ۷۰٫۱ Schroepfer GJ (1981). "Sterol biosynthesis". Annual Review of Biochemistry. 50: 585–621. doi:10.1146/annurev.bi.50.070181.003101. PMID 7023367.
- ↑ Lees ND, Skaggs B, Kirsch DR, Bard M (March 1995). "Cloning of the late genes in the ergosterol biosynthetic pathway of Saccharomyces cerevisiae--a review". Lipids. 30 (3): 221–6. doi:10.1007/BF02537824. PMID 7791529.
- ↑ Himmelreich R, Hilbert H, Plagens H, Pirkl E, Li BC, Herrmann R (November 1996). "Complete sequence analysis of the genome of the bacterium Mycoplasma pneumoniae". Nucleic Acids Research. 24 (22): 4420–49. doi:10.1093/nar/24.22.4420. PMC 146264. PMID 8948633.
- ↑ Ibba M, Söll D (May 2001). "The renaissance of aminoacyl-tRNA synthesis". EMBO Reports. 2 (5): 382–7. doi:10.1093/embo-reports/kve095. PMC 1083889. PMID 11375928. Archived from the original on 1 May 2011.
- ↑ Lengyel P, Söll D (June 1969). "Mechanism of protein biosynthesis". Bacteriological Reviews. 33 (2): 264–301. doi:10.1128/MMBR.33.2.264-301.1969. PMC 378322. PMID 4896351.
- ↑ ۷۵٫۰ ۷۵٫۱ Rudolph FB (January 1994). "The biochemistry and physiology of nucleotides". The Journal of Nutrition. 124 (1 Suppl): 124S–127S. doi:10.1093/jn/124.suppl_1.124S. PMID 8283301. Zrenner R, Stitt M, Sonnewald U, Boldt R (2006). "Pyrimidine and purine biosynthesis and degradation in plants". Annual Review of Plant Biology. 57: 805–36. doi:10.1146/annurev.arplant.57.032905.105421. PMID 16669783.
- ↑ Stasolla C, Katahira R, Thorpe TA, Ashihara H (November 2003). "Purine and pyrimidine nucleotide metabolism in higher plants". Journal of Plant Physiology. 160 (11): 1271–95. doi:10.1078/0176-1617-01169. PMID 14658380.
- ↑ Davies O, Mendes P, Smallbone K, Malys N (April 2012). "Characterisation of multiple substrate-specific (d)ITP/(d)XTPase and modelling of deaminated purine nucleotide metabolism" (PDF). BMB Reports. 45 (4): 259–64. doi:10.5483/BMBRep.2012.45.4.259. PMID 22531138.
- ↑ Smith JL (December 1995). "Enzymes of nucleotide synthesis". Current Opinion in Structural Biology. 5 (6): 752–7. doi:10.1016/0959-440X(95)80007-7. PMID 8749362.
- ↑ Testa B, Krämer SD (October 2006). "The biochemistry of drug metabolism--an introduction: part 1. Principles and overview". Chemistry & Biodiversity. 3 (10): 1053–101. doi:10.1002/cbdv.200690111. PMID 17193224.
- ↑ Danielson PB (December 2002). "The cytochrome P450 superfamily: biochemistry, evolution and drug metabolism in humans". Current Drug Metabolism. 3 (6): 561–97. doi:10.2174/1389200023337054. PMID 12369887.
- ↑ King CD, Rios GR, Green MD, Tephly TR (September 2000). "UDP-glucuronosyltransferases". Current Drug Metabolism. 1 (2): 143–61. doi:10.2174/1389200003339171. PMID 11465080.
- ↑ Sheehan D, Meade G, Foley VM, Dowd CA (November 2001). "Structure, function and evolution of glutathione transferases: implications for classification of non-mammalian members of an ancient enzyme superfamily". The Biochemical Journal. 360 (Pt 1): 1–16. doi:10.1042/0264-6021:3600001. PMC 1222196. PMID 11695986.
- ↑ Galvão TC, Mohn WW, de Lorenzo V (October 2005). "Exploring the microbial biodegradation and biotransformation gene pool". Trends in Biotechnology. 23 (10): 497–506. doi:10.1016/j.tibtech.2005.08.002. PMID 16125262.
- ↑ Janssen DB, Dinkla IJ, Poelarends GJ, Terpstra P (December 2005). "Bacterial degradation of xenobiotic compounds: evolution and distribution of novel enzyme activities" (PDF). Environmental Microbiology. 7 (12): 1868–82. doi:10.1111/j.1462-2920.2005.00966.x. PMID 16309386.
- ↑ Davies KJ (1995). "Oxidative stress: the paradox of aerobic life". Biochemical Society Symposium. 61: 1–31. doi:10.1042/bss0610001. PMID 8660387.
- ↑ Tu BP, Weissman JS (February 2004). "Oxidative protein folding in eukaryotes: mechanisms and consequences". The Journal of Cell Biology. 164 (3): 341–6. doi:10.1083/jcb.200311055. PMC 2172237. PMID 14757749.
- ↑ Sies H (March 1997). "Oxidative stress: oxidants and antioxidants". Experimental Physiology. 82 (2): 291–5. doi:10.1113/expphysiol.1997.sp004024. PMID 9129943.
- ↑ Vertuani S, Angusti A, Manfredini S (2004). "The antioxidants and pro-antioxidants network: an overview". Current Pharmaceutical Design. 10 (14): 1677–94. doi:10.2174/1381612043384655. PMID 15134565.
- ↑ von Stockar U, Liu J (August 1999). "Does microbial life always feed on negative entropy? Thermodynamic analysis of microbial growth". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1412 (3): 191–211. doi:10.1016/S0005-2728(99)00065-1. PMID 10482783.
- ↑ Demirel Y, Sandler SI (June 2002). "Thermodynamics and bioenergetics". Biophysical Chemistry. 97 (2–3): 87–111. doi:10.1016/S0301-4622(02)00069-8. PMID 12050002.
- ↑ Albert R (November 2005). "Scale-free networks in cell biology". Journal of Cell Science. 118 (Pt 21): 4947–57. arXiv:q-bio/0510054. Bibcode:2005q.bio....10054A. doi:10.1242/jcs.02714. PMID 16254242.
- ↑ Brand MD (January 1997). "Regulation analysis of energy metabolism". The Journal of Experimental Biology. 200 (Pt 2): 193–202. doi:10.1242/jeb.200.2.193. PMID 9050227.
- ↑ Soyer OS, Salathé M, Bonhoeffer S (January 2006). "Signal transduction networks: topology, response and biochemical processes". Journal of Theoretical Biology. 238 (2): 416–25. Bibcode:2006JThBi.238..416S. doi:10.1016/j.jtbi.2005.05.030. PMID 16045939.
- ↑ ۹۴٫۰ ۹۴٫۱ Salter M, Knowles RG, Pogson CI (1994). "Metabolic control". Essays in Biochemistry. 28: 1–12. PMID 7925313.
- ↑ Westerhoff HV, Groen AK, Wanders RJ (January 1984). "Modern theories of metabolic control and their applications (review)". Bioscience Reports. 4 (1): 1–22. doi:10.1007/BF01120819. PMID 6365197.
- ↑ Fell DA, Thomas S (October 1995). "Physiological control of metabolic flux: the requirement for multisite modulation". The Biochemical Journal. 311 (Pt 1) (Pt 1): 35–9. doi:10.1042/bj3110035. PMC 1136115. PMID 7575476.
- ↑ Hendrickson WA (November 2005). "Transduction of biochemical signals across cell membranes". Quarterly Reviews of Biophysics. 38 (4): 321–30. doi:10.1017/S0033583506004136. PMID 16600054.
- ↑ Cohen P (December 2000). "The regulation of protein function by multisite phosphorylation--a 25 year update". Trends in Biochemical Sciences. 25 (12): 596–601. doi:10.1016/S0968-0004(00)01712-6. PMID 11116185.
- ↑ Lienhard GE, Slot JW, James DE, Mueckler MM (January 1992). "How cells absorb glucose". Scientific American. 266 (1): 86–91. Bibcode:1992SciAm.266a..86L. doi:10.1038/scientificamerican0192-86. PMID 1734513.
- ↑ Roach PJ (March 2002). "Glycogen and its metabolism". Current Molecular Medicine. 2 (2): 101–20. doi:10.2174/1566524024605761. PMID 11949930.
- ↑ Newgard CB, Brady MJ, O'Doherty RM, Saltiel AR (December 2000). "Organizing glucose disposal: emerging roles of the glycogen targeting subunits of protein phosphatase-1" (PDF). Diabetes. 49 (12): 1967–77. doi:10.2337/diabetes.49.12.1967. PMID 11117996.
- ↑ Smith E, Morowitz HJ (September 2004). "Universality in intermediary metabolism". Proceedings of the National Academy of Sciences of the United States of America. 101 (36): 13168–73. Bibcode:2004PNAS..10113168S. doi:10.1073/pnas.0404922101. PMC 516543. PMID 15340153.
- ↑ Romano AH, Conway T (1996). "Evolution of carbohydrate metabolic pathways". Research in Microbiology. 147 (6–7): 448–55. doi:10.1016/0923-2508(96)83998-2. PMID 9084754.
- ↑ Ouzounis C, Kyrpides N (July 1996). "The emergence of major cellular processes in evolution". FEBS Letters. 390 (2): 119–23. doi:10.1016/0014-5793(96)00631-X. PMID 8706840.
- ↑ Ebenhöh O, Heinrich R (January 2001). "Evolutionary optimization of metabolic pathways. Theoretical reconstruction of the stoichiometry of ATP and NADH producing systems". Bulletin of Mathematical Biology. 63 (1): 21–55. doi:10.1006/bulm.2000.0197. PMID 11146883.
- ↑ Meléndez-Hevia E, Waddell TG, Cascante M (September 1996). "The puzzle of the Krebs citric acid cycle: assembling the pieces of chemically feasible reactions, and opportunism in the design of metabolic pathways during evolution". Journal of Molecular Evolution. 43 (3): 293–303. Bibcode:1996JMolE..43..293M. doi:10.1007/BF02338838. PMID 8703096.
- ↑ Caetano-Anollés G, Kim HS, Mittenthal JE (May 2007). "The origin of modern metabolic networks inferred from phylogenomic analysis of protein architecture". Proceedings of the National Academy of Sciences of the United States of America. 104 (22): 9358–63. Bibcode:2007PNAS..104.9358C. doi:10.1073/pnas.0701214104. PMC 1890499. PMID 17517598.
- ↑ Schmidt S, Sunyaev S, Bork P, Dandekar T (June 2003). "Metabolites: a helping hand for pathway evolution?". Trends in Biochemical Sciences. 28 (6): 336–41. doi:10.1016/S0968-0004(03)00114-2. PMID 12826406.
- ↑ Light S, Kraulis P (February 2004). "Network analysis of metabolic enzyme evolution in Escherichia coli". BMC Bioinformatics. 5: 15. doi:10.1186/1471-2105-5-15. PMC 394313. PMID 15113413. Alves R, Chaleil RA, Sternberg MJ (July 2002). "Evolution of enzymes in metabolism: a network perspective". Journal of Molecular Biology. 320 (4): 751–70. doi:10.1016/S0022-2836(02)00546-6. PMID 12095253.
- ↑ Kim HS, Mittenthal JE, Caetano-Anollés G (July 2006). "MANET: tracing evolution of protein architecture in metabolic networks". BMC Bioinformatics. 7: 351. doi:10.1186/1471-2105-7-351. PMC 1559654. PMID 16854231.
- ↑ Teichmann SA, Rison SC, Thornton JM, Riley M, Gough J, Chothia C (December 2001). "Small-molecule metabolism: an enzyme mosaic". Trends in Biotechnology. 19 (12): 482–6. doi:10.1016/S0167-7799(01)01813-3. PMID 11711174.
- ↑ Spirin V, Gelfand MS, Mironov AA, Mirny LA (June 2006). "A metabolic network in the evolutionary context: multiscale structure and modularity". Proceedings of the National Academy of Sciences of the United States of America. 103 (23): 8774–9. Bibcode:2006PNAS..103.8774S. doi:10.1073/pnas.0510258103. PMC 1482654. PMID 16731630.
- ↑ Lawrence JG (December 2005). "Common themes in the genome strategies of pathogens". Current Opinion in Genetics & Development. 15 (6): 584–8. doi:10.1016/j.gde.2005.09.007. PMID 16188434. Wernegreen JJ (December 2005). "For better or worse: genomic consequences of intracellular mutualism and parasitism". Current Opinion in Genetics & Development. 15 (6): 572–83. doi:10.1016/j.gde.2005.09.013. PMID 16230003.
- ↑ Pál C, Papp B, Lercher MJ, Csermely P, Oliver SG, Hurst LD (March 2006). "Chance and necessity in the evolution of minimal metabolic networks". Nature. 440 (7084): 667–70. Bibcode:2006Natur.440..667P. doi:10.1038/nature04568. PMID 16572170.
- ↑ Rennie MJ (November 1999). "An introduction to the use of tracers in nutrition and metabolism". The Proceedings of the Nutrition Society. 58 (4): 935–44. doi:10.1017/S002966519900124X. PMID 10817161.
- ↑ Phair RD (December 1997). "Development of kinetic models in the nonlinear world of molecular cell biology". Metabolism. 46 (12): 1489–95. doi:10.1016/S0026-0495(97)90154-2. PMID 9439549.
- ↑ Sterck L, Rombauts S, Vandepoele K, Rouzé P, Van de Peer Y (April 2007). "How many genes are there in plants (... and why are they there)?". Current Opinion in Plant Biology. 10 (2): 199–203. doi:10.1016/j.pbi.2007.01.004. PMID 17289424.
- ↑ Borodina I, Nielsen J (June 2005). "From genomes to in silico cells via metabolic networks". Current Opinion in Biotechnology. 16 (3): 350–5. doi:10.1016/j.copbio.2005.04.008. PMID 15961036.
- ↑ Gianchandani EP, Brautigan DL, Papin JA (May 2006). "Systems analyses characterize integrated functions of biochemical networks". Trends in Biochemical Sciences. 31 (5): 284–91. doi:10.1016/j.tibs.2006.03.007. PMID 16616498.
- ↑ Duarte NC, Becker SA, Jamshidi N, Thiele I, Mo ML, Vo TD, et al. (February 2007). "Global reconstruction of the human metabolic network based on genomic and bibliomic data". Proceedings of the National Academy of Sciences of the United States of America. 104 (6): 1777–82. Bibcode:2007PNAS..104.1777D. doi:10.1073/pnas.0610772104. PMC 1794290. PMID 17267599.
- ↑ Goh KI, Cusick ME, Valle D, Childs B, Vidal M, Barabási AL (May 2007). "The human disease network". Proceedings of the National Academy of Sciences of the United States of America. 104 (21): 8685–90. Bibcode:2007PNAS..104.8685G. doi:10.1073/pnas.0701361104. PMC 1885563. PMID 17502601.
- ↑ Lee DS, Park J, Kay KA, Christakis NA, Oltvai ZN, Barabási AL (July 2008). "The implications of human metabolic network topology for disease comorbidity". Proceedings of the National Academy of Sciences of the United States of America. 105 (29): 9880–5. Bibcode:2008PNAS..105.9880L. doi:10.1073/pnas.0802208105. PMC 2481357. PMID 18599447.
- ↑ Csete M, Doyle J (September 2004). "Bow ties, metabolism and disease". Trends in Biotechnology. 22 (9): 446–50. doi:10.1016/j.tibtech.2004.07.007. PMID 15331224.
- ↑ Ma HW, Zeng AP (July 2003). "The connectivity structure, giant strong component and centrality of metabolic networks". Bioinformatics. 19 (11): 1423–30. CiteSeerX 10.1.1.605.8964. doi:10.1093/bioinformatics/btg177. PMID 12874056.
- ↑ Zhao J, Yu H, Luo JH, Cao ZW, Li YX (August 2006). "Hierarchical modularity of nested bow-ties in metabolic networks". BMC Bioinformatics. 7: 386. arXiv:q-bio/0605003. Bibcode:2006q.bio.....5003Z. doi:10.1186/1471-2105-7-386. PMC 1560398. PMID 16916470.
- ↑ Thykaer J, Nielsen J (January 2003). "Metabolic engineering of beta-lactam production". Metabolic Engineering. 5 (1): 56–69. doi:10.1016/S1096-7176(03)00003-X. PMID 12749845.
- ↑ González-Pajuelo M, Meynial-Salles I, Mendes F, Andrade JC, Vasconcelos I, Soucaille P (2005). "Metabolic engineering of Clostridium acetobutylicum for the industrial production of 1,3-propanediol from glycerol". Metabolic Engineering. 7 (5–6): 329–36. doi:10.1016/j.ymben.2005.06.001. PMID 16095939.
{{cite journal}}:|hdl-access=requires|hdl=(help) - ↑ Krämer M, Bongaerts J, Bovenberg R, Kremer S, Müller U, Orf S, et al. (October 2003). "Metabolic engineering for microbial production of shikimic acid". Metabolic Engineering. 5 (4): 277–83. doi:10.1016/j.ymben.2003.09.001. PMID 14642355.
- ↑ Koffas M, Roberge C, Lee K, Stephanopoulos G (1999). "Metabolic engineering". Annual Review of Biomedical Engineering. 1: 535–57. doi:10.1146/annurev.bioeng.1.1.535. PMID 11701499.
- ↑ "metabolism | Origin and meaning of metabolism by Online Etymology Dictionary". www.etymonline.com (به انگلیسی). Retrieved 2020-07-23.
- ↑ Eknoyan G (1999). "Santorio Sanctorius (1561-1636) - founding father of metabolic balance studies". American Journal of Nephrology. 19 (2): 226–33. doi:10.1159/000013455. PMID 10213823.
- ↑ Manchester KL (December 1995). "Louis Pasteur (1822-1895)--chance and the prepared mind". Trends in Biotechnology. 13 (12): 511–5. doi:10.1016/S0167-7799(00)89014-9. PMID 8595136.
- ↑ Kinne-Saffran E, Kinne RK (1999). "Vitalism and synthesis of urea. From Friedrich Wöhler to Hans A. Krebs". American Journal of Nephrology. 19 (2): 290–4. doi:10.1159/000013463. PMID 10213830.
- ↑ Eduard Buchner's 1907 Nobel lecture at http://nobelprize.org Accessed 20 March 2007
- ↑ Kornberg H (December 2000). "Krebs and his trinity of cycles". Nature Reviews. Molecular Cell Biology. 1 (3): 225–8. doi:10.1038/35043073. PMID 11252898.
- ↑ Krebs HA, Henseleit K (1932). "Untersuchungen über die Harnstoffbildung im tierkorper". Z. Physiol. Chem. 210 (1–2): 33–66. doi:10.1515/bchm2.1932.210.1-2.33.
- ↑ Krebs HA, Johnson WA (April 1937). "Metabolism of ketonic acids in animal tissues". The Biochemical Journal. 31 (4): 645–60. doi:10.1042/bj0310645. PMC 1266984. PMID 16746382.
