آرانای پیامرسان

آرانای پیامرسان یا آرانای پیک (به انگلیسی: Messenger RNA یا به اختصار mRNA) گونهٔ مهمی از آرانایها است که یک الگوی ژنتیکی را برای ساخت یک محصول پروتئینی (پلیپپتید)، بهصورت رمزه (کدون) (توالی ۳ نوکلئوتیدی در آرانای پیک) رمزگذاری میکند. آرانای پیک، از یک الگوی دیانای، رونویسی (ساخته) میشود و اطلاعات رمزگذاریشده را به مکانهای ساخت پروتئین یعنی ریبوزومها، میبرد.
در ریبوزوم، رمزههای آرانای پیک تعیین میکنند که کدام آمینواسیدها باید در ساختار پلیپپتید قرار بگیرند. به ساخته شدن پلیپپتید از روی اطلاعات آرانای پیک، ترجمه میگویند. آرانایهای حامل دارای آمینواسید، با خواندن اطلاعات رمزهها آمینواسید مناسب را انتخاب میکنند و فرایند ترجمه را انجام میدهند.[۱]
در هر رمزه، یک آمینواسید، رمز شدهاست. البته رمزههای پایان هیچ آمینواسیدی را رمز نمیکنند بلکه فرایند ترجمه را به پایان میرسانند.[۱]
این فرایند، به انواع دیگری از آرانای نیز نیاز دارد: آرانای حامل (tRNA) که کار شناسایی رمزهها را انجام میدهد و آمینواسیدهای متناظر را تأمین میکند و همچنین آرانای ریبوزومی (rRNA)، که جزء مرکزی در دستگاه تولید پروتئین ریبوزوم است.[۱]
سنتز، پردازش و عملکرد[ویرایش]
بهطور خلاصه عملکرد آرانای پیک با رونویسی از روی دیانای آغاز شده و با از هم پاشیدگی تمام میشود. در طول زندگی، آرانای پیک ممکن است که پردازش شده، تغییر یابد یا قبل از ترجمه، منتقل شود. مولکولهای آرانای پیک یوکاریوتی، معمولاً نیازمند پردازش و انتقال اطلاعات بیشتری هستند، در حالیکه در پروکاریوتها اینگونه نیست.
رونویسی[ویرایش]
در طول رونویسی، آرانای پلیمراز (در نقش آنزیم)، یک کپی از دیانای به آرانای پیک درست میکند. این فرایند در یوکاریوتها و پروکاریوتها مانند هم است. یک تفاوت مهم این است که آرانای پلیمرازهای پروکاریوتی در ارتباط با آنزیمهای پردازشی آرانای پیک در طول رونویسی هستند تا پردازش بتواند پس از شروع رونویسی به سرعت پیشرفت نماید. محصول چندروزه، پردازش نشده یا بهصورت جزئی پردازش شده و پیشساز آرانای پیک یا آرانای پیک نابالغ (pre-mRNA) نام میگیرد و هنگامیکه پردازش کامل شود، محصول، آرانای پیک بالغ نامیده میشود.

پردازش پیشساز آرانای پیک یوکاریوتی[ویرایش]
پردازش آرانای پیک در یوکاریوتها، باکتریها و پروکاریوتها بسیار متفاوت است. آرانای پیک پروکاریوتی پس از رونویسی بالغ میشود و بهجز در مواردی اندک، به هیچ پردازشی نیاز ندارد. پیشساز آرانای پیک یوکاریوتی اما نیازمند پردازش گستردهای است.
کلاهکگذاری '۵[ویرایش]
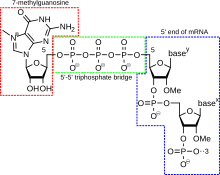
کلاهک '۵ یک نوکلئوتید گوانین تغییریافتهاست که به انتهای ۵' یک آرانای پیک یوکاریوتی، کمی پس از شروع رونویسی اضافه میشود. کلاهک ۵َ شامل یک پایانهٔ ۷-متیلگوانوزین است که توسط یک پیوند ۵َ-۵َ-تریفسفات به نخستین نوکلئوتید ترجمهشده متصل میشود. حضور آن برای شناسایی توسط ریبوزوم و محافظت در برابر ریبونوکلئازها حیاتی است. کلاهک با رونویسی همراه میشود و رونویسی همکارانه ایجاد میشود بهطوریکه هرکدام روی دیگری تأثیر میگذارد. کمی پس از شروع رونویسی، انتهای ۵' مربوط به آرانای پیک که در حال سنتز شدن است توسط یک کمپلکس سنتزکنندهٔ کلاهک که در ارتباط با آرانای پلیمراز است، محدود میشود. این کمپلکس آنزیمی، واکنشهای شیمیایی را که برای کلاهکگذاری '۵ لازم است کاتالیز میکند. سنتز بهعنوان یک واکنش چندمرحلهای بیوشیمیایی به پیش میرود.
پیرایش آرانای[ویرایش]
پیرایش آرانای (RNA Splicing) فرایندی است که در آن پیشسازهای آرانای پیک، تغییر یافته تا امتدادهای خاصی از رشتههای کُدنشده که اینترون نامیده میشوند را حذف کند. امتدادهایی که باقی میمانند شامل رشتههای کدشدهٔ پروتئینی هستند و اگزون نامیده میشوند. گاهی پیشسازهای آرانای پیک ممکن است که به چندین روش مختلف پیرایش شوند که سبب میشود تا یک ژن چندین پروتئین را رمز کند. این فرایند، پیرایش جایگزین نامیده میشود. پیرایش، معمولاً توسط یک کمپلکس پروتئین-آرانای که اسپلایسوزوم نامیده میشود انجام میشود اما بعضی از مولکولهای آرانای نیز توانایی کاتالیز پیرایش خود را دارند.
ویرایش آرانای[ویرایش]
در بعضی موارد، یک آرانای پیک ویرایش میشود و ترکیب نوکلئوتیدی آن تغییر مییابد. یک مثال در انسانها آرانای پیک آپولیپوپروتئین ب است که در بعضی از بافتها ویرایش میشود اما در بقیه نمیشود. ویرایش، یک کدون خاتمه زودرس ایجاد میکند که در هنگام ترجمه، یک پروتئین کوتاهتر تولید میکند.
پلیآدنیله شدن[ویرایش]
پلیآدنیله شدن پیوند شیمیایی یک نیمهٔ پلیآدنیلی با یک مولکول آرانای پیامرسان است. در یوکاریوتها، بیشتر مولکولهای آرانای پیامرسان، در انتهای ۳' پلیآدنیله هستند. دنبالهٔ پلیآدنیله (poly(A) tail) و پروتئینی که به آن متصل میشود، آرانای پیامرسان را از ازهمپاشیدگی بهوسیلهٔ اگزونوکلئازها محافظت میکند. پُلیآدنیلاسیون همچنین برای اتمام رونویسی، خارج کردن آرانای پیامرسان از هسته و ترجمه کردن مهم است. آرانای پیامرسان همچنین میتواند در ارگانیسمهای پروکاریوتی پلیآدنیله شود، جایی که دنبالهٔ پلیآدنیله فعالیت کرده تا ازهمپاشیدگی اگزونوکلئولیتیک را مهیا کند، بهجای آنکه مانع آن شود. پُلیآدنیلاسیون، در مدتزمان و همچنین بلافاصله پس از رونویسی دیانای در آرانای رخ میدهد. پس از آنکه رونویسی به اتمام رسید، زنجیرهٔ آرانای پیامرسان در حین فعالیت کمپلکس اندونوکلئاز که همراه با آرانای پلیمراز است، شکسته میشود. پس از آنکه آرانای پیامرسان شکسته شد، در حدود ۲۵۰ اسیدآمینه آدنین به انتهای ۳' آزاد در محل شکستن اضافه میشوند. این واکنش، بهوسیلهٔ پلیمراز پلیآدنیلیزه کاتالیز میشود. همانند پیرایش جایگزین، بیش از یکی از انواع پُلیآدنیلاسیون یک آرانای پیامرسان میتواند موجود باشد.
انتقال[ویرایش]
تفاوت دیگر یوکاریوتها و پروکاریوتها در انتقال آرانای پیامرسان است، چون رونویسی و ترجمهٔ یوکاریوتی از همدیگر جدا هستند، آرانایهای پیامرسان یوکاریوتی باید از هسته سلول به سیتوپلاسم خارج شوند. آرانایهای پیامرسان بالغ بهوسیلهٔ تغییرات پردازششدهشان شناسایی میشوند و سپس از طریق منافذ هستهای خارج میشوند.[۲] در نورونها، آرانای پیامرسان باید از جسم سلولی به دندریتها انتقال یابد، جایی که ترجمهٔ محلی در پاسخ به محرک خارجی رخ میدهد.[۳] بسیاری از پیامها توسط «زیپکد» ها که انتقالشان را به یک مکان خاص در نظر میگیرند، نشانهگذاری میشوند.[۴]
ترجمه[ویرایش]
از آنجایی که آرانای پیامرسان پروکاریوتی نیازی ندارد که پردازش شود یا انتقال پیدا کند، ترجمه توسط ریبوزومها میتواند سریعاً پس از پایان رونویسی آغاز شود؛ بنابراین میتوان گفت که ترجمهٔ پروکاریوتی با رونویسی همراه میشود و رونویسی همکارانه رخ میدهد.
آرانای پیامرسان یوکاریوتی که پردازش شده و به سیتوپلاسم منتقل شدهاست (به عبارت دیگر آرانای پیامرسان بالغ)، میتواند توسط ریبوزوم ترجمه شود. ترجمه ممکن است که در ریبوزومهای آزاد شناور در سیتوپلاسم رخ دهد یا مستقیماً توسط ذرهٔ شناسایی سیگنال در شبکهٔ آندوپلاسمی اتفاق افتد؛ بنابراین برخلاف پروکاریوتها، ترجمهٔ یوکاریوتی بهطور مستقیم با رونویسی همراه نیست.
ساختار[ویرایش]
کلاهک ۵'[ویرایش]
کلاهک ۵' یک نوکلئوتید گوانین تغییر یافتهاست که به پیشساز آرانای پیامرسان با استفاده از یک پیوند ۵َ-۵َ-فسفات اضافه شدهاست. این تغییر برای شناسایی و ضمیمهٔ درست آرانای پیامرسان به ریبوزوم، حیاتی است همانطور که محافظت از ۵' اگزونوکلئاز مهم است. همچنین برای فرایندهای حیاتی دیگر همچون پیرایش و انتقال مهم است.
مناطق کدینگ[ویرایش]
مناطق کدینگ از کدونها تشکیل شدهاست که این مناطق بهوسیلهٔ ریبوزومها (در یوکاریوتها معمولاً به یکی و در پروکاریوتها معمولاً به چند) پروتئین، رمزگشایی شده و ترجمه میشوند. مناطق کدینگ با یک کدون آغاز شروع شده و با یک کدون خاتمه پایان مییابند. بهطور کلی کدون آغازین، سهتایی AUG و کدون توقف، UAA, UAG یا UGA است. مناطق کدینگ تمایل دارند که بهوسیلهٔ جفتبازهای داخلی ایجاد شوند که این مدل مانع از ازهمپاشیدگی میشود.[۵][۶] علاوه بر اینکه کدینگ پروتئینی هستند، قسمتهایی از مناطق کدینگ ممکن است که به عنوان رشتههای تنظیمی در پیشساز آرانای پیامرسان باشند.
مناطق ترجمهنشده[ویرایش]
مناطق ترجمهنشده (UTRs) بخشهایی از آرانای پیامرسان هستند که پیش از کدون آغازین و پس از کدون خاتمه قرار دارند که ترجمه نشدهاند که به ترتیب منطقهٔ ترجمهنشدهٔ ۵' (5' UTR) و منطقهٔ ترجمهنشدهٔ ۳' (3' UTR) نامگذاری میشوند. این مناطق با منطقهٔ کدینگ رونویسی میشوند و بنابراین اگزون هستند چون در آرانای پیامرسان بالغ موجود هستند. چندین نقش در بیان ژن مربوط به مناطق ترجمهنشدهاست، همچون پایداری آرانای پیامرسان، محلی نمودن آرانای پیامرسان و کارایی ترجمهای. توانمندی یک UTR برای انجام این اعمال بستگی به رشتهٔ UTR دارد و در بین آرانایهای پیامرسان میتواند متفاوت باشد. پایداری آرانایهای پیامرسان توسط 5' UTR یا 3'UTR بر حسب تفاوت در وابستگی برای آنزیمهای کاهشی آرانای که ریبونوکلئاز نامیده میشوند کنترل میشوند و همچنین نیز برای پروتئینهای کمکی که میتوانند کاهش آرانای را بیشتر کرده یا از آن جلوگیری به عمل آورند.
کارایی ترجمهای که گاهی شامل ممانعت کامل از ترجمه است توسط UTRها کنترل میشود. پروتئینهایی که به ۳' یا ۵' UTR متصل میشوند ممکن است توسط تأثیری که بر توانایی ریبوزوم در اتصال به آرانای پیامرسان دارند در ترجمه تأثیرگذار باشند. ریزآرانایهایی که به 3'UTR نیز متصل میشوند ممکن است در کارایی ترجمهای یا پایداری آرانای پیامرسان تأثیر بگذارند. محلیسازی سیتوپلاسمی آرانای پیامرسان بهعنوان عملکردی از 3' UTR در نظر گرفته میشود. پروتئینهایی که در مناطق خاصی از سلول مورد نیاز هستند، میتوانند در آنجا ترجمه شوند، در این موارد، 3'UTR ممکن است شامل رشتههایی باشد که اجازه دهد تا رونویسی در این محل برای ترجمه، محلی شود. بعضی از عناصر که در مناطق ترجمهنشده قرار دارند، هنگامی که به آرانای، رونویسی میشوند، مشخصهای با عنوان ساختار دوم ایجاد میکنند. این عناصر، در تنظیم آرانایهای پیامرسان دخالت میکنند.
دنبالهٔ پلی (آ)[ویرایش]
دنباله پلی (آ) ۳' یک رشتهٔ بلند از نوکلئوتیدهای آدنین (حدود ۲۰۰ آدنین) است که به انتهای ۳' پیشساز آرانای پیامرسان اضافه میشود. این دنباله، خارج شدن از هسته و ترجمه را افزایش داده و آرانای پیامرسان را از ازهمپاشیدگی محافظت میکند.
آرانای پیامرسان تکژنی و چندژنی[ویرایش]
هنگامی که آرانای پیامرسان دارای اطلاعات ژنتیکی یک ژن است و تنها یک پروتئین را ترجمه میکند، آرانای پیامرسان تکژنی نام میگیرد که این موضوع، در مورد بیشتر آرانایهای پیامرسان یوکاریوتی صدق میکند.[۷][۸] در مقابل، آرانای پیامرسان چندژنی یا پلیسیسترونیک، اطلاعات چندین ژن را حمل میکند که به چندین پروتئین ترجمه میشود.
حلقوی شدن آرانای پیامرسان[ویرایش]
در یوکاریوتها مولکولهای آرانای پیامرسان برحسب تعامل مابین cap binding complex و poly(A)-binding protein تشکیل ساختارهای حلقهای میدهند.[۹] حلقوی شدن سبب پیشرفت در بازسازی ریبوزومها بر روی یک پیام میشود که به ترجمهٔ کاراتری منجر میشود.
ازهمپاشیدگی[ویرایش]
آرانایهای پیامرسان متفاوت درون یک سلول، دورهٔ فعالیت و پایداری متفاوتی دارند. در سلولهای باکتریایی، هر یک از آرانایهای پیامرسان از چند ثانیه تا بیش از یک ساعت میتوانند فعالیت نمایند. در سلولهای پستانداران، دورهٔ فعالیت آرانایهای پیامرسان از چند دقیقه تا چند روز متغیر است. پایداری بیشتر آرانایهای پیامرسان سبب تولید پروتئینهای بیشتر از آن آراِناِی پیامرسان میشود. دورهٔ زندگی محدود آرانایهای پیامرسان، یک سلول را قادر میکند تا بهسرعت در پاسخ به نیازمندیهای در حال تغییر، سنتز پروتئین را تغییر دهد. مکانیسمهای زیادی وجود دارند که به نابود نمودن یک آرانایهای پیامرسان منجر میشود.
ازهمپاشیدگی آرانایهای پیامرسان پروکاریوتی[ویرایش]
بهطور کلی دورهٔ فعالیت آرانایهای پیامرسان پروکاریوتی کوتاهتر از یوکاریوتها است. پروکاریوتها پیامها را با استفاده از ترکیبی از ریبونوکلئازها که شامل اندونوکلئازها، اگزونوکلئازهای ۳' و ۵' هستند، از بین میبرند. در بعضی موارد، مولکولهای کوچک آرانایهای پیامرسان (sRNAها) با طول دهها تا صدها نوکلئوتید، توسط جفت شدن بازها با رشتههای مکمل میتوانند موجب ازهمپاشیدگی آرانایهای پیامرسان خاصی شده و شکسته شدن آنها توسط ریبونوکلئازها را فراهم نمایند. اخیراً نشان داده شدهاست که باکتریها نیز گونهای از کلاهک ۵' را دارند که شامل یک تریفسفات روی انتهای ۵' است.[۱۰] حذف دو فسفات، یک ۵َ مونوفسفات بر جا میگذارد که سبب میشود تا اگزونوکلئاز «ریبونوکلئاز جِی» پیام را نابود کند.
تجدید آرانایهای پیامرسان یوکاریوتی[ویرایش]
درون سلولهای یوکاریوتی، تعادلی بین فرایندهای ترجمه و حذف آرانایهای پیامرسان وجود دارد. پیامهایی که در حال ترجمه شدن هستند توسط ریبوزومها، فاکتورهای آغازین یوکاریوتی: eIF-4E و eIF-4G و پروتئین متصل به پلی (آ) گرفتار میشوند. eIF-4E و eIF-4G آنزیم کلاهکبرداری DCP2 را مهار میکنند و پروتئین پلی (آ)-بایندینگ، کمپلکس اگزوزوم را که انتهای پیام را محافظت میکند، مهار میکند. تعادل بین ترجمه و فروپاشی در اندازه و فراوانی ساختارهای سیتوپلاسمی با عنوان اجسام پی (P-bodies)[۱۱] نمود پیدا میکند. دنبالهٔ پلی (آ) از آرانایهای پیامرسان توسط اگزونوکلئاز خاصی که توسط ترکیبی از رشتههای تنظیمی سیس بر روی آراِناِی و پروتئینهای ترانس مقید شده به آرانای (trans-acting RNA-binding proteins) که به آرانایهای پیامرسان خاصی هدفگیری میشوند، کوتاه میشود. حذف دنبالهٔ پلی (آ) در نظر گرفته میشود که ساختار حلقوی پیام را از بین میبرد و کمپلکس اتصال به کلاهک (Cap binding complex) را بیثبات مینماید. پیام سپس با کمپلکس اگزوزوم یا کمپلکس کلاهکبردار به ازهمپاشیدگی مربوط میشود. با این روش، پیامهای ترجمهای غیرفعال میتوانند بهسرعت نابود شوند، درحالی که پیامهای فعال، سالم میمانند. مکانیسمی که با آن ترجمه توقف مییابد و پیامی که کمپلکسها را از بین میبرد بهطور کامل شناخته نشدهاست.
از بین رفتن عنصر غنی از آدنیلات-اوریدیلات[ویرایش]
حضور عناصر غنی از آدنیلات-اوریدیلات (AU-rich) در تعدادی از آرانایهای پیامرسان پستانداران سبب میشود تا رونوشتهایی که در خلال فعالیت پروتئینهای سلولی متصل به این رشتهها است بیثبات شوند و حذف نمودن دنبالهٔ پلی (آ) را سبب شود. نبود دنبالهٔ پلی (آ) سبب پیشرفت ازهمپاشیدگی آرانایهای پیامرسان توسط حمله کردن کمپلکس اگزوزوم و کمپلکس کلاهکبرداری میشود.[۱۲][۱۳] ازهمپاشیدگی سریع توسط عناصر AU-rich، یک مکانیسم حیاتی برای جلوگیری از تولید اضافی سیتوکینهای قوی همچون فاکتور فساد تومور (Tumor necrosis factor(TNF)) و فاکتور تحریک گرانولوسیت ماکروفاژ کولونی (granulocyte-macrophage colony stimulating factor (GM-CSF)) است.[۱۴] عناصر AU-rich همچنین بیوسنتز فاکتورهای رونویسی پروتو-اُنکوژنیک (proto-oncogenic transcription factors) همچون c-Jun و c-Fos را تنظیم مینمایند.[۱۵]
فروپاشی با واسطهٔ بیمعنی[ویرایش]
پیامهای یوکاریوتی توسط فروپاشی واسطهٔ بیمعنی (NMD) نظارت میشوند که برای وقوع کدونهای خاتمهٔ نابالغ (کدونهای بیمعنی) در پیام بررسی میشوند. اینها میتوانند از طریق پیرایش کاملنشده به وجود آیند. یافتن کدون خاتمه نابالغ سبب ازهمپاشیدگی آرانایهای پیامرسان بهوسیلهٔ کلاهکبرداری ۵'، حذف دنبالهٔ پلی (آ) ۳' یا بُرش اندونوکئازی (Endonucleolytic cleavage) میشود.[۱۶]
آرانایهای کوچک مداخلهکننده[ویرایش]
در جانوران، آرانایهای کوچک مداخلهگر (siRNAها) که توسط دایسر (Dicer) پردازش شدهاند، در کمپلکسی با عنوان کمپلکس خاموشی ناشی از آرانای (RNA-induced silencing complex (RISC)) جاگرفتهاند. این کمپلکس دارای یک اندونوکلئاز است که بهطور کامل، پیامهای مکمل را که siRNAها به آنها متصل میشوند را میشکافد. سپس بخشهای آراِناِی پیامرسان حاصل، بهوسیلهٔ اگزونوکلئاز نابود میشوند. siRNAها بهطور معمول در آزمایشگاهها برای مسدود کردن عملکرد ژنها در کشت سلولها بهکار میروند و همچنین بهعنوان بخشی از دستگاه ایمنی ذاتی بهعنوان یک مکانیسم دفاعی در برابر آرانای ویروسهای دورشتهای در نظر گرفته میشوند.[۱۷]
ریزآرانای[ویرایش]
ریزآرانایها آرانایهای کوچکی هستند که بهطور جزئی مکمل برخی رشتههای آرانایهای پیامرسان در جانوران هستند.[۱۸] اتصال یک ریزآرانای به یک پیام میتواند ترجمهٔ آن پیام را از بین ببرد و حذف دنبالهٔ پلی (آ) را سرعت بخشد و بدین وسیله ازهمپاشیدگی آرانایهای پیامرسان را سرعت ببخشد. سازوکار فعالیت ریزآرانایها اکنون موضوع تحقیقات بسیاری است.[۱۹]
سایر مکانیسمهای فروپاشی[ویرایش]
راههای دیگری نیز برای از بین رفتن پیامها وجود دارد که شامل فروپاشی بدون توقف (Non-stop decay)، خاموشی توسط آرانای تعاملکننده با پیوی (piRNA) و فرایندهایی دیگر هستند.
منابع[ویرایش]
- ↑ ۱٫۰ ۱٫۱ ۱٫۲ زیستشناسی (۳)، پایهٔ دوازدهم دورهٔ دوم متوسطه - ۱۱۲۲۱۶.
- ↑ D, Kierzkowski; M, Kmieciak; P, Piontek; P, Wojtaszek; Z, Szweykowska-Kulinska; A, Jarmolowski (Fall 2009). "The Arabidopsis CBP20 targets the cap-binding complex to the nucleus, and is stabilized by CBP80". The Plant journal: for cell and molecular biology (به انگلیسی). 59 (5). doi:10.1111/j.1365-313X.2009.03915.x. ISSN 1365-313X. PMID 19453442.
- ↑ Job, C.; Eberwine, J. (1912), "Localization and translation of mRNA in dendrites and axons" (w), Nat Rev Neurosci, 2001 (12): 889–98, doi:10.1038/35104069, PMID 11733796
- ↑ Ainger, Kevin; Avossa, Daniela; Diana, Amy S.; Barry, Christopher; Barbarese, Elisa; Carson, John H. (1997), "Transport and Localization Elements in Myelin Basic Protein mRNA", The Journal of Cell Biology, 138 (5): 1077–1087, doi:10.1083/jcb.138.5.1077, PMC 2136761, PMID 9281585
- ↑ Shabalina SA, Ogurtsov AY, Spiridonov NA (2006), "A periodic pattern of mRNA secondary structure created by the genetic code", Nucleic Acids Res., 34 (8): 2428–37, doi:10.1093/nar/gkl287, PMC 1458515, PMID 16682450
{{citation}}: نگهداری یادکرد:نامهای متعدد:فهرست نویسندگان (link) - ↑ Katz, Luba; Burge, Christopher B. (Summer 2003). "Widespread selection for local RNA secondary structure in coding regions of bacterial genes". Genome Research. 13 (9): 2042–2051. doi:10.1101/gr.1257503. ISSN 1088-9051. PMID 12952875.
- ↑ Kozak, M. (Winter 1983). "Comparison of initiation of protein synthesis in procaryotes, eucaryotes, and organelles". Microbiological Reviews. 47 (1): 1–45. doi:10.1128/mr.47.1.1-45.1983. ISSN 0146-0749. PMID 6343825.
- ↑ Niehrs, C.; Pollet, N. (1999-12-02). "Synexpression groups in eukaryotes". Nature. 402 (6761): 483–487. doi:10.1038/990025. ISSN 0028-0836. PMID 10591207.
- ↑ Wells, S. E.; Hillner, P. E.; Vale, R. D.; Sachs, A. B. (Summer 1998). "Circularization of mRNA by eukaryotic translation initiation factors". Molecular Cell. 2 (1): 135–140. doi:10.1016/s1097-2765(00)80122-7. ISSN 1097-2765. PMID 9702200.
- ↑ Deana, Atilio; Celesnik, Helena; Belasco, Joel G. (2008), "The bacterial enzyme RppH triggers messenger RNA degradation by 5' pyrophosphate removal", Nature, 451 (7176): 355, doi:10.1038/nature06475, PMID 18202662
- ↑ Parker, R.; Sheth, U. (2007), "P Bodies and the Control of mRNA Translation and Degradation" (w), Molecular Cell, 25 (5): 635–646, doi:10.1016/j.molcel.2007.02.011, PMID 17349952
- ↑ Chen, C.Y.; Gherzi, R.; Ong, S.E.; Chan, E.L.; Raijmakers, R.; Pruijn, G.J.M.; Stoecklin, G.; Moroni, C.; Mann, M. (2001), "AU Binding Proteins Recruit the Exosome to Degrade ARE-Containing mRNAs", Cell, 107 (4): 451–464, doi:10.1016/S0092-8674(01)00578-5, PMID 11719186, archived from the original on 17 September 2011, retrieved 28 June 2011
- ↑ Fenger-Grøn, Martin; Fillman, Christy; Norrild, Bodil; Lykke-Andersen, Jens (2005-12-22). "Multiple processing body factors and the ARE binding protein TTP activate mRNA decapping". Molecular Cell. 20 (6): 905–915. doi:10.1016/j.molcel.2005.10.031. ISSN 1097-2765. PMID 16364915.
- ↑ Shaw, G.; Kamen, R. (1986-08-29). "A conserved AU sequence from the 3' untranslated region of GM-CSF mRNA mediates selective mRNA degradation". Cell. 46 (5): 659–667. doi:10.1016/0092-8674(86)90341-7. ISSN 0092-8674. PMID 3488815.
- ↑ Chen, C.Y.A.; Shyu, A.B. (1995), "AU-rich elements: characterization and importance in mRNA degradation", Trends in Biochemical Sciences, 20 (11): 465–470, doi:10.1016/S0968-0004(00)89102-1, PMID 8578590
- ↑ Isken, O.; Maquat, L.E. (2007), "Quality control of eukaryotic mRNA: safeguarding cells from abnormal mRNA function", Genes & Development, 21 (15): 1833, doi:10.1101/gad.1566807, PMID 17671086
- ↑ Obbard, D.J.; Gordon, K.H.J.; Buck, A.H.; Jiggins, F.M. (2009), "Review. The evolution of RNAi as a defence against viruses and transposable elements", Philosophical Transactions of the Royal Society B: Biological Sciences, 364 (1513): 99, doi:10.1098/rstb.2008.0168, PMC 2592633, PMID 18926973
- ↑ Brennecke, Julius; Stark, Alexander; Russell, Robert B; Cohen, Stephen M (Winter 2005). "Principles of MicroRNA–Target Recognition". PLoS Biology. 3 (3): e85. doi:10.1371/journal.pbio.0030085. ISSN 1544-9173. PMC 1043860. PMID 15723116.
- ↑ Eulalio, Ana; Huntzinger, Eric; Nishihara, Tadashi; Rehwinkel, Jan; Fauser, Maria; Izaurralde, Elisa (Winter 2009). "Deadenylation is a widespread effect of miRNA regulation". RNA (New York, N.Y.). 15 (1): 21–32. doi:10.1261/rna.1399509. ISSN 1469-9001. PMC 2612776. PMID 19029310.
