پلیآدنیله شدن

پلیآدنیله شدن (به انگلیسی: Polyadenylation)، افزودن دُم پُلی(A) به آرانای پیامرسان (mRNA) است. دم پلی(A) شامل چندین آدنوزین مونوفسفات است؛ به بیان دیگر، یک توالی از آرانای است که تنها شامل بازهای آدنین میباشد. در یوکاریوتها، پلیآدنیله شدن، بخشی از فرایند تولید آرانای پیامرسان بالغ جهت ترجمه است. در بسیاری از باکتریها، دم پلی(A)، تجزیهٔ آرانای پیامرسان را ترغیب میکند؛ بنابراین بخشی از فرایند بیان ژن را تشکیل میدهد.
فرایند پلیآدنیله شدن، پس از پایان رونویسی یک ژن آغاز میگردد. قطعهای از پیش-mRNA تازه ساخته شده که در سمت '۳ قرار دارد، ابتدا توسط مجموعهای از پروتئینها جدا شده؛ سپس این پروتئینها دم پلی(A) را در انتهای '۳ سنتز میکنند. در برخی از ژنها، این پروتئینها دم پلی(A) جدیدی را در یکی از چندین مکان ممکن میافزایند؛ بنابراین، پلیآدنیله شدن، توانایی تولید بیش از یک رونوشت را از یک ژن واحد داراست (پلیآدنیله شدن جایگزین)، که مشابه پیرایش جایگزین است.[۱]
دم پلی(A) در خروج از هسته، ترجمه و پایداری آرانای پیامرسان اهمیت دارد. دم طی زمان کوتاه شده، و هنگامی که به میزان کافی کوتاه شود، آرانای پیامرسان با استفاده از آنزیمها تجزیه میگردد.[۲] با این حال، در برخی از انواع سلولها، آرانایهای پیامرسانی با دمهای کوتاه پلی(A) برای فعالسازیهای بعدی به کمک باز-پلیآدنیله شدن در سیتوزول، ذخیره میگردند.[۳] در مقایسه، هنگامی که در باکتریها پلیآدنیله شدگی رخ دهد، تجزیهٔ آرانای را ترغیب میکند.[۴] چنین اتفاقی برخی مواقع در آرانایهای غیر-کدکننده یوکاریوتی نیز رخ میدهد.[۵][۶]
مولکولهای آرانای پیامرسان در هردو دستهٔ پروکاریوتها و یوکاریوتها، دارای انتهای '۳ پلیآدنیلهاند، به گونهای که دمهای پلی(A) پروکاریوتی عمدتاً کوتاهتر بوده و مولکولهای آرانای پیامرسان پلیآدنیلهشدهٔ کمتری دارند.[۷]
مروری بر آرانای[ویرایش]
آرانایها نوعی از مولکولهای زیستی بزرگی هستند که از بلوکهای منحصر به فردی بهنام نوکلئوتید (آدنین، سیتوزین، گوانین و یوراسیل) ساخته شدهاند. آرانای از روی الگوی دیانای ساخته میشود. طبق قرارداد، دنبالهٔ آرانای از انتهای ۵'به سمت انتهای ۳' نوشته میشود؛ بنابراین، انتهای ۵' زودتر از انتهای ۳' رونویسی میشود. همچنین، انتهای ۳' محلی است که دنبالهٔ آدنین برای چندآدنینهشدن قرار میگیرد.[۸]
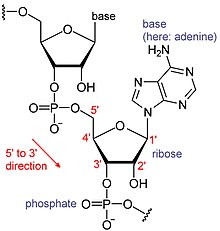
آرانای پیامرسان، نوعی آرانای دارای نواحی رمزشدهای است که برای تولید پروتئین (ترجمه)استفاده میشوند. دیگر قسمتهای آرانای پیامرسان، نواحی ترجمهنشدهای هستند که میزان فعال بودن آرانای پیامرسان را تنظیم میکنند.[۹] تعداد زیادی آرانای وجود دارد که ترجمه نشدهاند و آرانای بیرمز نام دارند. مشابه نواحی ترجمهنشده، بیشتر آرانایهای بیرمز، نقش تنظیمکننده دارند.[۱۰]
چندآدنینهشدن هستهای[ویرایش]
وظیفه[ویرایش]
در پلیآدنیلهشدن هستهای، دنبالهٔ آدنین به آرانای در انتهای رونویسی اضافه میشود. در آرانای پیامرسان، دنبالهٔ آدنین مولکول در آرانای پیامرسان را از تجزیهٔ آنزیمی در سیتوپلاسم سلول محافظت میکند و در پایان رونویسی، به خروج آرانای پیامرسان از هسته و ترجمهٔ آن کمک میکند. تقریباً تمام آرانایهای پیامرسان یوکاریوتی چندآدنینه میشوند.[۱۱] به استثنای هیستون آرانایهای پیامرسان وابسته به تکثیر حیوانات.[۱۲] این آرانایهای پیامرسان تنها آرانایهای پیامرسانی در یوکاریوتها هستند که دنبالهٔ آدنین ندارند و به جای آن در یک ساختار ساقه-حلقه به دنبال یک توالی غنی از پورین که هیستون عنصر پاییندست نامیده میشوند و مشخص میکنند چه زمانی آرانای قطع شدهاست و در نتیجه انتهای ۳' هیستون آرانای پیامرسان شکل گرفتهاست، پایان مییابند.[۱۳] بیشتر آرانایهای بیرمز یوکاریوتی در انتهای رونویسی چندآدنینه میشوند. آرانایهای کوچکی وجود دارند، مانند ریزآرانای، که دنبالهٔ آدنین فقط در شکلهای میانی آنها دیده میشوند نه در شکل کامل آن.[۱۴][۱۵] اما برای بیشتر آرانایهای بیرمز طولانی، دنبالهٔ آدنین بخشی از آرانای کامل است.[۱۶]
مکانیزم[ویرایش]
سیستم پلیآدنیلهسازی در هستهٔ یوکاریوتها، روی محصولات آرانای پلیمراز II کار میکند، مانند آرانایهای پیامرسان پیشرو. در این، جا یک مجموعه شامل چند پروتئین، بخش ۳' از آرانای جدید تولیدشده را میشکافند و انتهای ناحیهٔ شکافتهشده را پلیآدنیله میکنند. این شکاف کاتالیزشده توسط آنزیم CPSF و در ۱۰–۳۰ نوکلئوتید پایینتر از محل اتصال، رخ میدهد.[۱۷] در این ناحیه روی آرانای اغلب دنبالهٔ AAUAAA وجود دارد، اما انواع مختلف آن که بهصورت ضعیف به CPSF وصل شدهاند نیز وجود دارد.[۱۸] دو پروتئین دیگر نیز بهطور اختصاصی به آرانای وصل میشوند: CstF و CFI.[۱۹]CstF ککک به ناحیهٔ غنی از GU و پایینتر از ناحیهٔ CPSF وصل میشود. CFI ناحیهٔ سوم آرانای (مجموعهای از UGUAAها در پستانداران[۲۰][۲۱][۲۲]) را به رسمیت میشناسد و حتی اگر دنبالهٔ AAUAAA وجود نداشته باشد بازهم میتواند CPSF را بهکار گیرد.[۲۳][۲۴] سیگنال پلیآدنیلهشدن –دنبالهای از الگوهای شناخته شده توسط مجموعه شکاف دهنده آرانای- بین گروههای یوکاریوتی متفاوت است. بیشتر ناحیههای پلیآدنیله شدن انسانها شامل AAUAAA است اما این دنباله در بین گیاهان و قارچها کمتر رایج است.[۲۵] آرانای معمولاً قبل از پایان رونویسی شکافته میشود و CstF به آرانای پلیمرازΙΙمتصل میشود.[۲۶] پروتئین CFI همچنین در عملیات شکافتن نقش دارد (اگر چگونگی آن هنوز ناشناخته است).[۲۷] ناحیهٔ شکاف میتواند تا حدود ۵۰ نوکلئوتید متفاوت باشد.[۲۸] وقتی آرانای شکافته شد، فرایند چندآدنینی شروع شده و توسط پلیمراز چندآدنینی کاتالیز میشود. پلیمراز چندآدنینی، دنبالهٔ آدنین را با اضافه کردن ادنوزین منو فسفات از ادنوزین تری فسفات (با شکافتن پیروفسفات) میسازد. پروتئین دیگر، PAB2،[۲۹] به دنبالهٔ آدنین کوتاه جدید متصل میشود و میل پلیمراز چندآدنینی برای اضافه کردن طول دنبالهٔ آدنین به آرانای را افزایش میدهد.[۳۰][۳۱] وقتی طول دنبالهٔ آدنین به حدود ۲۵۰ نوکلئوتید رسید، آنزیم دیگر نمیتواند CPSF را متصل نگه دارد و فرایند پلیآدنیلهشدن متوقف میشود و به این ترتیب طول دنبالهٔ آدنین مشخص میشود. CPSF با آرانای پلیمرازΙΙ در ارتباط است و اجازه ارسال سیگنال پایان رونویسی به پلیمراز را میدهد.[۳۲][۳۳] مکانیزم چندآدنینیشدن همچنین بهطور فیزیکی با پیرایش که مجموعهای که اینترونها را از آرانای حذف میکنند، ارتباط دارد.
اثرهای پاییندست[ویرایش]
دنبالهٔ آدنین بهعنوان ناحیهٔ ضروری برای پروتئینهای ضروری آن عمل میکند. این پروتئینها اجازه خروج آرانای پیامرسان از هسته سلول و ترجمه آن را میدهند و از تجزیه آن جلوگیری میکنند.[۳۴] این پروتئینها قبل از خروج آرانای پیامرسان از هسته به آن متصل میشوند و در مخمرها، همچنین هستهٔ دنباله به کار گرفته میشود تا با استفاده از آن دنبالهٔ آدنین کوتاه شده و اجازه خروج آرانای پیامرسان داده شود. این پروتئینها همراه با آرانای به سیتوپلاسم صادر میشوند و آرانای پیامرسانی که صادر نشده باشد توسط اگزوزوم تجزیه میشود.[۳۵][۳۶] همچنین، این پروتئینها میتوانند به پروتئینهای دیگری که روی ترجمه اثر میگذارند متصل شوند و از آنها استفاده کنند. یکی از این پروتئینها فاکتور شروع-4G است که به نوبه خود باعث شروع به کار واحد کوچک ریبوزوم(40S) میشود.[۳۷] با اینحال، دنبالهٔ آدنین برای ترجمهٔ همهٔ آرانای پیامرسانها ضروری نیست.[۳۸]
حذف گروهی از پروتئینها(deadenylation)[ویرایش]
در سلولهای سوماتیک یوکاریوتی، طول دنبالهٔ آدنینی در سیتوپلاسم به تدریج کوتاهتر میشود، و آرانایهای پیامرسانی با دنبالهٔ کوتاه آدنینی کمتر ترجمه شده و در نتیجه زودتر تجزیه میشوند.[۳۹] با این حال، این تجزیه چندین ساعت طول خواهد کشید.[۴۰] این فرایند حذف و تجزیه میتواند توسط میکروآرانایهای مکمل ناحیهٔ ترجمه نشده ۳’ تسریع شود.[۴۱] در سلولهای تخم نابالغ، آرانایهای پیامرسانی که طول دنباله آدننین کوتاه دارند تجزیه نمیشوند، بلکه بدون ترجمه شدن ذخیره میشوند. سپس در دوران فعالیت تخم و بعد از لقاح توسط پلیآدنیلهشدن سیتوپلاسمی فعال میشوند.[۴۲] در حیوانات، پلی آدنین ریبونوکلئاز (PARN) میتواند به کلاه ۵’ متصل شده و نوکلئوتیدهای دنبالهٔ آدنین را حذف کند. سطح دسترسی به کلاه ۵’ و دنبالهٔ آدنین در کنترل اینکه چه مدت دیگر آرانای پیامرسانی باید تجزیه شود تأثیر دارد. PARN پروتئین کمتری را حذف میکند اگر آرانای توسط فاکتورهای شروع 4E در کلاه ۵’ و 4Gدر دنباله آدنین محدود شده باشد که در نتیجه عمل ترجمه باعث کاهش حذف پروتئین میشود. نرخ حذف شدن ممکن است توسط پروتئینهای ضروری آرانای تنظیم شود. وقتی که دنبالهٔ آدنین حذف شد، مجموعه پروتئینهای حذفکنندهٔ کلاه، کلاه ۵’ را حذف میکنند و در نتیجه آرانای تجزیه میشود. چندین آنزیم دیگر که در حذف پروتئینها از مخمها نقش دارند نیز شناخته شدهاند.[۴۳]
چندآدنینهشدن جایگزین[ویرایش]
بیشتر ژنهای کدکننده پروتئین بیشتر از یک محل برای چندآدنینه شدن دارند و در نتیجه برای یک ژن چندین نسخه متفاوت آرانایهای پیامرسانی که در انتهای ۳’شان متفاوتند وجود دارد.[۴۴][۴۵] چون چندآدنینه شدن جایگزین طول ناحیه ترجمه نشده ۳’ را تغییر میدهد، میتواند باعث تغییر مکانهایی که برای اتصال میکرو آرانای در ناحیههای ترجمه نشده ۳’ است، شوند.[۴۶] میکرو آرانای تمایل به سرکوب ترجمه و تجزیه آرانایهای پیامرسانی که به آنها متصلاند را دارند، اگر چه مثالهایی از میکرو RNAهایی که رونوشت پایداری دارند نیز وجود دارد.[۴۷][۴۸] چندآدنینه شدن جایگزین میتواند باعث کوتاه شدن ناحیه رمز نگاری شده شود که باعث میشود ایجاد کد آرانای پیامرسان برای یک پروتئین متفاوت شود.[۴۹][۵۰] اما این اتفاق نسبت به اینکه فقط ناحیه ترجمه نشده ۳’ کوتاه شود، کمتر رایج است.

انتخاب ناحیه پلیآدنین میتواند توسط محرکهای خارج سلولی و وابسته به بیان پروتئینهایی که در چندآدنینه شدن نقش دارند، تأثیر پذیرد.[۵۱][۵۲] برای مثال، بیان CstF-64، یک زیرواحد از فاکتور تحریککننده شکاف (CstF)، در ماکروفاژهای پاسخ به لیپوپلیساکارید (یک گروه از ترکیبات باکتریایی که باعث یک پاسخ ایمنی میشوند) افزایش مییابد. این اتفاق نتیجهٔ انتخاب محلهای پلی ادنین ضعیف است و در نتیجه رونوشت کوتاهتر میشود. این عناصر تنظیمکننده در ناحیه ترجمه نشده۳’ از آرانای پیامرسان برای محصولات مرتبط با دفاع مثل لیزوزم و عامل نکروز توموری آلفا را حذف میکند. این آرانایهای پیامرسان نیمه-عمر طولانیتری دارند و تعداد بیشتری از این پروتئینها را تولید میکنند. پروتئینهای ضروری آرانای نسبت به دیگر پروتئینهایی که در مکانیزم چندآدنینه شدن نقش دارند میتوانند این که آیا یک سایت برای چندآدنینه شدن استفاده شود یا نه[۵۲][۵۳][۵۴][۵۵] و همچنین دیانای متیلاسیون نزدیک نواحی چندآدنینه شدن را تحت تأثیر قرار دهند.[۵۶]
چندآدنینه شدن سیتوپلاسمی[ویرایش]
چندآدنینه شدن در سیتوزول برخی از انواع سلولهای حیوانی وجود دارد، یعنی در راستای نطفه، در رویانزایی و در محلهای پسا-سیناپسی در یاخته عصبی. این پلیآدنیله شدن سیتوزولی سبب افزایش طول دنباله آدنین در آرانایهای پیامرسان با دنباله ادنین کوتاه میشود بنابراین این آرانایهای پیامرسان ترجمه خواهند شد.[۵۷] طولهای کوتاه حدوداً ۲۰ نوکلئوتید دارند و به حدود ۸۰–۱۵۰ نوکلئوتید افزایش پیدا خواهند کرد. در جنین ابتدایی موش، چندآدنینه شدن سیتوزولی در آرانایهای مادر سلول تخم، اجازه میدهند سلول زنده مانده و رشد کند حتی اگر رونویسی تا میانههای سطح ۲-سلول (سطح ۴سلول در انسان) آغاز نشده باشد.[۵۸][۵۹] درمغز، چندآدنینه شدن سیتوزولی در طول یادگیری فعال شده و میتواند نقش مهمی را در تقویت انتقال سیگنال از یک سلول عصبی به سلول دیگر و برای یادگیری و شکلگیری حافظه داشته باشد.[۶۰] چندآدنینه شدن سیتوپلاسمی به پروتئینهای CPSF و CPEB ویا حتی پروتئینهای دیگری مثل پومیلیو نیاز دارد.[۶۱] بسته به نوع سلول، پلیمراز میتواند همنوع پلیمراز چندآدنینی (PAP)که در فرایند هسته استفاده میشود یا پلیمراز سیتوپلاسمی GLD-2 باشد.[۶۲]
برچسب تجزیه در یوکاریوتها[ویرایش]
برای بسیاری از آرانایهای بی رمز، مثل آرانای حامل، و آرانای ریبوزومی و آرانای کوچک هستهای، پلیآدنیله شدن یک راه برای علامتگذاری برای تجزیه شدن آرانای است، حداقل در مخمرها.[۶۳] این پلیآدنیله شدن در هسته و توسط ترکیب TRAMP انجام میشود، به این ترتیب که یک دنباله با طول حدود ۴۰ نوکلئوتید را به انتهای ۳’ اضافه میکند.[۶۴] سپس آرانای توسط اگزوزوم تجزیه میشود.[۶۵] دنبالهٔ آدنین همچنین در بخشهایی از آرانای ریبوزومی انسان در هر دو شکل هموپلیمر (فقط شامل آدنین) و هتروپلیمر (بیشتر شامل آدنین) یافته شدهاست.[۶۶]
در یوکاریوتها و اندامکها[ویرایش]
در بسیاری از باکتریها هم آرانایهای پیامرسان و هم آرانایهای بی رمز میتوانند چندآدنینه شوند. این دنبالهٔ آدنین، تجزیه توسط دیگرادوزوم که شامل دو آنزیم تجزیهکنندهٔ آرانای است (پلیمراز چندآدنینه و RNase E)را جلو میاندازد. پلیمراز چندآدنینه به انتهای ۳’ آرانای متصل شده و به این ترتیب پسوند۳’ تولید شده توسط دنبالهٔ آدنین اجازه پیدا میکند که به آرانای متصل شود و ساختار دوم برای آن ایجاد شده و انتهای ۳’ مسدود شود. دورهای بعدی چندآدنینه شدن و تجزیه انتهای ۳’ توسط پلیمراز چندآدنینه اجازه میدهند که دگرادوزوم بر این ساختارهای دوم غلبه کند. دنبالهٔ آدنین همچنین میتواند با بهکارگیری RNases آرانای را به دو قسمت تقسیم کند.[۶۷] این دنبالههای آدنینی باکتریایی حدود ۳۰ نوکلئوتید طول دارند.[۶۸]

در حیوانات و متهتنسانان، میتوکندری شامل هر دو حالت دنبالهٔ آدنین پایدار و ناپایدار است. چندآدنینه شدن ناپایدار آرانایهای پیامرسان و آرانایهای بی رمز را هدف قرار میدهد. دنبالهٔ آدنین بهطور متوسط ۴۳ نوکلئوتید طول دارد که در حالت پایدار از رمز پایان شروع میشوند و بدون آنها رمز پایان (UAA) مانند ژنومی که فقط ناحیه U یا UA را کد میکند، کامل نیست. میتوکندری گیاهان فقط چند آدنینیه ناپایدار دارند و میتوکندری مخمرها بهطور کلی چندآدنینه شدن ندارد.[۶۹] بیشتر باکتریها و میتوکندری علاوه بر پلیمراز چند، نوع دیگری از چندآدنینه شدن که توسط خود فسفریلاز پلی نکلئوتید اجرا میشود، را نیز دارند. این آنزیم در باکتری،[۷۰] میتوکندری[۷۱] و دیسه[۷۲] یافت شده و همچنین در اگزوزوم ارکیا هم وجود دارد (در آرکیهایی که اگزوزوم دارند).[۷۳] این آنزیم میتواند پسوند ۳’ در محلی که بیشتر آدنین وجود دارد را سنتز کند. مانند باکتری و آرکیها، پلیآدنیله شدن توسط فسفوریلاز پلی نوکلئوتید تجزیه آرانای را در دیسه[۷۴]ها جلو میاندازد.
سیر تکاملی[ویرایش]
اگرچه چندآدنینه شدن در تقریباً تمام موجودات دیده میشود، اما حقیقتی فراگیر نیست.[۷۵][۷۶] با این حال، توزیع گسترده از این تغییر و این که در حال حاضر در تمام سه دامنه موجودات زنده وجود دارد نشان دهنده این است که در جد مشترک فرض شده برای تمامی این موجودات نیز سیستم چندآدنینه شدن وجود داشتهاست. و اینکه در تعداد کمی از موجودات چندآدنینه شدن آرانای پیامرسان ندارند، نشان دهنده این است که در روند تکاملی این موجودات، این سیستم از بین رفتهاست. اگرچه هیچ مثالی از موجودات یوکاریوتی که چندآدنینه شدن نداشته باشند وجود ندارد، اما آرانایهای پیامرسان باکتری مایکوپلاسما گالی سپتیکوم و آرکیهای بدون قابلیت تحمل نمک هالوفراکس ولکانی این نقص را دارند.[۷۷][۷۸] قدیمیترین آنزیم چندآدنینی، فسفوریلاز پلی نکلئوتید است.[۷۹] این آنزیم بخشی از هر عامل تجزیه باکتری و اگزوزوم ارکیا است، دو ترکیب نسبتاً نزدیک که آرانای را به نوکلئوتیدهای سازنده اش بازیافت میکند. این آنزیم ا آرانای را با حمله به پیوند بین نکلئوتیدهای انتهای ۳’ با فسفاتها و شکستن نکلئوتید فسفات، تجزیه میکند. این عملی بازگشتپذیر است و بنابراین این آنزیم میتواند با اضافه کردن نوکلئوتیدها، آرانای را گسترش دهد.[۸۰]
تاریخچه[ویرایش]
چندآدنینه شدن برای اولین بار در دهه ۱۹۶۰ به عنوان یک فعالیت آنزیمی در هسته سلول شناخته شد که میتوانست ATP را به پلیآدنین تبدیل کند.[۸۱][۸۲] اگرچه در بسیاری از سلولها وجود این آنزیم مشخص شد اما تا سال ۱۹۷۱ که دنباله پلی آدنین در آرانایهای پیامرسان شناخته شد، هیچ وظیفهای برای این فعالیت تشخیص داده نشد.[۸۳][۸۴] در ابتدا تصور میشد تنها وظیفهٔ این دنبالهها محافظت انتهای ۳’ آرانای از نوکلئوتید هاست اما بعداً نقش چندآدنینه شدن در خروج آرانای پیامرسان از هسته و ترجمهٔ آن شناخته شد. پلیمراز چندآدنینه شدن اولین بار در دههٔ ۱۹۶۰ و ۱۹۷۰ شناسایی شد اما پروتئینهای جانبی آن که این فرایند را کنترل میکنند در ابتدای دههٔ ۱۹۹۰ کشف شد.
منابع[ویرایش]
- ↑ Proudfoot NJ, Furger A, Dye MJ (February 2002). "Integrating mRNA processing with transcription". Cell. 108 (4): 501–12. doi:10.1016/S0092-8674(02)00617-7. PMID 11909521. S2CID 478260.
- ↑ Guhaniyogi J, Brewer G (March 2001). "Regulation of mRNA stability in mammalian cells". Gene. 265 (1–2): 11–23. doi:10.1016/S0378-1119(01)00350-X. PMC 3340483. PMID 11255003.
- ↑ خطای یادکرد: خطای یادکرد:برچسب
<ref> غیرمجاز؛ متنی برای یادکردهای با نامRichterوارد نشده است. (صفحهٔ راهنما را مطالعه کنید.). - ↑ Steege DA (August 2000). "Emerging features of mRNA decay in bacteria". RNA. 6 (8): 1079–90. doi:10.1017/S1355838200001023. PMC 1369983. PMID 10943888.
- ↑ Zhuang Y, Zhang H, Lin S (June 2013). "Polyadenylation of 18S rRNA in algae(1)". Journal of Phycology. 49 (3): 570–9. doi:10.1111/jpy.12068. PMID 27007045. S2CID 19863143.
- ↑ Anderson JT (August 2005). "RNA turnover: unexpected consequences of being tailed". Current Biology. 15 (16): R635-8. doi:10.1016/j.cub.2005.08.002. PMID 16111937. S2CID 19003617.
- ↑ Sarkar N (June 1997). "Polyadenylation of mRNA in prokaryotes". Annual Review of Biochemistry. 66 (1): 173–97. doi:10.1146/annurev.biochem.66.1.173. PMID 9242905.
- ↑ Lehninger, Albert L. ; Nelson, David L. ; Cox, Michael M. , eds. (1993). Principles of biochemistry (2nd ed.). New York: Worth. ISBN 978-0-87901-500-8
- ↑ Abaza, I. ; Gebauer, F. (2008). "Trading translation with RNA-binding proteins". RNA. 14 (3): 404–9. doi:10.1261/rna.848208. PMC 2248257Freely accessible. PMID 18212021
- ↑ Mattick, J. S. (2006). "Non-coding RNA". Human Molecular Genetics. 15 (90001): R17–29. doi:10.1093/hmg/ddl046. Hunt, Arthur G; Xu, Ruqiang; Addepalli, Balasubrahmanyam; Rao, Suryadevara; Forbes, Kevin P; Meeks, Lisa R; Xing, Denghui; Mo, Min; Zhao, Hongwei (2008). "Arabidopsis mRNA polyadenylation machinery: comprehensive
- ↑ Hunt, Arthur G; Xu, Ruqiang; Addepalli, Balasubrahmanyam; Rao, Suryadevara; Forbes, Kevin P; Meeks, Lisa R; Xing, Denghui; Mo, Min; Zhao, Hongwei (2008). "Arabidopsis mRNA polyadenylation machinery: comprehensive analysis of protein-protein interactions and gene expression profiling". BMC Genomics. 9: 220. doi:10.1186/1471-2164-9-220. PMC 2391170Freely accessible. PMID 18479511
- ↑ Davila Lopez, M. ; Samuelsson, T. (2007). "Early evolution of histone mRNA 3' end processing". RNA. 14 (1): 1–10. doi:10.1261/rna.782308. PMC 2151031Freely accessible. PMID 17998288
- ↑ Marzluff, William F. ; Gongidi, Preetam; Woods, Keith R. ; Jin, Jianping; Maltais, Lois J. (2002). "The Human and Mouse Replication-Dependent Histone Genes". Genomics. 80 (5): 487–98. doi:10.1016/S0888-7543(02)96850-3. PMID 12408966
- ↑ Saini, H. K. ; Griffiths-Jones, S. ; Enright, A. J. (2007). "Genomic analysis of human microRNA transcripts". Proceedings of the National Academy of Sciences. 104 (45): 17719–24. doi:10.1073/pnas.0703890104
- ↑ Yoshikawa, M. (2005). "A pathway for the biogenesis of trans-acting siRNAs in Arabidopsis". Genes & Development. 19 (18): 2164–75. doi:10.1101/gad.1352605. PMC 1221887Freely accessible. PMID 16131612
- ↑ Amaral, Paulo P. ; Mattick, John S. (2008). "Noncoding RNA in development". Mammalian Genome. 19 (7–8): 454–92. doi:10.1007/s00335-008-9136-7. PMID 18839252
- ↑ Liu, D. ; Brockman, J. M. ; Dass, B. ; Hutchins, L. N. ; Singh, P. ; McCarrey, J. R. ; MacDonald, C. C. ; Graber, J. H. (2006). "Systematic variation in mRNA 3'-processing signals during mouse spermatogenesis". Nucleic Acids Research. 35 (1): 234–46. doi:10.1093/nar/gkl919. PMC 1802579Freely accessible. PMID 17158511
- ↑ Lutz, Carol S. (2008). "Alternative Polyadenylation: A Twist on mRNA 3′ End Formation". ACS Chemical Biology. 3 (10): 609–17. doi:10.1021/cb800138w. PMID 18817380
- ↑ Beaudoing, E. ; Freier, S; Wyatt, JR; Claverie, JM; Gautheret, D (2000). "Patterns of Variant Polyadenylation Signal Usage in Human Genes". Genome Research. 10 (7): 1001–10. doi:10.1101/gr.10.7.1001. PMC 310884Freely accessible. PMID 10899149
- ↑ Brown, Kirk M; Gilmartin, Gregory M (2003). "A Mechanism for the Regulation of Pre-mRNA 3′ Processing by Human Cleavage Factor Im". Molecular Cell. 12 (6): 1467–76. doi:10.1016/S1097-2765(03)00453-2. PMID 14690600
- ↑ Yang, Q. ; Gilmartin, G. M. ; Doublie, S. (2010). "Structural basis of UGUA recognition by the Nudix protein CFIm25 and implications for a regulatory role in mRNA 3' processing". Proceedings of the National Academy of Sciences. 107 (22): 10062–7. doi:10.1073/pnas.1000848107
- ↑ Yang, Qin; Coseno, Molly; Gilmartin, Gregory M. ; Doublié, Sylvie (2011). "Crystal Structure of a Human Cleavage Factor CFIm25/CFIm68/RNA Complex Provides an Insight into Poly(A) Site Recognition and RNA Looping". Structure. 19 (3): 368–77. doi:10.1016/j.str.2010.12.021. PMC 3056899Freely accessible. PMID 21295486
- ↑ Venkataraman, K. (2005). "Analysis of a noncanonical poly(A) site reveals a tripartite mechanism for vertebrate poly(A) site recognition". Genes & Development. 19 (11): 1315–27. doi:10.1101/gad.1298605. PMC 1142555Freely accessible. PMID 15937220
- ↑ Millevoi, Stefania; Loulergue, Clarisse; Dettwiler, Sabine; Karaa, Sarah Zeïneb; Keller, Walter; Antoniou, Michael; Vagner, StéPhan (2006). "An interaction between U2AF 65 and CF Im links the splicing and 3′ end processing machineries". The EMBO Journal. 25 (20): 4854–64. doi:10.1038/sj.emboj.7601331. PMC 1618107Freely accessible. PMID 17024186
- ↑ Shen, Y. ; Ji, G. ; Haas, B. J. ; Wu, X. ; Zheng, J. ; Reese, G. J. ; Li, Q. Q. (2008). "Genome level analysis of rice mRNA 3'-end processing signals and alternative polyadenylation". Nucleic Acids Research. 36 (9): 3150–61. doi:10.1093/nar/gkn158. PMC 2396415Freely accessible. PMID 18411206
- ↑ Glover-Cutter, Kira; Kim, Soojin; Espinosa, Joaquin; Bentley, David L (2007). "RNA polymerase II pauses and associates with pre-mRNA processing factors at both ends of genes". Nature Structural & Molecular Biology. 15 (1): 71–8. doi:10.1038/nsmb1352
- ↑ Stumpf, G. ; Domdey, H. (1996). "Dependence of Yeast Pre-mRNA 3'-End Processing on CFT1: A Sequence Homolog of the Mammalian AAUAAA Binding Factor". Science. 274 (5292): 1517–20. doi:10.1126/science.274.5292.1517. PMID 8929410
- ↑ Iseli, C. ; Stevenson, B. J. ; De Souza, S. J. ; Samaia, H. B. ; Camargo, A. A. ; Buetow, K. H. ; Strausberg, R. L. ; Simpson, A. J.G. ; Bucher, P. (2002). "Long-Range Heterogeneity at the 3' Ends of Human mRNAs". Genome Research. 12 (7): 1068–74. doi:10.1101/gr.62002. PMC 186619Freely accessible. PMID 12097343
- ↑ Balbo, Paul B. ; Bohm, Andrew (2007). "Mechanism of Poly(A) Polymerase: Structure of the Enzyme-MgATP-RNA Ternary Complex and Kinetic Analysis". Structure. 15 (9): 1117–31. doi:10.1016/j.str.2007.07.010. PMC 2032019Freely accessible. PMID 17850751
- ↑ Viphakone, N. ; Voisinet-Hakil, F. ; Minvielle-Sebastia, L. (2008). "Molecular dissection of mRNA poly(A) tail length control in yeast". Nucleic Acids Research. 36 (7): 2418–33. doi:10.1093/nar/gkn080. PMC 2367721Freely accessible. PMID 18304944
- ↑ Wahle, Elmar (1995). "Poly(A) Tail Length Control Is Caused by Termination of Processive Synthesis". Journal of Biological Chemistry. 270 (6): 2800–8. doi:10.1074/jbc.270.6.2800 (inactive 2010-03-18). PMID 7852352.
- ↑ Dichtl, B. ; Blank, D; Sadowski, M; Hübner, W; Weiser, S; Keller, W (2002). "Yhh1p/Cft1p directly links poly(A) site recognition and RNA polymerase II transcription termination". The EMBO Journal. 21 (15): 4125–35. doi:10.1093/emboj/cdf390. PMC 126137Freely accessible. PMID 12145212
- ↑ Nag, Anita; Narsinh, Kazim; Martinson, Harold G (2007). "The poly(A)-dependent transcriptional pause is mediated by CPSF acting on the body of the polymerase". Nature Structural & Molecular Biology. 14 (7): 662–9. doi:10.1038/nsmb1253
- ↑ Coller, J. M. ; Gray, N. K. ; Wickens, M. P. (1998). "mRNA stabilization by poly(A) binding protein is independent of poly(A) and requires translation". Genes & Development. 12 (20): 3226–35. doi:10.1101/gad.12.20.3226
- ↑ Siddiqui, N. ; Mangus, D. A. ; Chang, T. -C. ; Palermino, J. -M. ; Shyu, A. -B. ; Gehring, K. (2007). "Poly(A) Nuclease Interacts with the C-terminal Domain of Polyadenylate-binding Protein Domain from Poly(A)-binding Protein". Journal of Biological Chemistry. 282 (34): 25067–75. doi:10.1074/jbc.M701256200. PMID 17595167
- ↑ Vinciguerra, Patrizia; Stutz, FrançOise (2004). "mRNA export: an assembly line from genes to nuclear pores". Current Opinion in Cell Biology. 16 (3): 285–92. doi:10.1016/j.ceb.2004.03.013. PMID 15145353
- ↑ Gray, N. K. ; Coller, JM; Dickson, KS; Wickens, M (2000). "Multiple portions of poly(A)-binding protein stimulate translation in vivo". The EMBO Journal. 19 (17): 4723–33. doi:10.1093/emboj/19.17.4723. PMC 302064Freely accessible. PMID 10970864
- ↑ Meaux, S. ; Van Hoof, A (2006). "Yeast transcripts cleaved by an internal ribozyme provide new insight into the role of the cap and poly(A) tail in translation and mRNA decay". RNA. 12 (7): 1323–37. doi:10.1261/rna.46306. PMC 1484436Freely accessible. PMID 16714281
- ↑ Meijer, H. A. ; Bushell, M. ; Hill, K. ; Gant, T. W. ; Willis, A. E. ; Jones, P. ; De Moor, C. H. (2007). "A novel method for poly(A) fractionation reveals a large population of mRNAs with a short poly(A) tail in mammalian cells". Nucleic Acids Research. 35 (19): e132–e132. doi:10.1093/nar/gkm830
- ↑ Lehner, B. ; Sanderson, CM (2004). "A Protein Interaction Framework for Human mRNA Degradation". Genome Research. 14 (7): 1315–23. doi:10.1101/gr.2122004. PMC 442147Freely accessible. PMID 15231747
- ↑ Wu, L. (2006). "From the Cover: MicroRNAs direct rapid deadenylation of mRNA". Proceedings of the National Academy of Sciences. 103 (11): 4034–9. doi:10.1073/pnas.0510928103
- ↑ Cui, J. ; Sackton, K. L. ; Horner, V. L. ; Kumar, K. E. ; Wolfner, M. F. (2008). "Wispy, the Drosophila Homolog of GLD-2, Is Required During Oogenesis and Egg Activation". Genetics. 178 (4): 2017–29. doi:10.1534/genetics.107.084558. PMC 2323793Freely accessible. PMID 18430932
- ↑ Wilusz, Carol J. ; Wormington, Michael; Peltz, Stuart W. (2001). "The cap-to-tail guide to mRNA turnover". Nature Reviews Molecular Cell Biology. 2 (4): 237–46. doi:10.1038/35067025. PMID 11283721
- ↑ Tian, B. ; Hu, J; Zhang, H; Lutz, CS (2005). "A large-scale analysis of mRNA polyadenylation of human and mouse genes". Nucleic Acids Research. 33 (1): 201–12. doi:10.1093/nar/gki158. PMC 546146Freely accessible. PMID 15647503
- ↑ Danckwardt, Sven; Hentze, Matthias W; Kulozik, Andreas E (2008). "3′ end mRNA processing: molecular mechanisms and implications for health and disease". The EMBO Journal. 27 (3): 482–98. doi:10.1038/sj.emboj.7601932. PMC 2241648Freely accessible. PMID 18256699
- ↑ Sandberg, R. ; Neilson, J. R. ; Sarma, A. ; Sharp, P. A. ; Burge, C. B. (2008). "Proliferating Cells Express mRNAs with Shortened 3' Untranslated Regions and Fewer MicroRNA Target Sites". Science. 320 (5883): 1643–7. doi:10.1126/science.1155390. PMC 2587246Freely accessible. PMID 18566288
- ↑ Tili, Esmerina; Michaille, Jean-Jacques; Calin, George Adrian (2008). "Expression and function of micro-RNAs in immune cells during normal or disease state". International Journal of Medical Sciences. 5 (2): 73–9. PMC 2288788Freely accessible. PMID 18392144
- ↑ Ghosh, T. ; Soni, K. ; Scaria, V. ; Halimani, M. ; Bhattacharjee, C. ; Pillai, B. (2008). "MicroRNA-mediated up-regulation of an alternatively polyadenylated variant of the mouse cytoplasmic -actin gene". Nucleic Acids Research. 36 (19): 6318–32. doi:10.1093/nar/gkn624. PMC 2577349Freely accessible. PMID 18835850
- ↑ Alt, F; Bothwell, AL; Knapp, M; Siden, E; Mather, E; Koshland, M; Baltimore, D (1980). "Synthesis of secreted and membrane-bound immunoglobulin mu heavy chains is directed by mRNAs that differ at their 3′ ends". Cell. 20 (2): 293–301. doi:10.1016/0092-8674(80)90615-7. PMID 6771018
- ↑ Tian, B. ; Pan, Z. ; Lee, J. Y. (2007). "Widespread mRNA polyadenylation events in introns indicate dynamic interplay between polyadenylation and splicing". Genome Research. 17 (2): 156–65. doi:10.1101/gr.5532707. PMC 1781347Freely accessible. PMID 17210931
- ↑ Shell, S. A. ; Hesse, C; Morris Jr, SM; Milcarek, C (2005). "Elevated Levels of the 64-kDa Cleavage Stimulatory Factor (CstF-64) in Lipopolysaccharide-stimulated Macrophages Influence Gene Expression and Induce Alternative Poly(A) Site Selection". Journal of Biological Chemistry. 280 (48): 39950–61. doi:10.1074/jbc.M508848200. PMID 16207706
- ↑ ۵۲٫۰ ۵۲٫۱ Danckwardt, Sven; Gantzert, Anne-Susan; Macher-Goeppinger, Stephan; Probst, Hans Christian; Gentzel, Marc; Wilm, Matthias; Gröne, Hermann-Josef; Schirmacher, Peter; Hentze, Matthias W. (2011). "p38 MAPK Controls Prothrombin Expression by Regulated RNA 3′ End Processing". Molecular Cell. 41 (3): 298–310. doi:10.1016/j.molcel.2010.12.032. PMID 21292162
- ↑ Licatalosi, Donny D. ; Mele, Aldo; Fak, John J. ; Ule, Jernej; Kayikci, Melis; Chi, Sung Wook; Clark, Tyson A. ; Schweitzer, Anthony C. ; Blume, John E. (2008). "HITS-CLIP yields genome-wide insights into brain alternative RNA processing". Nature. 456 (7221): 464–9. doi:10.1038/nature07488. PMC 2597294Freely accessible. PMID 18978773
- ↑ Hall-Pogar, T. ; Liang, S. ; Hague, L. K. ; Lutz, C. S. (2007). "Specific trans-acting proteins interact with auxiliary RNA polyadenylation elements in the COX-2 3'-UTR". RNA. 13 (7): 1103–15. doi:10.1261/rna.577707. PMC 1894925Freely accessible. PMID 17507659
- ↑ Danckwardt, Sven; Kaufmann, Isabelle; Gentzel, Marc; Foerstner, Konrad U; Gantzert, Anne-Susan; Gehring, Niels H; Neu-Yilik, Gabriele; Bork, Peer; Keller, Walter (2007). "Splicing factors stimulate polyadenylation via USEs at non-canonical 3′ end formation signals". The EMBO Journal. 26 (11): 2658–69. doi:10.1038/sj.emboj.7601699. PMC 1888663Freely accessible. PMID 17464285
- ↑ Wood, A. J. ; Schulz, R. ; Woodfine, K. ; Koltowska, K. ; Beechey, C. V. ; Peters, J. ; Bourc'his, D. ; Oakey, R. J. (2008). "Regulation of alternative polyadenylation by genomic imprinting". Genes & Development. 22 (9): 1141–6. doi:10.1101/gad.473408
- ↑ Jung, M. -Y. ; Lorenz, L. ; Richter, J. D. (2006). "Translational Control by Neuroguidin, a Eukaryotic Initiation Factor 4E and CPEB Binding Protein". Molecular and Cellular Biology. 26 (11): 4277–87. doi:10.1128/MCB.02470-05. PMC 1489097Freely accessible. PMID 16705177
- ↑ Sakurai, Takayuki; Sato, Masahiro; Kimura, Minoru (2005). "Diverse patterns of poly(A) tail elongation and shortening of murine maternal mRNAs from fully grown oocyte to 2-cell embryo stages". Biochemical and Biophysical Research Communications. 336 (4): 1181–9. doi:10.1016/j.bbrc.2005.08.250. PMID 16169522
- ↑ Taft, R (2008). "Virtues and limitations of the preimplantation mouse embryo as a model system". Theriogenology. 69 (1): 10–6. doi:10.1016/j.theriogenology.2007.09.032. PMC 2239213Freely accessible. PMID 18023855
- ↑ Richter, J (2007). "CPEB: a life in translation". Trends in Biochemical Sciences. 32 (6): 279–85. doi:10.1016/j.tibs.2007.04.004. PMID 17481902
- ↑ Piqué, Maria; López, José Manuel; Foissac, Sylvain; Guigó, Roderic; Méndez, Raúl (2008). "A Combinatorial Code for CPE-Mediated Translational Control". Cell. 132 (3): 434–48. doi:10.1016/j.cell.2007.12.038. PMID 18267074
- ↑ Benoit, P. ; Papin, C. ; Kwak, J. E. ; Wickens, M. ; Simonelig, M. (2008). "PAP- and GLD-2-type poly(A) polymerases are required sequentially in cytoplasmic polyadenylation and oogenesis in Drosophila". Development. 135 (11): 1969–79. doi:10.1242/dev.021444. PMID 18434412
- ↑ Reinisch, Karin M; Wolin, Sandra L (2007). "Emerging themes in non-coding RNA quality control". Current Opinion in Structural Biology. 17 (2): 209–14. doi:10.1016/j.sbi.2007.03.012. PMID 17395456
- ↑ Lacava, John; Houseley, Jonathan; Saveanu, Cosmin; Petfalski, Elisabeth; Thompson, Elizabeth; Jacquier, Alain; Tollervey, David (2005). "RNA Degradation by the Exosome Is Promoted by a Nuclear Polyadenylation Complex". Cell. 121 (5): 713–24. doi:10.1016/j.cell.2005.04.029. PMID 15935758
- ↑ Martin, G. ; Keller, W. (2007). "RNA-specific ribonucleotidyl transferases". RNA. 13 (11): 1834–49. doi:10.1261/rna.652807. PMC 2040100Freely accessible. PMID 17872511
- ↑ Slomovic, S. ; Laufer, D; Geiger, D; Schuster, G (2006). "Polyadenylation of ribosomal RNA in human cells". Nucleic Acids Research. 34 (10): 2966–75. doi:10.1093/nar/gkl357. PMC 1474067Freely accessible. PMID 16738135
- ↑ Régnier, Philippe; Arraiano, Cecília Maria (2000). "Degradation of mRNA in bacteria: emergence of ubiquitous features". BioEssays. 22 (3): 235–44. doi:10.1002/(SICI)1521-1878(200003)22:3<235::AID-BIES5>3.0.CO;2-2. PMID 10684583
- ↑ Anantharaman, V. ; Koonin, EV; Aravind, L (2002). "Comparative genomics and evolution of proteins involved in RNA metabolism". Nucleic Acids Research. 30 (7): 1427–64. doi:10.1093/nar/30.7.1427. PMC 101826Freely accessible. PMID 11917006
- ↑ Slomovic, Shimyn; Portnoy, Victoria; Liveanu, Varda; Schuster, Gadi (2006). "RNA Polyadenylation in Prokaryotes and Organelles; Different Tails Tell Different Tales". Critical Reviews in Plant Sciences. 25: 65–77. doi:10.1080/07352680500391337
- ↑ Chang, S. A. ; Cozad, M. ; MacKie, G. A. ; Jones, G. H. (2007). "Kinetics of Polynucleotide Phosphorylase: Comparison of Enzymes from Streptomyces and Escherichia coli and Effects of Nucleoside Diphosphates". Journal of Bacteriology. 190 (1): 98–106. doi:10.1128/JB.00327-07. PMC 2223728Freely accessible. PMID 17965156
- ↑ Nagaike, T; Suzuki, T; Ueda, T (2008). "Polyadenylation in mammalian mitochondria: Insights from recent studies". Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1779 (4): 266–9. doi:10.1016/j.bbagrm.2008.02.001
- ↑ Walter, M. ; Kilian, J; Kudla, J (2002). "PNPase activity determines the efficiency of mRNA 3'-end processing, the degradation of tRNA and the extent of polyadenylation in chloroplasts". The EMBO Journal. 21 (24): 6905–14. doi:10.1093/emboj/cdf686. PMC 139106Freely accessible. PMID 12486011
- ↑ Portnoy, V. ; Schuster, G. (2006). "RNA polyadenylation and degradation in different Archaea; roles of the exosome and RNase R". Nucleic Acids Research. 34 (20): 5923–31. doi:10.1093/nar/gkl763. PMC 1635327Freely accessible. PMID 17065466
- ↑ Yehudai-Resheff, S. (2003). "Domain Analysis of the Chloroplast Polynucleotide Phosphorylase Reveals Discrete Functions in RNA Degradation, Polyadenylation, and Sequence Homology with Exosome Proteins". The Plant Cell Online. 15 (9): 2003–19. doi:10.1105/tpc.013326
- ↑ Sarkar, Nilima (1997). "POLYADENYLATION OFmRNA IN PROKARYOTES". Annual Review of Biochemistry. 66: 173–97. doi:10.1146/annurev.biochem.66.1.173. PMID 9242905
- ↑ Slomovic, S; Portnoy, V; Schuster, G (2008). "RNA Turnover in Prokaryotes, Archaea and Organelles". Methods in Enzymology. 447: 501–20. doi:10.1016/S0076-6879(08)02224-6. شابک ۹۷۸−۰−۱۲−۳۷۴۳۷۷−۰.
- ↑ Portnoy, Victoria; Evguenieva-Hackenberg, Elena; Klein, Franziska; Walter, Pamela; Lorentzen, Esben; Klug, Gabriele; Schuster, Gadi (2005). "RNA polyadenylation in Archaea: not observed in Haloferax while the exosome polynucleotidylates RNA in Sulfolobus". EMBO Reports. 6 (12): 1188–93. doi:10.1038/sj.embor.7400571. PMC 1369208Freely accessible. PMID 16282984
- ↑ Portnoy, Victoria; Schuster, Gadi (2008). "Mycoplasma gallisepticum as the first analyzed bacterium in which RNA is not polyadenylated". FEMS Microbiology Letters. 283 (1): 97–103. doi:10.1111/j.1574-6968.2008.01157.x. PMID 18399989
- ↑ Evguenieva-Hackenberg, Elena; Roppelt, Verena; Finsterseifer, Pamela; Klug, Gabriele (2008). "Rrp4 and Csl4 Are Needed for Efficient Degradation but Not for Polyadenylation of Synthetic and Natural RNA by the Archaeal Exosome†". Biochemistry. 47 (50): 13158–68. doi:10.1021/bi8012214. PMID 19053279
- ↑ Slomovic, S; Portnoy, V; Yehudairesheff, S; Bronshtein, E; Schuster, G (2008). "Polynucleotide phosphorylase and the archaeal exosome as poly(A)-polymerases". Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1779 (4): 247–55. doi:10.1016/j.bbagrm.2007.12.004
- ↑ Edmonds, Mary; Abrams, Richard (1960). "Polynucleotide Biosynthesis: Formation of a Sequence of Adenylate Units from Adenosine Triphosphate by an Enzyme from Thymus Nuclei". The Journal of Biological Chemistry. 235 (4): 1142–9. PMID 13819354
- ↑ Colgan, D. F. ; Manley, J. L. (1997). "Mechanism and regulation of mRNA polyadenylation". Genes & Development. 11 (21): 2755–66. doi:10.1101/gad.11.21.2755
- ↑ Edmonds, M (2002). "Progress in Nucleic Acid Research and Molecular Biology Volume 71". Progress in Nucleic Acid Research and Molecular Biology. 71: 285–389. doi:10.1016/S0079-6603(02)71046-5. شابک ۹۷۸−۰−۱۲−۵۴۰۰۷۱−۸.
- ↑ Edmonds, M. (1971). "Polyadenylic Acid Sequences in the Heterogeneous Nuclear RNA and Rapidly-Labeled Polyribosomal RNA of HeLa cells: Possible Evidence for a Precursor Relationship". Proceedings of the National Academy of Sciences. 68 (6): 1336–40. doi:10.1073/pnas.68.6.1336
- مشارکتکنندگان ویکیپدیا. «Polyadenylation». در دانشنامهٔ ویکیپدیای انگلیسی.
