نانوفناوری دیانای
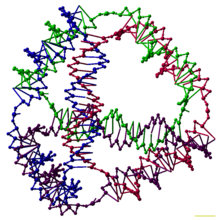
نانوفناوری دیانای (به انگلیسی: DNA nanotechnology) طراحی و ساخت ساختارهای نوکلئیک اسید مصنوعی برای مصارف تکنولوژی است. در این زمینه، اسیدهای نوکلئیک به عنوان مواد مهندسی غیربیولوژیکی برای نانوتکنولوژی و نه به عنوان حامل اطلاعات ژنتیکی در سلولهای زنده مورد استفاده قرار میگیرند. دانشمندان در این زمینه ساختارهای ایستایی مانند شبکههای کریستالی دو و سه بعدی، نانولولهها، شکلهای چندوجهی و دلخواه، و دستگاههای کاربردی مثل دستگاههای مولکولی و دیانای رایانه را ایجاد کردهاند. این زمینه به عنوان ابزاری برای حل مشکلات اساسی علوم در زیستشناسی ساختاری و بیوفیزیک، از جمله کاربردهای موجود در کریستالوگرافی اشعه ایکس و طیفسنجی رزونانس مغناطیسی هسته ای پروتئینها برای تعیین ساختار استفاده میشود. برنامههای کاربردی بالقوه در الکترونیک در مقیاس مولکولی و نانوپزشکی نیز مورد بررسی قرار گرفتهاست.
پایه مفهومی فناوری DNA اولین بار توسط نادریان سیمن در اوایل دهه۱۹۸۰ گذاشته شد و این زمینه در اواسط دهه۲۰۰۰ شروع به جلب توجه گسترده کرد. این استفاده از اسیدهای نوکلئیک با قوانین سخت جفتباز آنها امکانپذیر است، که باعث میشود فقط بخشهایی از رشتهها با توالی اسید نوکلئیک به یکدیگر متصل شوند تا ساختار مارپیچی دوتایی قوی و سفت و سخت تشکیل شود. این امر امکان طراحی منطقی توالیهای پایه را فراهم میکند که بهطور انتخابی جمع میشوند تا ساختارهای هدف پیچیدهای با ویژگیهای نانومقیاس دقیق کنترل شده را تشکیل دهند. چندین روش مونتاژ برای ساخت این سازهها استفاده میشود، از جمله سازههای مبتنی بر کاشی که از سازههای کوچکتر فراهم میشوند، سازههای تاشو با استفاده از روش دیانای اوریگامی و ساختارهای قابل تنظیم پویا با استفاده از روشهای جابجایی رشته. نام این زمینه بهطور خاص به DNA ارجاع میدهد، اما همین اصول با انواع دیگر اسیدهای نوکلئیک نیز به کار رفتهاست و منجر به استفاده گاهوبیگاه از نام جایگزین نانوتکنولوژی اسید نوکلئیک میشود.
طراحی
[ویرایش]نانوساختارهای DNA باید معقولانه طراحی شوند تا رشتههای اسیدنوکلئیک فردی در ساختارهای موردنظر جمع شوند. این روند معمولاً با مشخص کردن ساختار یا عملکرد هدف مورد نظر آغاز میشود. سپس ساختار دوم کلی کمپلکس هدف تعیین میشود، ترتیب رشتههای اسیدنوکلئیک درون ساختار را مشخص میکند، و کدام بخشهای آن رشتهها باید به یکدیگر متصل شوند. مرحله آخر طراحی ساختار اولیه است که مشخصات توالی پایه واقعی هر رشته اسیدنوکلئیک است.[۲][۳]
طراحی ساختاری
[ویرایش]اولین گام در طراحی نانوساختار نوکلئیک اسید این است که تصمیم بگیرید چگونه یک ساختار معین با یک ترکیب خاص از رشتههای اسیدنوکلئیک ارائه شود. این مرحله از طراحی ساختار ثانویه یا موقعیت جفت پایه که رشتههای جداگانه را در یک شکل مورد نظر نگه میدارند، تعیین میکند.[۲] رویکردهای مختلفی نشان داده شدهاست:
- سازههای مبتنی بر کاشی
- این رویکرد ساختار هدف را به واحدهای کوچکتر با اتصال قوی بین رشتههای موجود در هر واحد و تعامل ضعیف تر بین واحدها میشکند. این غالباً برای ساختن شبکههای دوره ای مورد استفاده قرار میگیرد، اما میتواند برای اجرای خودمونتاژ الگوریتمی نیز مورد استفاده قرار گیرد و آنها را به سکویی برای محاسبات DNA تبدیل کند. این استراتژی طراحی غالب است که از اواسط دهه ۱۹۹۰ تا اواسط دهه ۲۰۰۰ میلادی، هنگامی که روش دیانای اوریگامی استفاده شد، مورد استفاده قرار گرفت.[۲][۴]
- سازههای تاشو
- یک جایگزین برای رویکرد مبتنی بر کاشی، رویکردهای تاشو ساختار نانو را از یک رشته طولانی میسازد که یا میتواند یک توالی طراحی شده داشته باشد که به دلیل تعامل آن با خود برابر میشود، یا میتوان آن را با استفاده از رشته «اصلی» کوتاهتر به شکل دلخواه خم کرد. این روش اخیر اوریگامی DNA گفته میشود که اجازه میدهد شکلهای نانومقیاس دو و سه بعدی شکل بگیرد. (ساختارهای گسسته بالارا ببینید).[۵][۶]
- مونتاژ پویا
- این روش بهطور مستقیم کنترلکننده سینتیک خود مونتاژ DNA است و علاوه بر محصول نهایی، همه مراحل حدواسط در مکانیسم واکنش را مشخص میکند. این کار با استفاده از مواد اولیه که ساختار مویی را اتخاذ میکنند، انجام میشود؛ سپس اینها در یک واکنش آبشاری، با یک ترتیب خاص در ساختار نهایی جمع میشوند (آبشارهای جابه جایی رشته را در زیر مشاهده کنید). این روش از مزیت روند ایزوترمی در یک دمای ثابت برخوردار است. این بر خلاف رویکردهای ترمودینامیکی است، که در آن نیاز به یک مرحله بازپخت حرارتی است، که نیاز به یک تغییر دما برای تحریک مونتاژ و شکلگیری مناسب ساختار مورد نظر وجود دارد.[۵][۷]
طراحی توالی
[ویرایش]مواد و روشها
[ویرایش]
توالی رشتههای DNA که یک ساختار هدف را تشکیل میدهند، با استفاده از نرمافزار مدلسازی مولکولی و مدلسازی ترمودینامیکی بهطور محاسباتی طراحی شدهاند.[۳][۸] سپس اسیدهای نوکلئیک خود با استفاده از روشهای استاندارد سنتز الیگونوکلئوتید سنتز میشوند که معمولاً به صورت خودکار در یک ترکیب کننده الیگونوکلئوتید انجام میشوند و رشتههای دنبالههای سفارشی از نظر تجاری دردسترس هستند.[۹]
رشتهها میتوانند با الکتروفورز ژل دناتوره کننده در صورت لزوم خالص شوند،[۱۰] و غلظتهای دقیق از طریق هر یک از چندین روش سنجش نوکلئیک اسید با استفاده از طیفسنجی مرئی-فرابنفش تعیین میگردد.[۱۱]
ساختارهای هدف کاملاً شکل یافته را میتوان با استفاده از الکتروفورز ژل طبیعی، که اطلاعات اندازه و شکل را برای کمپلکسهای نوکلئیک اسید میدهد، تأیید کرد. یک روش تغییر تحرک الکتروفورز میتواند ارزیابی کند که آیا یک ساختار همهٔ رشتههای مورد نظر را شامل میشود یا خیر.[۱۲] برچسب زدن فلورسنت و انتقال انرژی رزونانسی فورستر(FRET) گاهی برای توصیف ساختار کمپلکسها مورد استفاده قرار میگیرد.[۱۳]
ساختارهای اسیدنوکلئیک را میتوان مستقیماً با میکروسکوپ نیروی اتمی نشان داد، که برای ساختارهای دو بعدی گسترده مناسب است، اما به دلیل تعامل نوک میکروسکوپ با ساختار اسیدنوکلئیک شکننده برای ساختارهای سه بعدی گسسته مفید نیست، میکروسکوپ الکترونی عبوری و میکروسکوپ الکترونی کرایو اغلب در این مورد استفاده میشود. شبکههای توسعه یافته سه بعدی توسط بلورشناسی پرتو ایکس تجزیه و تحلیل میشوند.[۱۴][۱۵]
جستارهای وابسته
[ویرایش]- en:International Society for Nanoscale Science, Computation, and Engineering
- en:Comparison of nucleic acid simulation software
- en:Molecular models of DNA
- زیستنانوفناوری
منابع
[ویرایش]- ↑ DNA polyhedra: Goodman, Russel P.; Schaap, Iwan A. T.; Tardin, C. F.; Erben, Christof M.; Berry, Richard M.; Schmidt, C.F.; Turberfield, Andrew J. (9 December 2005). "Rapid chiral assembly of rigid DNA building blocks for molecular nanofabrication". Science. 310 (5754): 1661–1665. Bibcode:2005Sci...310.1661G. doi:10.1126/science.1120367. PMID 16339440.
- ↑ ۲٫۰ ۲٫۱ ۲٫۲ Design: Feldkamp, U.; Niemeyer, C. M. (13 March 2006). "Rational design of DNA nanoarchitectures". Angewandte Chemie International Edition. 45 (12): 1856–1876. doi:10.1002/anie.200502358. PMID 16470892.
- ↑ ۳٫۰ ۳٫۱ خطای یادکرد: خطای یادکرد:برچسب
<ref> غیرمجاز؛ متنی برای یادکردهای با نامBrenneman02وارد نشده است. (صفحهٔ راهنما را مطالعه کنید.). - ↑ Overview: Lin, Chenxiang; Liu, Yan; Rinker, Sherri; Yan, Hao (11 August 2006). "DNA tile based self-assembly: building complex nanoarchitectures". ChemPhysChem. 7 (8): 1641–1647. doi:10.1002/cphc.200600260. PMID 16832805.
- ↑ ۵٫۰ ۵٫۱ خطای یادکرد: خطای یادکرد:برچسب
<ref> غیرمجاز؛ متنی برای یادکردهای با نامShihYanChallengesوارد نشده است. (صفحهٔ راهنما را مطالعه کنید.). - ↑ DNA origami: Rothemund, Paul W. K. (16 March 2006). "Folding DNA to create nanoscale shapes and patterns" (PDF). Nature. 440 (7082): 297–302. Bibcode:2006Natur.440..297R. doi:10.1038/nature04586. PMID 16541064. S2CID 4316391.
- ↑ Kinetic assembly: Yin, Peng; Choi, Harry M. T.; Calvert, Colby R.; Pierce, Niles A. (17 January 2008). "Programming biomolecular self-assembly pathways" (PDF). Nature. 451 (7176): 318–322. Bibcode:2008Natur.451..318Y. doi:10.1038/nature06451. PMID 18202654. S2CID 4354536.
- ↑ خطای یادکرد: خطای یادکرد:برچسب
<ref> غیرمجاز؛ متنی برای یادکردهای با نامparadigmsوارد نشده است. (صفحهٔ راهنما را مطالعه کنید.). - ↑ Methods: Ellington, A.; Pollard, J. D. (1 May 2001). Synthesis and Purification of Oligonucleotides. Current Protocols in Molecular Biology. Vol. Chapter 2. pp. 2.11.1–2.11.25. doi:10.1002/0471142727.mb0211s42. ISBN 978-0-471-14272-0. PMID 18265179.
- ↑ Methods: Ellington, A.; Pollard, J. D. (1 May 2001). Purification of Oligonucleotides Using Denaturing Polyacrylamide Gel Electrophoresis. Current Protocols in Molecular Biology. Vol. Chapter 2. pp. Unit2.12. doi:10.1002/0471142727.mb0212s42. ISBN 978-0-471-14272-0. PMID 18265180.
- ↑ Methods: Gallagher, S. R.; Desjardins, P. (1 July 2011). "Quantitation of nucleic acids and proteins". Current Protocols Essential Laboratory Techniques. doi:10.1002/9780470089941.et0202s5. ISBN 978-0-470-08993-4.
- ↑ Methods: Chory, J.; Pollard, J. D. (1 May 2001). Separation of Small DNA Fragments by Conventional Gel Electrophoresis. Current Protocols in Molecular Biology. Vol. Chapter 2. pp. Unit2.7. doi:10.1002/0471142727.mb0207s47. ISBN 978-0-471-14272-0. PMID 18265187.
- ↑ Methods: Walter, N. G. (1 February 2003). "Probing RNA structural dynamics and function by fluorescence resonance energy transfer (FRET)". Current Protocols in Nucleic Acid Chemistry. Current Protocols in Nucleic Acid Chemistry. Vol. Chapter 11. pp. 11.10.1–11.10.23. doi:10.1002/0471142700.nc1110s11. ISBN 978-0-471-14270-6. PMID 18428904.
- ↑ Methods: Lin, C.; Ke, Y.; Chhabra, R.; Sharma, J.; Liu, Y.; Yan, H. (2011). "Synthesis and Characterization of Self-Assembled DNA Nanostructures". In Zuccheri, G.; Samorì, B (eds.). DNA Nanotechnology: Methods and Protocols. Methods in Molecular Biology. Vol. 749. pp. 1–11. doi:10.1007/978-1-61779-142-0_1. ISBN 978-1-61779-141-3. PMID 21674361.
- ↑ Methods: Bloomfield, Victor A.; Crothers, Donald M.; Tinoco, Jr. , Ignacio (2000). Nucleic acids: structures, properties, and functions. Sausalito, Calif: University Science Books. pp. 84–86, 396–407. ISBN 978-0-935702-49-1.
{{cite book}}: نگهداری یادکرد:نامهای متعدد:فهرست نویسندگان (link)
پیوند به بیرون
[ویرایش]- What is Bionanotechnology?—a video introduction to DNA nanotechnology
