مطالعه همباشی سراسرژنوم: تفاوت میان نسخهها
بدون خلاصۀ ویرایش |
|||
| خط ۱: | خط ۱: | ||
در [[ژنتیک]] |
در [[ژنتیک]] یک '''مطالعه همخوانی سراسر ژنوم (به انگلیسی: genome-wide association study مخفف: GWAS یا GWA study)''' یک بررسی روی تنوعهای ژنتیکی سراسر ژنوم در افراد مختلف است و هدف آن مشاهده ارتباطات معنیدار (همبستگی، همخوانی) بین یک تنوع ژنتیکی (genetic variant) و روی دادن یک صفت (ظاهری) است. |
||
این مطالعات معمولاً روی بررسی ارتباط بین چندریختیهای تک-نوکلئوتیدی (SNP) و صفتهای انسانی مثل بیماریهای عمده انسانی تمرکز میکند اما میتوان از آن در بسیاری از ارگانیسمهای دیگر استفاده کرد. |
این مطالعات معمولاً روی بررسی ارتباط بین چندریختیهای تک-نوکلئوتیدی (SNP) و صفتهای انسانی مثل بیماریهای عمده انسانی تمرکز میکند اما میتوان از آن در بسیاری از ارگانیسمهای دیگر استفاده کرد. |
||
[[پرونده:Manhattan_Plot.png|جایگزین=Manhattan plot of a GWAS|بندانگشتی|546x546پیکسل| |
[[پرونده:Manhattan_Plot.png|جایگزین=Manhattan plot of a GWAS|بندانگشتی|546x546پیکسل|یک نمودار منهتن که چند [[جایگاه کروموزومی]] (loci) که به شدت مستعد همبستگی با مکانیزم میکروسیرکولاسیون (جریان خون در عروق بسیار کوچک و انتهایی) هستند را به تصویر میکشد. هر نقطه نمایندهٔ یک چندریختی تک-نوکلئوتیدی ([[چندریختی تک-نوکلئوتید|SNP]]) است که محور X نشاندهنده جایگاه ژنومیک چندریختی و محور Y منفی لگارتیم پی-مقدار را به عنوان تابعی از جایگاه کروموزومی نشان میدهد<ref name="pmid 21060863">{{cite journal|title=Four novel Loci (19q13, 6q24, 12q24, and 5q14) influence the microcirculation in vivo|date=October 2010|journal=PLoS Genet.|issue=10|doi=10.1371/journal.pgen.1001184|volume=6|pages=e1001184|pmc=2965750|pmid=21060863|display-authors=etal|author=Ikram MK|author2=Sim X|author3=Xueling S|editor1-last=McCarthy|editor1-first=Mark I}}</ref>]] |
||
زمانی که مطالعه روی دادههای انسانی اعمال شود، دیاناِی افراد شرکتکننده در مطالعه که در یک صفت یا بیماری خاص فنوتیپهای متفاوتی دارند مقایسه میشود. شرکتکنندگان در یک مطالعه همخوانی سراسر ژنوم به دو گروه با بیماری (یا صفت خاص) که به آنها مورد (case) و بدون بیماری (یا صفت) که به آنها شاهد (control) گفتهمیشود تقسیم میشوند. به این روش اول-فنوتیپ (first-phenotype) گفته میشود که در آن افراد ابتدا بر اساس ظاهر بالینی تقسیم میشوند و مقابل این روش اول-ژنوتیپ (first-genotype) مطرح است. بعد از این مرحله نمونه ژنتیکی افراد که همان دیانای است استخراج میگردد، حال اگر تناوب یک آلل (تنوع) خاص در گروه بیمار به طور معنیداری بیشتر از گروه شاهد باشد، مطالعه ادعا میکند آن تنوع با بیماری ''همبسته (همخوان یا مرتبط) ''است؛ بنابراین چندریختیهای همبسته با بیماری برای نشانهگذاری نواحی مرتبط با بیماری استفاده میشوند. در این مطالعه به جای در نظر گرفتن نواحی محدودی در ژنوم که مستعد همبستگی با بیماری (یا صفت) هستند، کل ژنوم را در نظر میگیریم، بنابراین به این رویکرد، غیر-نامزد-محور میگوییم که در مقابل رویکرد نامزد-محور قرار میگیرد. مطالعات همخوانی سراسر ژنوم توانایی یافتن ژنهایی را که ''دلیل'' رخدادن بیماریها هستند ندارد، گرچه با این مطالعات میتوان تنوّعهای همبسته با بیماریها را تشخیص داد. (دقت کنید رابطهٔ علیّت هم ارز رابطه همبستگی نیست)<ref name="pmid 20647212">{{Cite journal|title=Genomewide association studies and assessment of the risk of disease|last=Manolio TA|last2=Guttmacher|first2=Alan E.|date=July 2010|journal=N. Engl. J. Med.|issue=2|doi=10.1056/NEJMra0905980|volume=363|pages=166–76|pmid=20647212|last3=Manolio|first3=Teri A.}}</ref><ref name="pmid 18349094">{{cite journal|title=How to interpret a genome-wide association study|date=March 2008|journal=JAMA|issue=11|doi=10.1001/jama.299.11.1335|volume=299|pages=1335–44|pmid=18349094|author=Pearson TA|author2=Manolio TA}}</ref><ref>{{cite web|url=http://www.genome.gov/20019523|title=Genome-Wide Association Studies|publisher=[[National Human Genome Research Institute]]}}</ref> |
|||
نتایج اولین مطالعه موفق در سال ۲۰۰۵ منتشر شد. این مطالعه روی بیمارانی صورت گرفت که به [[تحلیلرفتن عضلانی]] مرتبط با سن دچار بودند، دو چندریختی (اسنیپ) یافت شد که به شکل معنیداری در تناوب آلل با گروه شاهد تفاوت داشت.<ref name="pmid 15761122">{{cite journal|title=Complement Factor H Polymorphism in Age-Related Macular Degeneration|date=April 2005|journal=Science|issue=5720|doi=10.1126/science.1109557|volume=308|pages=385–9|pmc=1512523|pmid=15761122|display-authors=4|author=Klein RJ|author2=Zeiss C|author3=Chew EY|author4=Tsai JY|author5=Sackler RS|author6=Haynes C|author7=Henning AK|author8=SanGiovanni JP|author9=Mane SM|author10=Mayne ST|author11=Bracken MB|author12=Ferris FL|author13=Ott J|author14=Barnstable C|author15=Hoh J}}</ref> |
|||
از سال ۲۰۱۱ صدها یا هزاران نفر آزمایش شدهاند، بیش از ۱۲۰۰ مطالعه همخوانی سراسر ژنوم انسانی روی بیش از ۲۰۰ بیماری و صفت صورت گرفته و تقریباً ۴۰۰۰ همبستگی برای چندریختیها (اسنیپ) کشف شدهاند.<ref name="pmid 19161620">{{Cite journal|title=An Open Access Database of Genome-wide Association Results|last=Johnson AD|last2=O'Donnell CJ|date=2009|journal=BMC Med. Genet.|doi=10.1186/1471-2350-10-6|volume=10|pages=6|pmc=2639349|pmid=19161620}}</ref> تعدادی از مطالعات با انتقاداتی مبنی بر عدم دقت در آزمایش همراه بودهاند، گرچه مطالعات جدید این مشکلات را مرتفع کردهاند. در هر حال روشهای مورد استفاده مخالفانی دارد. |
|||
== پیش زمینه == |
|||
[[پرونده:GWAS_Disease_allele_effects.png|بندانگشتی|340x340پیکسل|مطالعات همخوانی سراسر ژنوم به طور معمول تنوعهای رایج را با اندازه اثر کوچک میشناسند (''پایین سمت راست'').<ref name="pmid 23300413">{{Cite journal|title=Chapter 11: genome-wide association studies|last=Bush WS|last2=Moore JH|date=2012|journal=PLoS Comput Biol|issue=12|doi=10.1371/journal.pcbi.1002822|volume=8|pages=e1002822|pmc=3531285|pmid=23300413}}</ref>]] |
|||
هر دو [[ژنوم انسان]]، در میلیونها مورد با هم متفاوتند. این تفاوت به صورتهای مختلفی وجود دارد. تفاوتهای کوچک در تک نوکلئوتیدهای ژنوم که همان چندریختیها هستند، تفاوتهای بزرگتر مانند درجها حذفها و تنوعهای کپی-تعداد هر کدام از این تغییرات میتوانند باعث دگرشکلیهایی در صفات یا فنوتیپ شوند که میتواند بیماری، یا هر صفت با نمود فیزیکی باشد.<ref name="Strachan">{{cite book|title=Human Molecular Genetics|publisher=Garland Science|isbn=978-0-8153-4149-9|edition=4th|pages=467–495|author=Strachan T|author2=Read A}}</ref> در حدود سال ۲۰۰۰، پیش از معرفی مطالعه همخوانی سراسر ژنوم، روش اولیه تحقیق در این زمینه از طریق [[پیوند ژنتیکی]] در خانوادهها بود. این روش به عنوان یک روش بسیار کارا برای مطالعه [[اختلالات تک ژنی]] (اختلالاتی که تنها یک ژن در رخدادن آنها دخیل است) شناخته میشد.<ref>{{cite web|url=http://www.omim.org|title=Online Mendelian Inheritance in Man|accessdate=2011-12-06}}</ref> در هر حال برای بیماریهای رایج و پیچیده، نتایج حاصل از مطالعات پیوند ژنتیکی به سختی قابل تعمیم بودند.<ref name="Strachan">{{cite book|title=Human Molecular Genetics|publisher=Garland Science|isbn=978-0-8153-4149-9|edition=4th|pages=467–495|author=Strachan T|author2=Read A}}</ref><ref name="pmid 11565063">{{cite journal|title=Genomewide Scans of Complex Human Diseases: True Linkage Is Hard to Find|date=November 2001|journal=Am. J. Hum. Genet.|issue=5|doi=10.1086/324069|volume=69|pages=936–50|pmc=1274370|pmid=11565063|display-authors=4|author=Altmüller J|author2=Palmer LJ|author3=Fischer G|author4=Scherb H|author5=Wjst M}}</ref> یک پیشنهاد جایگزین برای مطالعات پیوندی، مطالعه همبستگی ژنتیکی بود. این نوع مطالعه میپرسد آیا [[الل|آلل]] یک تنوع ژنتیکی بیشتر از میزان مورد انتظار در افراد دارای فنوتیپ مورد علاقه یافت میشود یا نه. محاسبات اولیه روی توان آماری نشان دادند این رویکرد میتواند در تشخیص اثرهای ضعیف ژنتیکی بهتر عمل کند.<ref name="pmid 8801636">{{cite journal|title=The future of genetic studies of complex human diseases|date=September 1996|journal=Science|issue=5281|doi=10.1126/science.273.5281.1516|volume=273|pages=1516–7|pmid=8801636|author=Risch N|author2=Merikangas K}}</ref> |
|||
علاوه بر چارچوب مفهومی، چندین عامل دیگر هم مطالعات همبستگی سراسر ژنوم را ممکن کردند. یکی از این عوامل ظهور بیوبانکهاست، که مخزن مواد ژنتیکی انسان هستند که به میزان چشمگیری هزینه و دشواری جمعآوری تعداد کافی از نمونههای زیستی برای مطالعه را کاهش داد.<ref name="pmid 17550341">{{cite journal|title=The uneasy ethical and legal underpinnings of large-scale genomic biobanks|date=2007|journal=Annu Rev Genomics Hum Genet|doi=10.1146/annurev.genom.7.080505.115721|volume=8|pages=343–64|pmid=17550341|author=Greely HT}}</ref> پروژههای زیستی بزرگ مانند [[پروژه بینالمللی هپمپ]] و [[پروژه ۱۰۰۰ ژنوم]] نیز با شناخت چندریختیهای جدید به کمک مطالعات همبستگی آمدند.<ref name="pmid 14685227">{{cite journal|title=The International HapMap Project|date=December 2003|journal=Nature|issue=6968|doi=10.1038/nature02168|volume=426|pages=789–96|pmid=14685227|author=The International HapMap Project, Gibbs RA, Belmont JW, Hardenbol P, Willis TD, Yu F, Yang H, Ch'Ang L-Y, Huang W}}</ref><ref name="pmid 7569999">{{cite journal|title=Quantitative monitoring of gene expression patterns with a complementary DNA microarray|date=October 1995|journal=Science|issue=5235|doi=10.1126/science.270.5235.467|volume=270|pages=467–70|pmid=7569999|author=Schena M|author2=Shalon D|author3=Davis RW|author4=Brown PO}}</ref> |
|||
== روش == |
|||
[[پرونده:Method_example_for_GWA_study_designs.png|بندانگشتی|300x300پیکسل|یک محاسبه نمونه که روش مورد-شاهد را در مطالعه همخوانی سراسر ژنوم نشان میدهد. تعداد آلل برای هر اسنیپِ اندازهگیریشده بررسی شدهاست و از تست خی-مربع برای بررسی همبستگیها استفاده شده. این مثال از یک مطالعه روی بیماری عروق کرونر در سال ۲۰۰۷ برداشته شدهاست.<ref name="pmid 17554300">{{cite journal|title=Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls|date=June 2007|journal=Nature|issue=7145|doi=10.1038/nature05911|volume=447|pages=661–78|pmc=2719288|pmid=17554300|author=Wellcome Trust Case Control Consortium}}</ref>]] |
|||
رایجترین رویکرد در مطالعه همخوانی سراسر ژنوم روش مورد-شاهد است که دو گروه بزرگ از افراد که یکی سالم (شاهد) و دیگری متأثر از یک بیماری (مورد) هستند را مقایسه میکند. ژنوتیپ همه افراد در هر گروه برای رایجترین اسنیپهای شناخته شده استخراج میشود. تعداد دقیق اسنیپها به تکنولوژی استخراج دادهها بازمیگردد، اما معمولاً این مقدار یک میلیون یا بیشتر است.<ref name="pmid 23300413">{{Cite journal|title=Chapter 11: genome-wide association studies|last=Bush WS|last2=Moore JH|date=2012|journal=PLoS Comput Biol|issue=12|doi=10.1371/journal.pcbi.1002822|volume=8|pages=e1002822|pmc=3531285|pmid=23300413}}</ref> |
|||
برای هر کدام از این اسنیپها این آزمون انجام میشود که آیا تناوب آلل به شکل معنیداری بین دو گروه مورد و شاهد متفاوت است یا نه.<ref name="pmid 21293453">{{Cite journal|title=Basic statistical analysis in genetic case-control studies|last=Clarke GM|last2=Anderson CA|date=February 2011|journal=Nat Protoc|issue=2|doi=10.1038/nprot.2010.182|volume=6|pages=121–33|pmc=3154648|pmid=21293453|last3=Pettersson FH|last4=Cardon LR|last5=Morris AP|last6=Zondervan KT|display-authors=4}}</ref> در چنین آزمونهایی واحد پایهای برای بیان [[اندازه اثر]]، [[نسبت شانس]] (odds ratio) است. نسبت شانس نسبت دو شانس است که در زمینه مطالعه همبستگی سراسر ژنوم شانس بیماری برای افرادی با یک آلل خاص و شانس بیماری برای افراد بدون همان آلل اند. زمانی که تناوب آل در گروه مورد (بیمار یا داری صفت خاص) بسیار بیشتر از گروه شاهد باشد، نسبت شانس به طور معنیداری بیشتر از یک خواهد بود و این رویه برای تناوب کمتر برعکس است. علاوه بر این، معمولاً یک [[پی-مقدار]] ([[پی-مقدار|P-value]]) برای معنیدار بودن نسبت شانس است توسط یک آزمون خی-مربع (chi-squared test) ساده بدست میآید. پیدا کردن نسبت شانسهایی که به طور معنیداری با ۱ فاصله دارند هدف مطالعه سراسری ژنوم است زیرا این امر نمایانگر همبسته بودن اسنیپ (چندریختی) با بیماری خواهد بود.<ref name="pmid 21293453">{{Cite journal|title=Basic statistical analysis in genetic case-control studies|last=Clarke GM|last2=Anderson CA|date=February 2011|journal=Nat Protoc|issue=2|doi=10.1038/nprot.2010.182|volume=6|pages=121–33|pmc=3154648|pmid=21293453|last3=Pettersson FH|last4=Cardon LR|last5=Morris AP|last6=Zondervan KT|display-authors=4}}</ref> |
|||
انواع گوناگونی از رویکرد مورد-شاهد موجود است. یک جایگزین رایج برای مطالعه همبستگی مورد شاهدی، تحلیل دادههای فنوتیپیک کمّی است، مثلاً قد یا غلطت [[زیستنشان]]<nowiki/>ها یاحتی بررسی میزان بیان ژنها. به طور مشابه، آمارههای جایگزین که برای غالب (dominant) و مغلوب (recessive) طراحی شدهاند میتوانند استفاده شوند.<ref name="pmid 21293453">{{Cite journal|title=Basic statistical analysis in genetic case-control studies|last=Clarke GM|last2=Anderson CA|date=February 2011|journal=Nat Protoc|issue=2|doi=10.1038/nprot.2010.182|volume=6|pages=121–33|pmc=3154648|pmid=21293453|last3=Pettersson FH|last4=Cardon LR|last5=Morris AP|last6=Zondervan KT|display-authors=4}}</ref> محاسبات مطالعه معمولاً با یک نرمافزار بیوانفورماتیکی مانند SNPTEST و PLINK انجام میشود که شامل انواع آمارههای قابل استفاده هستند<ref>{{cite journal|title=Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls|date=2007|journal=Nature|issue=7145|doi=10.1038/nature05911|volume=447|pages=661–678|pmc=2719288|pmid=17554300}}</ref><ref name="pmid 17701901">{{cite journal|title=PLINK: A Tool Set for Whole-Genome Association and Population-Based Linkage Analyses|date=September 2007|journal=Am. J. Hum. Genet.|issue=3|doi=10.1086/519795|volume=81|pages=559–75|pmc=1950838|pmid=17701901|display-authors=4|author=Purcell S|author2=Neale B|author3=Todd-Brown K|author4=Thomas L|author5=Ferreira MA|author6=Bender D|author7=Maller J|author8=Sklar P|author9=de Bakker PI|author10=Daly MJ|author11=Sham PC}}</ref> |
|||
مطالعات پیشین همبستگی سراسر ژنوم روی تک اسنیپها تمرکز میکردند. در حالی که آزمایشها نشان میدهند که برهمکنشهای پیچیدهای بین دو یا چند اسنیپ روی میدهد که ممکن است در رخدادن بیماریهای پیچیده دخیل باشد که به این پدیده [[اپیستاسیس]] (epistasis) میگویند. به علاوه، محققان سعی میکنند تا دادههای همبستگی سراسر ژنوم را با بقیه دادههای زیستی مثل شبکه [[برهمکنشهای پروتئین-پروتئین]] مجتمع کنند تا بتوان نتایج حاوی اطلاعات بیشتری را بدست آورد.<ref>{{Cite journal|url=http://link.springer.com/article/10.1186/s13637-015-0025-6|title=MOBAS: identification of disease-associated protein subnetworks using modularity-based scoring|last=Ayati|first=Marzieh|last2=Erten|first2=Sinan|date=2015-06-30|journal=EURASIP Journal on Bioinformatics and Systems Biology|issue=1|doi=10.1186/s13637-015-0025-6|volume=2015|pages=1–14|language=en|issn=1687-4153|last3=Chance|first3=Mark R.|last4=Koyutürk|first4=Mehmet}}</ref><ref>{{Cite journal|url=http://doi.acm.org/10.1145/2808719.2808758|title=Assessing the Collective Disease Association of Multiple Genomic Loci|last=Ayati|first=Marzieh|last2=Koyutürk|first2=Mehmet|date=2015-01-01|journal=Proceedings of the 6th ACM Conference on Bioinformatics, Computational Biology and Health Informatics|publisher=ACM|doi=10.1145/2808719.2808758|series=BCB '15|location=New York, NY, USA|pages=376–385|isbn=978-1-4503-3853-0}}</ref> |
|||
یک گام کلیدی در اکثر مطالعات همبستگی سراسر ژنوم نسبتدادن ژنوتیپها به اسنیپها و نه به تراشه ژنوتیپ مورد استفاده در مطالعه است.<ref>{{Cite journal|title=Genotype imputation for genome-wide association studies|last=Marchini J|last2=Howie B|date=2010|journal=Nature Reviews Genetics|issue=7|doi=10.1038/nrg2796|volume=11|pages=499–511|pmid=20517342}}</ref> این روند تا حد زیادی تعداد اسنیپهایی که مورد آزمون میتوانند قرار گیرند را افزایش میدهد، توان مطالعه را بالا میبرد و فراتحلیل را روی گروههای مختلف فراهم میکند. نسبتدادن ژنوتیپ با روشهای آماری که دادههای مطالعه را با یک منبع مرجع از [[هپلوتایپ]]<nowiki/>ها ترکیب میکند، صورت میگیرد. |
|||
علاوه بر محاسبه همبستگی، رایج است که هر عامل که ممکن است به طور بالقوه نتیجه را مخدوش کند گزارش شود. جنسیت و سن رایجترین این [[عوامل مخدوشگر]] هستند. به علاوه، میدانیم که بسیاری از تمایزهای ژنتیکی با جوامع تاریخی و جغرافیایی همبستهاند.<ref name="pmid 18758442">{{Cite journal|title=Genes mirror geography within Europe|last=Novembre J|last2=Johnson T|date=November 2008|journal=Nature|issue=7218|doi=10.1038/nature07331|volume=456|pages=98–101|pmc=2735096|pmid=18758442|last3=Bryc K|last4=Kutalik Z|last5=Boyko AR|last6=Auton A|last7=Indap A|last8=King KS|last9=Bergmann S|display-authors=4}}</ref> به دلیل این همبستگی، مطالعات باید پیشینه قومی و جغرافیایی شرکتکنندگان را گزارش کنند که به این فرایند تعیین قشر جامعه میگویند. |
|||
پس از آنکه نسبهای شانس و پی-مقدارها برای همه اسنیپها محاسبه شدند، یک رویکرد رایج رسم یک نمودار منهتن است. در زمینه مطالعات همبستگی ژنوم، این نمودار منفی لگارتیم پی-مقدار را به عنوان تابعی از [[جایگاه کروموزومی]]، نشان میدهد؛ بنابراین اسنیپهایی که با بالاترین سطوح همبستگی در نمودار مشخص میشوند. آستانه پی-مقدار برای سطح معنیدار بودن به خاطر مسائل چند-آزمونی تصحیح میشود. آستانه دقیق برای آزمایشهای مختلف متفاوت است،<ref name="pmid 24473445">{{Cite journal|title=A novel computational biostatistics approach implies impaired dephosphorylation of growth factor receptors as associated with severity of autism|last=Wittkowski KM|last2=Sonakya V|date=January 2014|journal=Transl Psychiatry|issue=1|doi=10.1038/tp.2013.124|volume=4|pages=e354|pmc=3905234|pmid=24473445|last3=Bigio B|last4=Tonn MK|last5=Shic F|last6=Ascano M|last7=Nasca C|last8=Gold-Von Simson G|display-authors=4}}</ref> اما معمولاً آستانه <span style="white-space:nowrap">۵<span style="white-space:nowrap;margin-left:0.25em;margin-right:0.15em">×</span>۱۰<sup>−۸</sup></span> برای سطح معنی داری در هر مقیاسی کار میکند.<ref name="pmid23300413">{{Cite journal|title=Chapter 11: genome-wide association studies|last=Bush WS|last2=Moore JH|date=2012|journal=PLoS Comput Biol|issue=12|doi=10.1371/journal.pcbi.1002822|volume=8|pages=e1002822|pmc=3531285|pmid=23300413}}</ref><ref name="pmid 21293453">{{Cite journal|title=Basic statistical analysis in genetic case-control studies|last=Clarke GM|last2=Anderson CA|date=February 2011|journal=Nat Protoc|issue=2|doi=10.1038/nprot.2010.182|volume=6|pages=121–33|pmc=3154648|pmid=21293453|last3=Pettersson FH|last4=Cardon LR|last5=Morris AP|last6=Zondervan KT|display-authors=4}}</ref><ref>{{Cite journal|title=Guidelines for Genome-Wide Association Studies|last=Barsh GS|last2=Copenhaver GP|date=5 July 2012|journal=PLoS Genetics|issue=7|doi=10.1371/journal.pgen.1002812|volume=8|pages=e1002812|pmc=3390399|pmid=22792080|last3=Gibson G|last4=Williams SM}}</ref> |
|||
== نتایج == |
|||
[[پرونده:Regional_Association_Plot.png|بندانگشتی|نمودار همبستگی ناحیهای، نشاندهنده تک اسنیپها در ناحیه گیرنده LDL و همبستگی آنها با سطح منطقه و ارتباط آنها با سطح [[لیپوپروتئین کمچگالی|LDL-کلسترول]] است. نوع نمودار شبیه نمودار منهتن در بخش اول صفحه است اما برای قسمت محدودتری از ژننوم. [[هاپلوتیپ|ساختار]] هپلوبلاک با مقیاس رنگ نشان دادهشدهاست؛ و سطح همبستگی با محور Y نشان دادهشدهاست. ا نقطه به نمایندگی از rs73015013 SNP (بالا-وسط) بالا است و چون این اسنیپ تعدادی از تمایزهای LDL-کلسترول را توضیح میدهد.<ref name="pmid 21829380">{{Cite journal|title=Fine mapping of five loci associated with low-density lipoprotein cholesterol detects variants that double the explained heritability|last=Sanna S, Li B|last2=Mulas A|date=July 2011|journal=PLoS Genet.|issue=7|doi=10.1371/journal.pgen.1002198|volume=7|pages=e1002198|pmc=3145627|pmid=21829380|last3=Sidore C|last4=Kang HM|last5=Jackson AU|last6=Piras MG|last7=Usala G|last8=Maninchedda G|last9=Sassu A|display-authors=4}}</ref>]] |
|||
تلاشهایی برای تهیه یک فهرست جامع از اسنیپهایی که در مطالعات همبستگی سراسر ژنوم شناخته شدهبودند انجام شدهاست.<ref name="pmid 19474294">{{Cite journal|title=Potential etiologic and functional implications of genome-wide association loci for human diseases and traits|last=Hindorff LA|last2=Sethupathy P|date=June 2009|journal=Proc. Natl. Acad. Sci. U.S.A.|issue=23|doi=10.1073/pnas.0903103106|volume=106|pages=9362–7|pmc=2687147|pmid=19474294|last3=Junkins HA|last4=Ramos EM|last5=Mehta JP|last6=Collins FS|last7=Manolio TA|display-authors=4}}</ref> از سال ۲۰۰۹، هزاران اسنیپ همبسته با بیماریها به شناخته شدهاند.<ref name="pmid 19161620">{{Cite journal|title=An Open Access Database of Genome-wide Association Results|last=Johnson AD|last2=O'Donnell CJ|date=2009|journal=BMC Med. Genet.|doi=10.1186/1471-2350-10-6|volume=10|pages=6|pmc=2639349|pmid=19161620}}</ref> |
|||
اولین مطالعه همخوانی سراسر ژنوم، در سال ۲۰۰۵ انجام شد که در آن ۹۶ بیمار با تحلیل عضلانی مربوط به سن (ARMD) با ۵۰ شاهد مقایسه میشدند.<ref name="pmid 15761120">{{Cite journal|title=Complement Factor H Variant Increases the Risk of Age-Related Macular Degeneration|last=Haines JL|last2=Hauser MA|date=2005|journal=Science|issue=5720|doi=10.1126/science.1110359|volume=308|pages=419–421|pmid=15761120|last3=Schmidt S|last4=Scott WK|last5=Olson LM|last6=Gallins P|last7=Spencer KL|last8=Kwan SY|last9=Noureddine M|display-authors=4}}</ref> این مطالعه دو تا از اسنیپهایی که به شکل معنیداری تناوب آنها بین دو گروه متفاوت بود شناخته شدند. این اسنیپها روی فاکتور مکمل H قرار داشتند که یک دستآورد غیرمنتظره در مورد AMRD بود. یافتهها از این مطالعات اولیه به انجام پژوهشهای کاربردی بیشتر سرعت بخشید.<ref name="pmid 21860027">{{Cite journal|title=Design and development of TT30, a novel C3d-targeted C3/C5 convertase inhibitor for treatment of human complement alternative pathway–mediated diseases|last=Fridkis-Hareli M|last2=Storek M|date=October 2011|journal=Blood|issue=17|doi=10.1182/blood-2011-06-359646|volume=118|pages=4705–13|pmc=3208285|pmid=21860027|last3=Mazsaroff I|last4=Risitano AM|last5=Lundberg AS|last6=Horvath CJ|last7=Holers VM|display-authors=4}}</ref> یکی دیگر از نقاط عطف تاریخ این مطالعات مطالعه مورد-شاهدی کنسرسیوم ولکام تراست (WTCCC) در سال ۲۰۰۷ بود که بزرگترین مطالعه همخوانی سراسر ژنوم است که تا به حال انجام شدهاست. این مطالعه شامل ۱۴٬۰۰۰ مورد (بیمار) مبتلا به هفت بیماری شایع (~۲٬۰۰۰ نفر برای هر کدام از [[بیماری سرخرگ تاجی|عروق کرونر قلب، بیماری]]<nowiki/>های [[دیابت نوع یک|دیابت نوع ۱]]، با [[دیابت نوع ۲]]، با [[روماتیسم مفصلی|آرتریت روماتوئید]]، با [[بیماری کرون]]، با [[اختلال دوقطبی|اختلال دو قطبی]] و [[فشار خون بالا]]) و ۳۰۰۰ مورد مشترک بود.<ref name="WTCCC">{{Cite journal|title=Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls|last=Wellcome Trust Case Control Consortium, Burton PR|last2=Clayton DG|date=June 2007|journal=Nature|issue=7145|doi=10.1038/nature05911|volume=447|pages=661–78|pmc=2719288|pmid=17554300|last3=Cardon LR|display-authors=etal}}</ref> این مطالعه موفق به کشف بسیاری از ژنهای عامل این بیماریها است.<ref name="WTCCC">{{Cite journal|title=Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls|last=Wellcome Trust Case Control Consortium, Burton PR|last2=Clayton DG|date=June 2007|journal=Nature|issue=7145|doi=10.1038/nature05911|volume=447|pages=661–78|pmc=2719288|pmid=17554300|last3=Cardon LR|display-authors=etal}}</ref><ref>{{Cite press release|title=Largest ever study of genetics of common diseases published today|date=2007-06-06|publisher=Wellcome Trust Case Control Consortium|url=http://www.wtccc.org.uk/info/070606.shtml|accessdate=2008-06-19}}</ref> |
|||
بعد از این مطالعات بسیار مهم اولیه، دو روند کلی وجود داشتهاست.<ref name="pmid 19373277">{{Cite journal|url=http://www.ncbi.nlm.nih.gov/entrez/eutils/elink.fcgi?dbfrom=pubmed&tool=sumsearch.org/cite&retmode=ref&cmd=prlinks&id=19373277|title=Validating, augmenting and refining genome-wide association signals|last=Ioannidis JP|last2=Thomas G|date=2009|journal=Nat Rev Genet|issue=5|doi=10.1038/nrg2544|volume=10|pages=318–29|pmid=19373277|last3=Daly MJ}}</ref> یکی روند بررسی نمونههای بزرگ و بزرگتر بوده است. در پایان سال ۲۰۱۱، بزرگترین نمونهها در حدود ۲۰۰۰۰۰ نفر بود.<ref name="pmid 21909115">{{Cite journal|title=Genetic variants in novel pathways influence blood pressure and cardiovascular disease risk|last=Ehret GB|last2=Munroe PB|date=October 2011|journal=Nature|issue=7367|doi=10.1038/nature10405|volume=478|pages=103–9|pmc=3340926|pmid=21909115|last3=Rice KM|last4=Bochud M|last5=Johnson AD|last6=Chasman DI|last7=et al.|display-authors=4}}</ref> دلیل این ست که بتوان نسبت به نتایج فرض شده مطمئنتر بود. روند دیگر استفاده از قنوتیپهای با تعریف دقیقتر مانند [[چربی خون]]، [[پروانسولین]] یا زیستنشانهای مشابه بود.<ref name="pmid 19060906">{{Cite journal|title=Common variants at 30 loci contribute to polygenic dyslipidemia|last=Kathiresan S|last2=Willer CJ|date=January 2009|journal=Nat. Genet.|issue=1|doi=10.1038/ng.291|volume=41|pages=56–65|pmc=2881676|pmid=19060906|last3=Peloso GM|last4=Demissie S|last5=Musunuru K|last6=Schadt EE|last7=et al.|display-authors=4}}</ref><ref name="pmid 21873549">{{Cite journal|title=Genome-Wide Association Identifies Nine Common Variants Associated With Fasting Proinsulin Levels and Provides New Insights Into the Pathophysiology of Type 2 Diabetes|last=Strawbridge RJ|last2=Dupuis J|date=October 2011|journal=Diabetes|issue=10|doi=10.2337/db11-0415|volume=60|pages=2624–34|pmc=3178302|pmid=21873549|last3=Prokopenko I|last4=Barker A|last5=Ahlqvist E|last6=Rybin D|last7=et al.|display-authors=4}}</ref> این فنوتیپها به فنوتیپهای حد واسط معروفند و تحلیل آنها میتواند برای پروژهشهای کاربردی روی زیستنشانگرها با ارزش باشد<ref name="pmid 19901186">{{Cite journal|title=C-reactive protein and coronary disease: is there a causal link?|last=Danesh J|last2=Pepys MB|date=November 2009|journal=Circulation|issue=21|doi=10.1161/CIRCULATIONAHA.109.907212|volume=120|pages=2036–9|pmid=19901186}}</ref> |
|||
یک نقطه مرکزی بحث در GWA مطالعات بوده است که بسیاری از SNP تغییرات دربرداشت GWA مطالعات مرتبط با فقط با افزایش خطر ابتلا به این بیماری و فقط ارزش اخباری. میانه نسبت شانس است ۱٫۳۳ هر خطر-SNP تنها با چند نمایش شانس نسبت بالاتر از 3.0.<ref name="pmid 20647212">{{Cite journal|title=Genomewide association studies and assessment of the risk of disease|last=Manolio TA|last2=Guttmacher|first2=Alan E.|date=July 2010|journal=N. Engl. J. Med.|issue=2|doi=10.1056/NEJMra0905980|volume=363|pages=166–76|pmid=20647212|last3=Manolio|first3=Teri A.}}</ref><ref name="pmid 20300123">{{Cite journal|title=The pursuit of genome-wide association studies: where are we now?|last=Ku CS|last2=Loy EY|date=April 2010|journal=J. Hum. Genet.|issue=4|doi=10.1038/jhg.2010.19|volume=55|pages=195–206|pmid=20300123|last3=Pawitan Y|last4=Chia KS}}</ref> این قدر در نظر گرفته شده کوچک زیرا آنها را توضیح دهد مقدار زیادی از تنوع ارثی است. این ارثی تنوع شناخته شده است از وراثت مطالعات مبتنی بر [[دوقلو|همسان]]،<ref name="pmid 18987709">{{Cite journal|title=Personal genomes: The case of the missing heritability|last=Maher B|date=November 2008|journal=Nature|issue=7218|doi=10.1038/456018a|volume=456|pages=18–21|pmid=18987709}}</ref> به عنوان مثال مشخص شده است که ۸۰–۹۰٪ قد ارثی است. از این ۸۰–۹۰٪ اما GWA مطالعات حساب فقط به صورت یک اقلیت.<ref name="pmid 18987709">{{Cite journal|title=Personal genomes: The case of the missing heritability|last=Maher B|date=November 2008|journal=Nature|issue=7218|doi=10.1038/456018a|volume=456|pages=18–21|pmid=18987709}}</ref> |
|||
== منابع == |
== منابع == |
||
{{ |
{{پانویس|30em|۲}} |
||
<nowiki> |
|||
[[رده:ژنتیک]] |
[[رده:ژنتیک]] |
||
[[رده:پروژههای ژنوم انسانی]] |
[[رده:پروژههای ژنوم انسانی]]</nowiki> |
||
نسخهٔ ۳۱ دسامبر ۲۰۱۶، ساعت ۱۸:۵۷
در ژنتیک یک مطالعه همخوانی سراسر ژنوم (به انگلیسی: genome-wide association study مخفف: GWAS یا GWA study) یک بررسی روی تنوعهای ژنتیکی سراسر ژنوم در افراد مختلف است و هدف آن مشاهده ارتباطات معنیدار (همبستگی، همخوانی) بین یک تنوع ژنتیکی (genetic variant) و روی دادن یک صفت (ظاهری) است. این مطالعات معمولاً روی بررسی ارتباط بین چندریختیهای تک-نوکلئوتیدی (SNP) و صفتهای انسانی مثل بیماریهای عمده انسانی تمرکز میکند اما میتوان از آن در بسیاری از ارگانیسمهای دیگر استفاده کرد.
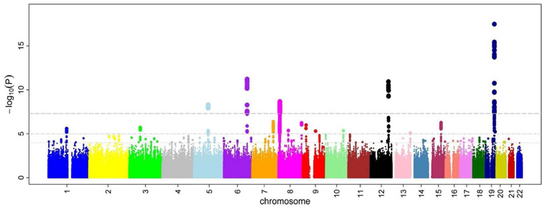
زمانی که مطالعه روی دادههای انسانی اعمال شود، دیاناِی افراد شرکتکننده در مطالعه که در یک صفت یا بیماری خاص فنوتیپهای متفاوتی دارند مقایسه میشود. شرکتکنندگان در یک مطالعه همخوانی سراسر ژنوم به دو گروه با بیماری (یا صفت خاص) که به آنها مورد (case) و بدون بیماری (یا صفت) که به آنها شاهد (control) گفتهمیشود تقسیم میشوند. به این روش اول-فنوتیپ (first-phenotype) گفته میشود که در آن افراد ابتدا بر اساس ظاهر بالینی تقسیم میشوند و مقابل این روش اول-ژنوتیپ (first-genotype) مطرح است. بعد از این مرحله نمونه ژنتیکی افراد که همان دیانای است استخراج میگردد، حال اگر تناوب یک آلل (تنوع) خاص در گروه بیمار به طور معنیداری بیشتر از گروه شاهد باشد، مطالعه ادعا میکند آن تنوع با بیماری همبسته (همخوان یا مرتبط) است؛ بنابراین چندریختیهای همبسته با بیماری برای نشانهگذاری نواحی مرتبط با بیماری استفاده میشوند. در این مطالعه به جای در نظر گرفتن نواحی محدودی در ژنوم که مستعد همبستگی با بیماری (یا صفت) هستند، کل ژنوم را در نظر میگیریم، بنابراین به این رویکرد، غیر-نامزد-محور میگوییم که در مقابل رویکرد نامزد-محور قرار میگیرد. مطالعات همخوانی سراسر ژنوم توانایی یافتن ژنهایی را که دلیل رخدادن بیماریها هستند ندارد، گرچه با این مطالعات میتوان تنوّعهای همبسته با بیماریها را تشخیص داد. (دقت کنید رابطهٔ علیّت هم ارز رابطه همبستگی نیست)[۲][۳][۴]
نتایج اولین مطالعه موفق در سال ۲۰۰۵ منتشر شد. این مطالعه روی بیمارانی صورت گرفت که به تحلیلرفتن عضلانی مرتبط با سن دچار بودند، دو چندریختی (اسنیپ) یافت شد که به شکل معنیداری در تناوب آلل با گروه شاهد تفاوت داشت.[۵]
از سال ۲۰۱۱ صدها یا هزاران نفر آزمایش شدهاند، بیش از ۱۲۰۰ مطالعه همخوانی سراسر ژنوم انسانی روی بیش از ۲۰۰ بیماری و صفت صورت گرفته و تقریباً ۴۰۰۰ همبستگی برای چندریختیها (اسنیپ) کشف شدهاند.[۶] تعدادی از مطالعات با انتقاداتی مبنی بر عدم دقت در آزمایش همراه بودهاند، گرچه مطالعات جدید این مشکلات را مرتفع کردهاند. در هر حال روشهای مورد استفاده مخالفانی دارد.
پیش زمینه
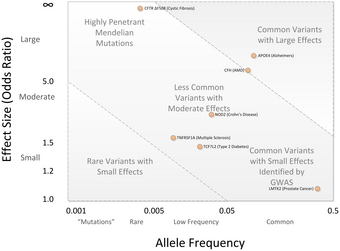
هر دو ژنوم انسان، در میلیونها مورد با هم متفاوتند. این تفاوت به صورتهای مختلفی وجود دارد. تفاوتهای کوچک در تک نوکلئوتیدهای ژنوم که همان چندریختیها هستند، تفاوتهای بزرگتر مانند درجها حذفها و تنوعهای کپی-تعداد هر کدام از این تغییرات میتوانند باعث دگرشکلیهایی در صفات یا فنوتیپ شوند که میتواند بیماری، یا هر صفت با نمود فیزیکی باشد.[۸] در حدود سال ۲۰۰۰، پیش از معرفی مطالعه همخوانی سراسر ژنوم، روش اولیه تحقیق در این زمینه از طریق پیوند ژنتیکی در خانوادهها بود. این روش به عنوان یک روش بسیار کارا برای مطالعه اختلالات تک ژنی (اختلالاتی که تنها یک ژن در رخدادن آنها دخیل است) شناخته میشد.[۹] در هر حال برای بیماریهای رایج و پیچیده، نتایج حاصل از مطالعات پیوند ژنتیکی به سختی قابل تعمیم بودند.[۸][۱۰] یک پیشنهاد جایگزین برای مطالعات پیوندی، مطالعه همبستگی ژنتیکی بود. این نوع مطالعه میپرسد آیا آلل یک تنوع ژنتیکی بیشتر از میزان مورد انتظار در افراد دارای فنوتیپ مورد علاقه یافت میشود یا نه. محاسبات اولیه روی توان آماری نشان دادند این رویکرد میتواند در تشخیص اثرهای ضعیف ژنتیکی بهتر عمل کند.[۱۱]
علاوه بر چارچوب مفهومی، چندین عامل دیگر هم مطالعات همبستگی سراسر ژنوم را ممکن کردند. یکی از این عوامل ظهور بیوبانکهاست، که مخزن مواد ژنتیکی انسان هستند که به میزان چشمگیری هزینه و دشواری جمعآوری تعداد کافی از نمونههای زیستی برای مطالعه را کاهش داد.[۱۲] پروژههای زیستی بزرگ مانند پروژه بینالمللی هپمپ و پروژه ۱۰۰۰ ژنوم نیز با شناخت چندریختیهای جدید به کمک مطالعات همبستگی آمدند.[۱۳][۱۴]
روش
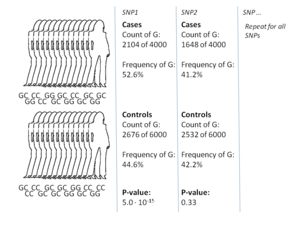
رایجترین رویکرد در مطالعه همخوانی سراسر ژنوم روش مورد-شاهد است که دو گروه بزرگ از افراد که یکی سالم (شاهد) و دیگری متأثر از یک بیماری (مورد) هستند را مقایسه میکند. ژنوتیپ همه افراد در هر گروه برای رایجترین اسنیپهای شناخته شده استخراج میشود. تعداد دقیق اسنیپها به تکنولوژی استخراج دادهها بازمیگردد، اما معمولاً این مقدار یک میلیون یا بیشتر است.[۷]
برای هر کدام از این اسنیپها این آزمون انجام میشود که آیا تناوب آلل به شکل معنیداری بین دو گروه مورد و شاهد متفاوت است یا نه.[۱۶] در چنین آزمونهایی واحد پایهای برای بیان اندازه اثر، نسبت شانس (odds ratio) است. نسبت شانس نسبت دو شانس است که در زمینه مطالعه همبستگی سراسر ژنوم شانس بیماری برای افرادی با یک آلل خاص و شانس بیماری برای افراد بدون همان آلل اند. زمانی که تناوب آل در گروه مورد (بیمار یا داری صفت خاص) بسیار بیشتر از گروه شاهد باشد، نسبت شانس به طور معنیداری بیشتر از یک خواهد بود و این رویه برای تناوب کمتر برعکس است. علاوه بر این، معمولاً یک پی-مقدار (P-value) برای معنیدار بودن نسبت شانس است توسط یک آزمون خی-مربع (chi-squared test) ساده بدست میآید. پیدا کردن نسبت شانسهایی که به طور معنیداری با ۱ فاصله دارند هدف مطالعه سراسری ژنوم است زیرا این امر نمایانگر همبسته بودن اسنیپ (چندریختی) با بیماری خواهد بود.[۱۶]
انواع گوناگونی از رویکرد مورد-شاهد موجود است. یک جایگزین رایج برای مطالعه همبستگی مورد شاهدی، تحلیل دادههای فنوتیپیک کمّی است، مثلاً قد یا غلطت زیستنشانها یاحتی بررسی میزان بیان ژنها. به طور مشابه، آمارههای جایگزین که برای غالب (dominant) و مغلوب (recessive) طراحی شدهاند میتوانند استفاده شوند.[۱۶] محاسبات مطالعه معمولاً با یک نرمافزار بیوانفورماتیکی مانند SNPTEST و PLINK انجام میشود که شامل انواع آمارههای قابل استفاده هستند[۱۷][۱۸]
مطالعات پیشین همبستگی سراسر ژنوم روی تک اسنیپها تمرکز میکردند. در حالی که آزمایشها نشان میدهند که برهمکنشهای پیچیدهای بین دو یا چند اسنیپ روی میدهد که ممکن است در رخدادن بیماریهای پیچیده دخیل باشد که به این پدیده اپیستاسیس (epistasis) میگویند. به علاوه، محققان سعی میکنند تا دادههای همبستگی سراسر ژنوم را با بقیه دادههای زیستی مثل شبکه برهمکنشهای پروتئین-پروتئین مجتمع کنند تا بتوان نتایج حاوی اطلاعات بیشتری را بدست آورد.[۱۹][۲۰]
یک گام کلیدی در اکثر مطالعات همبستگی سراسر ژنوم نسبتدادن ژنوتیپها به اسنیپها و نه به تراشه ژنوتیپ مورد استفاده در مطالعه است.[۲۱] این روند تا حد زیادی تعداد اسنیپهایی که مورد آزمون میتوانند قرار گیرند را افزایش میدهد، توان مطالعه را بالا میبرد و فراتحلیل را روی گروههای مختلف فراهم میکند. نسبتدادن ژنوتیپ با روشهای آماری که دادههای مطالعه را با یک منبع مرجع از هپلوتایپها ترکیب میکند، صورت میگیرد.
علاوه بر محاسبه همبستگی، رایج است که هر عامل که ممکن است به طور بالقوه نتیجه را مخدوش کند گزارش شود. جنسیت و سن رایجترین این عوامل مخدوشگر هستند. به علاوه، میدانیم که بسیاری از تمایزهای ژنتیکی با جوامع تاریخی و جغرافیایی همبستهاند.[۲۲] به دلیل این همبستگی، مطالعات باید پیشینه قومی و جغرافیایی شرکتکنندگان را گزارش کنند که به این فرایند تعیین قشر جامعه میگویند.
پس از آنکه نسبهای شانس و پی-مقدارها برای همه اسنیپها محاسبه شدند، یک رویکرد رایج رسم یک نمودار منهتن است. در زمینه مطالعات همبستگی ژنوم، این نمودار منفی لگارتیم پی-مقدار را به عنوان تابعی از جایگاه کروموزومی، نشان میدهد؛ بنابراین اسنیپهایی که با بالاترین سطوح همبستگی در نمودار مشخص میشوند. آستانه پی-مقدار برای سطح معنیدار بودن به خاطر مسائل چند-آزمونی تصحیح میشود. آستانه دقیق برای آزمایشهای مختلف متفاوت است،[۲۳] اما معمولاً آستانه ۵×۱۰−۸ برای سطح معنی داری در هر مقیاسی کار میکند.[۲۴][۱۶][۲۵]
نتایج

تلاشهایی برای تهیه یک فهرست جامع از اسنیپهایی که در مطالعات همبستگی سراسر ژنوم شناخته شدهبودند انجام شدهاست.[۲۷] از سال ۲۰۰۹، هزاران اسنیپ همبسته با بیماریها به شناخته شدهاند.[۶]
اولین مطالعه همخوانی سراسر ژنوم، در سال ۲۰۰۵ انجام شد که در آن ۹۶ بیمار با تحلیل عضلانی مربوط به سن (ARMD) با ۵۰ شاهد مقایسه میشدند.[۲۸] این مطالعه دو تا از اسنیپهایی که به شکل معنیداری تناوب آنها بین دو گروه متفاوت بود شناخته شدند. این اسنیپها روی فاکتور مکمل H قرار داشتند که یک دستآورد غیرمنتظره در مورد AMRD بود. یافتهها از این مطالعات اولیه به انجام پژوهشهای کاربردی بیشتر سرعت بخشید.[۲۹] یکی دیگر از نقاط عطف تاریخ این مطالعات مطالعه مورد-شاهدی کنسرسیوم ولکام تراست (WTCCC) در سال ۲۰۰۷ بود که بزرگترین مطالعه همخوانی سراسر ژنوم است که تا به حال انجام شدهاست. این مطالعه شامل ۱۴٬۰۰۰ مورد (بیمار) مبتلا به هفت بیماری شایع (~۲٬۰۰۰ نفر برای هر کدام از عروق کرونر قلب، بیماریهای دیابت نوع ۱، با دیابت نوع ۲، با آرتریت روماتوئید، با بیماری کرون، با اختلال دو قطبی و فشار خون بالا) و ۳۰۰۰ مورد مشترک بود.[۳۰] این مطالعه موفق به کشف بسیاری از ژنهای عامل این بیماریها است.[۳۰][۳۱]
بعد از این مطالعات بسیار مهم اولیه، دو روند کلی وجود داشتهاست.[۳۲] یکی روند بررسی نمونههای بزرگ و بزرگتر بوده است. در پایان سال ۲۰۱۱، بزرگترین نمونهها در حدود ۲۰۰۰۰۰ نفر بود.[۳۳] دلیل این ست که بتوان نسبت به نتایج فرض شده مطمئنتر بود. روند دیگر استفاده از قنوتیپهای با تعریف دقیقتر مانند چربی خون، پروانسولین یا زیستنشانهای مشابه بود.[۳۴][۳۵] این فنوتیپها به فنوتیپهای حد واسط معروفند و تحلیل آنها میتواند برای پروژهشهای کاربردی روی زیستنشانگرها با ارزش باشد[۳۶]
یک نقطه مرکزی بحث در GWA مطالعات بوده است که بسیاری از SNP تغییرات دربرداشت GWA مطالعات مرتبط با فقط با افزایش خطر ابتلا به این بیماری و فقط ارزش اخباری. میانه نسبت شانس است ۱٫۳۳ هر خطر-SNP تنها با چند نمایش شانس نسبت بالاتر از 3.0.[۲][۳۷] این قدر در نظر گرفته شده کوچک زیرا آنها را توضیح دهد مقدار زیادی از تنوع ارثی است. این ارثی تنوع شناخته شده است از وراثت مطالعات مبتنی بر همسان،[۳۸] به عنوان مثال مشخص شده است که ۸۰–۹۰٪ قد ارثی است. از این ۸۰–۹۰٪ اما GWA مطالعات حساب فقط به صورت یک اقلیت.[۳۸]
منابع
- ↑ Ikram MK; Sim X; Xueling S; et al. (October 2010). McCarthy, Mark I (ed.). "Four novel Loci (19q13, 6q24, 12q24, and 5q14) influence the microcirculation in vivo". PLoS Genet. 6 (10): e1001184. doi:10.1371/journal.pgen.1001184. PMC 2965750. PMID 21060863.
- ↑ ۲٫۰ ۲٫۱ Manolio TA; Guttmacher, Alan E.; Manolio, Teri A. (July 2010). "Genomewide association studies and assessment of the risk of disease". N. Engl. J. Med. 363 (2): 166–76. doi:10.1056/NEJMra0905980. PMID 20647212.
- ↑ Pearson TA; Manolio TA (March 2008). "How to interpret a genome-wide association study". JAMA. 299 (11): 1335–44. doi:10.1001/jama.299.11.1335. PMID 18349094.
- ↑ "Genome-Wide Association Studies". National Human Genome Research Institute.
- ↑ Klein RJ; Zeiss C; Chew EY; Tsai JY; et al. (April 2005). "Complement Factor H Polymorphism in Age-Related Macular Degeneration". Science. 308 (5720): 385–9. doi:10.1126/science.1109557. PMC 1512523. PMID 15761122.
- ↑ ۶٫۰ ۶٫۱ Johnson AD; O'Donnell CJ (2009). "An Open Access Database of Genome-wide Association Results". BMC Med. Genet. 10: 6. doi:10.1186/1471-2350-10-6. PMC 2639349. PMID 19161620.
- ↑ ۷٫۰ ۷٫۱ Bush WS; Moore JH (2012). "Chapter 11: genome-wide association studies". PLoS Comput Biol. 8 (12): e1002822. doi:10.1371/journal.pcbi.1002822. PMC 3531285. PMID 23300413.
- ↑ ۸٫۰ ۸٫۱ Strachan T; Read A. Human Molecular Genetics (4th ed.). Garland Science. pp. 467–495. ISBN 978-0-8153-4149-9.
- ↑ "Online Mendelian Inheritance in Man". Retrieved 2011-12-06.
- ↑ Altmüller J; Palmer LJ; Fischer G; Scherb H; et al. (November 2001). "Genomewide Scans of Complex Human Diseases: True Linkage Is Hard to Find". Am. J. Hum. Genet. 69 (5): 936–50. doi:10.1086/324069. PMC 1274370. PMID 11565063.
- ↑ Risch N; Merikangas K (September 1996). "The future of genetic studies of complex human diseases". Science. 273 (5281): 1516–7. doi:10.1126/science.273.5281.1516. PMID 8801636.
- ↑ Greely HT (2007). "The uneasy ethical and legal underpinnings of large-scale genomic biobanks". Annu Rev Genomics Hum Genet. 8: 343–64. doi:10.1146/annurev.genom.7.080505.115721. PMID 17550341.
- ↑ The International HapMap Project, Gibbs RA, Belmont JW, Hardenbol P, Willis TD, Yu F, Yang H, Ch'Ang L-Y, Huang W (December 2003). "The International HapMap Project". Nature. 426 (6968): 789–96. doi:10.1038/nature02168. PMID 14685227.
{{cite journal}}: نگهداری یادکرد:نامهای متعدد:فهرست نویسندگان (link) - ↑ Schena M; Shalon D; Davis RW; Brown PO (October 1995). "Quantitative monitoring of gene expression patterns with a complementary DNA microarray". Science. 270 (5235): 467–70. doi:10.1126/science.270.5235.467. PMID 7569999.
- ↑ Wellcome Trust Case Control Consortium (June 2007). "Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls". Nature. 447 (7145): 661–78. doi:10.1038/nature05911. PMC 2719288. PMID 17554300.
- ↑ ۱۶٫۰ ۱۶٫۱ ۱۶٫۲ ۱۶٫۳ Clarke GM; Anderson CA; Pettersson FH; Cardon LR; et al. (February 2011). "Basic statistical analysis in genetic case-control studies". Nat Protoc. 6 (2): 121–33. doi:10.1038/nprot.2010.182. PMC 3154648. PMID 21293453.
- ↑ "Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls". Nature. 447 (7145): 661–678. 2007. doi:10.1038/nature05911. PMC 2719288. PMID 17554300.
- ↑ Purcell S; Neale B; Todd-Brown K; Thomas L; et al. (September 2007). "PLINK: A Tool Set for Whole-Genome Association and Population-Based Linkage Analyses". Am. J. Hum. Genet. 81 (3): 559–75. doi:10.1086/519795. PMC 1950838. PMID 17701901.
- ↑ Ayati, Marzieh; Erten, Sinan; Chance, Mark R.; Koyutürk, Mehmet (2015-06-30). "MOBAS: identification of disease-associated protein subnetworks using modularity-based scoring". EURASIP Journal on Bioinformatics and Systems Biology (به انگلیسی). 2015 (1): 1–14. doi:10.1186/s13637-015-0025-6. ISSN 1687-4153.
- ↑ Ayati, Marzieh; Koyutürk, Mehmet (2015-01-01). "Assessing the Collective Disease Association of Multiple Genomic Loci". Proceedings of the 6th ACM Conference on Bioinformatics, Computational Biology and Health Informatics. BCB '15. New York, NY, USA: ACM: 376–385. doi:10.1145/2808719.2808758. ISBN 978-1-4503-3853-0.
- ↑ Marchini J; Howie B (2010). "Genotype imputation for genome-wide association studies". Nature Reviews Genetics. 11 (7): 499–511. doi:10.1038/nrg2796. PMID 20517342.
- ↑ Novembre J; Johnson T; Bryc K; Kutalik Z; et al. (November 2008). "Genes mirror geography within Europe". Nature. 456 (7218): 98–101. doi:10.1038/nature07331. PMC 2735096. PMID 18758442.
- ↑ Wittkowski KM; Sonakya V; Bigio B; Tonn MK; et al. (January 2014). "A novel computational biostatistics approach implies impaired dephosphorylation of growth factor receptors as associated with severity of autism". Transl Psychiatry. 4 (1): e354. doi:10.1038/tp.2013.124. PMC 3905234. PMID 24473445.
- ↑ Bush WS; Moore JH (2012). "Chapter 11: genome-wide association studies". PLoS Comput Biol. 8 (12): e1002822. doi:10.1371/journal.pcbi.1002822. PMC 3531285. PMID 23300413.
- ↑ Barsh GS; Copenhaver GP; Gibson G; Williams SM (5 July 2012). "Guidelines for Genome-Wide Association Studies". PLoS Genetics. 8 (7): e1002812. doi:10.1371/journal.pgen.1002812. PMC 3390399. PMID 22792080.
- ↑ Sanna S, Li B; Mulas A; Sidore C; Kang HM; et al. (July 2011). "Fine mapping of five loci associated with low-density lipoprotein cholesterol detects variants that double the explained heritability". PLoS Genet. 7 (7): e1002198. doi:10.1371/journal.pgen.1002198. PMC 3145627. PMID 21829380.
- ↑ Hindorff LA; Sethupathy P; Junkins HA; Ramos EM; et al. (June 2009). "Potential etiologic and functional implications of genome-wide association loci for human diseases and traits". Proc. Natl. Acad. Sci. U.S.A. 106 (23): 9362–7. doi:10.1073/pnas.0903103106. PMC 2687147. PMID 19474294.
- ↑ Haines JL; Hauser MA; Schmidt S; Scott WK; et al. (2005). "Complement Factor H Variant Increases the Risk of Age-Related Macular Degeneration". Science. 308 (5720): 419–421. doi:10.1126/science.1110359. PMID 15761120.
- ↑ Fridkis-Hareli M; Storek M; Mazsaroff I; Risitano AM; et al. (October 2011). "Design and development of TT30, a novel C3d-targeted C3/C5 convertase inhibitor for treatment of human complement alternative pathway–mediated diseases". Blood. 118 (17): 4705–13. doi:10.1182/blood-2011-06-359646. PMC 3208285. PMID 21860027.
- ↑ ۳۰٫۰ ۳۰٫۱ Wellcome Trust Case Control Consortium, Burton PR; Clayton DG; Cardon LR; et al. (June 2007). "Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls". Nature. 447 (7145): 661–78. doi:10.1038/nature05911. PMC 2719288. PMID 17554300.
- ↑ "Largest ever study of genetics of common diseases published today" (Press release). Wellcome Trust Case Control Consortium. 2007-06-06. Retrieved 2008-06-19.
- ↑ Ioannidis JP; Thomas G; Daly MJ (2009). "Validating, augmenting and refining genome-wide association signals". Nat Rev Genet. 10 (5): 318–29. doi:10.1038/nrg2544. PMID 19373277.
- ↑ Ehret GB; Munroe PB; Rice KM; Bochud M; et al. (October 2011). "Genetic variants in novel pathways influence blood pressure and cardiovascular disease risk". Nature. 478 (7367): 103–9. doi:10.1038/nature10405. PMC 3340926. PMID 21909115.
{{cite journal}}: Explicit use of et al. in:|last7=(help) - ↑ Kathiresan S; Willer CJ; Peloso GM; Demissie S; et al. (January 2009). "Common variants at 30 loci contribute to polygenic dyslipidemia". Nat. Genet. 41 (1): 56–65. doi:10.1038/ng.291. PMC 2881676. PMID 19060906.
{{cite journal}}: Explicit use of et al. in:|last7=(help) - ↑ Strawbridge RJ; Dupuis J; Prokopenko I; Barker A; et al. (October 2011). "Genome-Wide Association Identifies Nine Common Variants Associated With Fasting Proinsulin Levels and Provides New Insights Into the Pathophysiology of Type 2 Diabetes". Diabetes. 60 (10): 2624–34. doi:10.2337/db11-0415. PMC 3178302. PMID 21873549.
{{cite journal}}: Explicit use of et al. in:|last7=(help) - ↑ Danesh J; Pepys MB (November 2009). "C-reactive protein and coronary disease: is there a causal link?". Circulation. 120 (21): 2036–9. doi:10.1161/CIRCULATIONAHA.109.907212. PMID 19901186.
- ↑ Ku CS; Loy EY; Pawitan Y; Chia KS (April 2010). "The pursuit of genome-wide association studies: where are we now?". J. Hum. Genet. 55 (4): 195–206. doi:10.1038/jhg.2010.19. PMID 20300123.
- ↑ ۳۸٫۰ ۳۸٫۱ Maher B (November 2008). "Personal genomes: The case of the missing heritability". Nature. 456 (7218): 18–21. doi:10.1038/456018a. PMID 18987709.
[[رده:ژنتیک]] [[رده:پروژههای ژنوم انسانی]]
