ترمیم جفتشدن ناجور دیانای
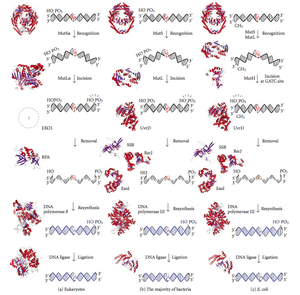
ترمیم جفتشدن ناجور دیانای[۱] (به انگلیسی: DNA mismatch repair (MMR))، یا تعمیر ناجوری دیانای[۲] سیستمی برای شناسایی و بازسازی درج، حذف و ترکیب نادرست بازهای نوکلئوتیدی است که میتواند در طول همانندسازی و نوترکیبی دیانای ایجاد شود، و همچنین بازسازی برخی از شکلهای آسیب دیانای انجام شود.[۳][۴]
هر رویداد جهشی که ساختار ابرمارپیچ دیانای را مختل کند، پتانسیل به خطر انداختن ثبات ژنتیکی یک سلول را به همراه دارد. این واقعیت که سیستمهای تشخیص و بازسازی آسیب به اندازه خود ماشینهای تکثیر پیچیده هستند، اهمیت فرگشت را به وفاداری دیانای نشان میدهد.
اجزای MMR در انسان
[ویرایش]در انسان، هفت پروتئین ترمیم جفتشدن ناجور دیانای (MMR) (MLH1، MLH3، MSH2، MSH3، MSH6، PMS1 و PMS2) بهطور هماهنگ در مراحل متوالی برای آغاز ترمیم جفتشدن ناجور دیانای کار میکنند.[۵] علاوه بر این، مسیرهای فرعی MMR وابسته به Exo1 و مستقل از Exo1 وجود دارد.[۶]
دیگر فرآوردههای ژنی دخیل در جفتشدن ناجور (پس از آغاز توسط ژنهای MMR) در انسان عبارتند از دیانای پلیمراز دلتا، PCNA , RPA , HMGB1، RFC و دیانای لیگاز I، همراه با عوامل اصلاحکننده هیستون و کروماتین.[۷][۸]
اصلاح هیستون H3K36me3، نشانهٔ اپیژنتیک کروماتین فعال، توانایی بهکارگیری کمپلکس MSH2-MSH6 (hMutSα) را دارد.[۹] بهطور مداوم، مناطقی از ژنوم انسان با سطوح بالایی از H3K36me3 جهشهای کمتری را به دلیل فعالیت MMR جمع میکنند.[۱۰]
منابع
[ویرایش]- ↑ یاسایی، وحیدرضا (۱۳۸۹). درسنامۀ ژنتیک - مقدمات علوم پایه. تهران: گروه ژنتیک دانشکدۀ پزشکی دانشگاه شهید بهشتی. ص. ۱۴۷.
- ↑ «ام. اس. آی». مؤسسه تحقيقات، آموزش و درمان سرطان.
- ↑ Iyer RR, Pluciennik A, Burdett V, Modrich PL (February 2006). "DNA mismatch repair: functions and mechanisms". Chemical Reviews. 106 (2): 302–23. doi:10.1021/cr0404794. PMID 16464007.
- ↑ Larrea AA, Lujan SA, Kunkel TA (May 2010). "SnapShot: DNA mismatch repair". Cell. 141 (4): 730–730.e1. doi:10.1016/j.cell.2010.05.002. PMID 20478261.
- ↑ Pal T, Permuth-Wey J, Sellers TA (August 2008). "A review of the clinical relevance of mismatch-repair deficiency in ovarian cancer". Cancer. 113 (4): 733–42. doi:10.1002/cncr.23601. PMC 2644411. PMID 18543306.
- ↑ Goellner EM, Putnam CD, Kolodner RD (August 2015). "Exonuclease 1-dependent and independent mismatch repair". DNA Repair. 32: 24–32. doi:10.1016/j.dnarep.2015.04.010. PMC 4522362. PMID 25956862.
- ↑ Li GM (January 2008). "Mechanisms and functions of DNA mismatch repair". Cell Research. 18 (1): 85–98. doi:10.1038/cr.2007.115. PMID 18157157.
- ↑ Li GM (July 2014). "New insights and challenges in mismatch repair: getting over the chromatin hurdle". DNA Repair. 19: 48–54. doi:10.1016/j.dnarep.2014.03.027. PMC 4127414. PMID 24767944.
- ↑ Li F, Mao G, Tong D, Huang J, Gu L, Yang W, Li GM (April 2013). "The histone mark H3K36me3 regulates human DNA mismatch repair through its interaction with MutSα". Cell. 153 (3): 590–600. doi:10.1016/j.cell.2013.03.025. PMC 3641580. PMID 23622243.
- ↑ Supek F, Lehner B (July 2017). "Clustered Mutation Signatures Reveal that Error-Prone DNA Repair Targets Mutations to Active Genes". Cell. 170 (3): 534–547.e23. doi:10.1016/j.cell.2017.07.003. PMID 28753428.
