ک تایی: تفاوت میان نسخهها
برچسبها: ویرایش همراه ویرایش از وبگاه همراه ویرایش پیشرفتهٔ همراه |
F.labbaf97 (بحث | مشارکتها) بند معرفی اضافه شد. |
||
| خط ۱: | خط ۱: | ||
در [[بیوانفورماتیک]]، k-تایی یک زیر رشته به طول k از رشته بیولوژیکی است. در ابتدا، k-تایی در [[ژنومیک محاسباتی]] و [[آنالیز دنباله]] استفاده میشد که در آن K-تایی تشکیل شده از [[نوکلئوتید|نوکلئوتایدها]] (A,T, C,G) هستند. k-تاییها به خاطر [[بازسازی توالی|بازسازی توالیهای DNA]]<ref>{{Cite journal|last=Compeau|first=Phillip E C|last2=Pevzner|first2=Pavel A|last3=Tesler|first3=Glenn|date=2011-11|title=How to apply de Bruijn graphs to genome assembly|url=http://www.nature.com/articles/nbt.2023|journal=Nature Biotechnology|language=en|volume=29|issue=11|pages=987–991|doi=10.1038/nbt.2023|issn=1087-0156|pmc=PMC5531759|pmid=22068540}}</ref>، بهبود بیان ژن heterologous <ref>{{Cite journal|last=Gustafsson|first=Claes|last2=Minshull|first2=Jeremy|last3=Gurney|first3=Austin|last4=Villalobos|first4=Alan|last5=Ness|first5=Jon E.|last6=Govindarajan|first6=Sridhar|last7=Welch|first7=Mark|date=2009-09-14|title=Design Parameters to Control Synthetic Gene Expression in Escherichia coli|url=https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0007002|journal=PLOS ONE|language=en|volume=4|issue=9|pages=e7002|doi=10.1371/journal.pone.0007002|issn=1932-6203|pmc=PMC2736378|pmid=19759823}}</ref><ref>{{Cite journal|last=Minshull|first=Jeremy|last2=Govindarajan|first2=Sridhar|last3=Gustafsson|first3=Claes|date=2004-07-01|title=Codon bias and heterologous protein expression|url=https://www.cell.com/trends/biotechnology/abstract/S0167-7799(04)00111-8|journal=Trends in Biotechnology|language=English|volume=22|issue=7|pages=346–353|doi=10.1016/j.tibtech.2004.04.006|issn=0167-7799|pmid=15245907}}</ref>، شناسایی گونه ها در نمونه های متاژنومی<ref>{{Cite journal|last=Beiko|first=Robert G.|last2=Perry|first2=Scott C.|date=2010-01-01|title=Distinguishing Microbial Genome Fragments Based on Their Composition: Evolutionary and Comparative Genomic Perspectives|url=https://academic.oup.com/gbe/article/doi/10.1093/gbe/evq004/568285|journal=Genome Biology and Evolution|language=en|volume=2|pages=117–131|doi=10.1093/gbe/evq004|pmc=PMC2839357|pmid=20333228}}</ref>، و تولید [[واکسن زنده]]<ref>{{Cite journal|last=Kunec|first=Dusan|last2=Osterrieder|first2=Nikolaus|last3=Trimpert|first3=Jakob|last4=Eschke|first4=Kathrin|date=2018-01-29|title=Attenuation of a very virulent Marek's disease herpesvirus (MDV) by codon pair bias deoptimization|url=https://journals.plos.org/plospathogens/article?id=10.1371/journal.ppat.1006857|journal=PLOS Pathogens|language=en|volume=14|issue=1|pages=e1006857|doi=10.1371/journal.ppat.1006857|issn=1553-7374|pmc=PMC5805365|pmid=29377958}}</ref> مطرح شدند. معمولا کلمه k-تایی به همه رشتههای دنبالهای به طول k اشاره دارد. به طوریکه رشتهی AGAT میتواند ۴ [[مونومر]] (A , G , A , T)، سه ۲-تایی (AG, GA, AT)، دو ۳-تایی (AGA, GAT) و یک ۴-تایی داشته باشد. که زیررشته ۴-تایی برابر خود رشته است. به طور کلی یک رشته به طول L میتواند L-k+1 رشته k-تایی داشته باشد. همچنین برای n مونومر، n^k تا k-تایی ممکن وجود دارد. که در رشته DNA تعداد مونومرها برابر با ۴ است. |
در [[بیوانفورماتیک]]، k-تایی یک زیر رشته به طول k از رشته بیولوژیکی است. در ابتدا، k-تایی در [[ژنومیک محاسباتی]] و [[آنالیز دنباله]] استفاده میشد که در آن K-تایی تشکیل شده از [[نوکلئوتید|نوکلئوتایدها]] (A,T, C,G) هستند. k-تاییها به خاطر [[بازسازی توالی|بازسازی توالیهای DNA]]<ref>{{Cite journal|last=Compeau|first=Phillip E C|last2=Pevzner|first2=Pavel A|last3=Tesler|first3=Glenn|date=2011-11|title=How to apply de Bruijn graphs to genome assembly|url=http://www.nature.com/articles/nbt.2023|journal=Nature Biotechnology|language=en|volume=29|issue=11|pages=987–991|doi=10.1038/nbt.2023|issn=1087-0156|pmc=PMC5531759|pmid=22068540}}</ref>، بهبود بیان ژن heterologous <ref>{{Cite journal|last=Gustafsson|first=Claes|last2=Minshull|first2=Jeremy|last3=Gurney|first3=Austin|last4=Villalobos|first4=Alan|last5=Ness|first5=Jon E.|last6=Govindarajan|first6=Sridhar|last7=Welch|first7=Mark|date=2009-09-14|title=Design Parameters to Control Synthetic Gene Expression in Escherichia coli|url=https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0007002|journal=PLOS ONE|language=en|volume=4|issue=9|pages=e7002|doi=10.1371/journal.pone.0007002|issn=1932-6203|pmc=PMC2736378|pmid=19759823}}</ref><ref>{{Cite journal|last=Minshull|first=Jeremy|last2=Govindarajan|first2=Sridhar|last3=Gustafsson|first3=Claes|date=2004-07-01|title=Codon bias and heterologous protein expression|url=https://www.cell.com/trends/biotechnology/abstract/S0167-7799(04)00111-8|journal=Trends in Biotechnology|language=English|volume=22|issue=7|pages=346–353|doi=10.1016/j.tibtech.2004.04.006|issn=0167-7799|pmid=15245907}}</ref>، شناسایی گونه ها در نمونه های متاژنومی<ref>{{Cite journal|last=Beiko|first=Robert G.|last2=Perry|first2=Scott C.|date=2010-01-01|title=Distinguishing Microbial Genome Fragments Based on Their Composition: Evolutionary and Comparative Genomic Perspectives|url=https://academic.oup.com/gbe/article/doi/10.1093/gbe/evq004/568285|journal=Genome Biology and Evolution|language=en|volume=2|pages=117–131|doi=10.1093/gbe/evq004|pmc=PMC2839357|pmid=20333228}}</ref>، و تولید [[واکسن زنده]]<ref>{{Cite journal|last=Kunec|first=Dusan|last2=Osterrieder|first2=Nikolaus|last3=Trimpert|first3=Jakob|last4=Eschke|first4=Kathrin|date=2018-01-29|title=Attenuation of a very virulent Marek's disease herpesvirus (MDV) by codon pair bias deoptimization|url=https://journals.plos.org/plospathogens/article?id=10.1371/journal.ppat.1006857|journal=PLOS Pathogens|language=en|volume=14|issue=1|pages=e1006857|doi=10.1371/journal.ppat.1006857|issn=1553-7374|pmc=PMC5805365|pmid=29377958}}</ref> مطرح شدند. معمولا کلمه k-تایی به همه رشتههای دنبالهای به طول k اشاره دارد. به طوریکه رشتهی AGAT میتواند ۴ [[مونومر]] (A , G , A , T)، سه ۲-تایی (AG, GA, AT)، دو ۳-تایی (AGA, GAT) و یک ۴-تایی داشته باشد. که زیررشته ۴-تایی برابر خود رشته است. به طور کلی یک رشته به طول L میتواند L-k+1 رشته k-تایی داشته باشد. همچنین برای n مونومر، n^k تا k-تایی ممکن وجود دارد. که در رشته DNA تعداد مونومرها برابر با ۴ است. |
||
== معرفی == |
|||
k-تاییها زیر رشتههایی به طول k هستند. برای مثال در جدول زیر تمام k-تاییها برای یک رشته نمونه از DNA را مشاهده میکنید. |
|||
{| class="wikitable" |
|||
|+k-تاییها برای رشته GTAGAGCTGT |
|||
!''k'' |
|||
!''k''-تایی |
|||
|- |
|||
|1 |
|||
|G, T, A, G, A, G, C, T, G, T |
|||
|- |
|||
|2 |
|||
|GT, TA, AG, GA, AG, GC, CT, TG, GT |
|||
|- |
|||
|3 |
|||
|GTA, TAG, AGA, GAG, AGC, GCT, CTG, TGT |
|||
|- |
|||
|4 |
|||
|GTAG, TAGA, AGAG, GAGC, AGCT, GCTG, CTGT |
|||
|- |
|||
|5 |
|||
|GTAGA, TAGAG, AGAGC, GAGCT, AGCTG, GCTGT |
|||
|- |
|||
|6 |
|||
|GTAGAG, TAGAGC, AGAGCT, GAGCTG, AGCTGT |
|||
|- |
|||
|7 |
|||
|GTAGAGC, TAGAGCT, AGAGCTG, GAGCTGT |
|||
|- |
|||
|8 |
|||
|GTAGAGCT, TAGAGCTG, AGAGCTGT |
|||
|- |
|||
|9 |
|||
|GTAGAGCTG, TAGAGCTGT |
|||
|- |
|||
|10 |
|||
|GTAGAGCTGT |
|||
|} |
|||
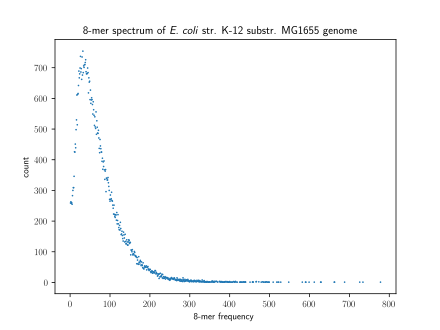
یک روش برای نمایش دادن k-تاییها استفاده از طیف k-تایی است. طیف k-تایی یک نمایش گرافیکی از یک پایگاه داده است که نشان میدهد چند تا k-تایی به تعداد مشخصی تکرار شدهاند. بنابراین محور x آن فرکانس تکرار k-تایی را نشان میدهد و محور y تعداد k-تاییهایی که به آن اندازه تکرار شدهاند.<ref>{{Cite journal|last=Clavijo|first=Bernardo J.|last2=Wright|first2=Jonathan|last3=Kettleborough|first3=George|last4=Garcia Accinelli|first4=Gonzalo|last5=Mapleson|first5=Daniel|date=2017-02-15|title=KAT: a K-mer analysis toolkit to quality control NGS datasets and genome assemblies|url=https://academic.oup.com/bioinformatics/article/33/4/574/2664339|journal=Bioinformatics|language=en|volume=33|issue=4|pages=574–576|doi=10.1093/bioinformatics/btw663|issn=1367-4803|pmc=PMC5408915|pmid=27797770}}</ref> شکل توزیع طیف k-تایی اطلاعات مفیدی از ویژگیهای نمونه بیولوژیکی را به ما میدهد.<ref>{{Cite journal|last=Chor|first=Benny|last2=Horn|first2=David|last3=Goldman|first3=Nick|last4=Levy|first4=Yaron|last5=Massingham|first5=Tim|date=2009|title=Genomic DNA k-mer spectra: models and modalities|url=https://www.ncbi.nlm.nih.gov/pubmed/19814784|journal=Genome Biology|volume=10|issue=10|pages=R108|doi=10.1186/gb-2009-10-10-r108|issn=1474-760X|pmc=2784323|pmid=19814784}}</ref> تعداد مدها در توزیع طیف k-تایی برای ژنوم یک گونه میتواند متفاوت باشد. در این بین [[:en:Unimodality|تکمدی]]<nowiki/>ها بیشترین آمار تعداد گونه را دارند. در حالیکه تمام [[پستانداران]] توزیع طیف k-تایی [[:en:Multimodality|چندمدی]] دارند. همچنین در یک طیف k-تایی، توزیع در مناطق مختلف ژنوم میتواند متفاوت باشد. برای مثال انسانها در [[:en:Five_prime_untranslated_region|مناطق ترجمه نشده '5]] و [[اگزون|اگزونها]] طیف تکمدی دارند و در [[:en:Three_prime_untranslated_region|مناطق ترجمه نشده '3]] و [[اینترون|اینترونها]] طیف چندمدی دارند. |
|||
[[File:E. coli 8-mer spectrum.svg|thumb|مثالی از ۸-تایی برای [[اشریشیا کلی]] که فرکانس تکرار ۸-تاییها را در مقابل تعداد تکرار آن نشان میدهد.|alt=|440x440px|راست]] |
|||
== منابع == |
== منابع == |
||
نسخهٔ ۱۶ ژوئیهٔ ۲۰۱۹، ساعت ۱۴:۳۰
در بیوانفورماتیک، k-تایی یک زیر رشته به طول k از رشته بیولوژیکی است. در ابتدا، k-تایی در ژنومیک محاسباتی و آنالیز دنباله استفاده میشد که در آن K-تایی تشکیل شده از نوکلئوتایدها (A,T, C,G) هستند. k-تاییها به خاطر بازسازی توالیهای DNA[۱]، بهبود بیان ژن heterologous [۲][۳]، شناسایی گونه ها در نمونه های متاژنومی[۴]، و تولید واکسن زنده[۵] مطرح شدند. معمولا کلمه k-تایی به همه رشتههای دنبالهای به طول k اشاره دارد. به طوریکه رشتهی AGAT میتواند ۴ مونومر (A , G , A , T)، سه ۲-تایی (AG, GA, AT)، دو ۳-تایی (AGA, GAT) و یک ۴-تایی داشته باشد. که زیررشته ۴-تایی برابر خود رشته است. به طور کلی یک رشته به طول L میتواند L-k+1 رشته k-تایی داشته باشد. همچنین برای n مونومر، n^k تا k-تایی ممکن وجود دارد. که در رشته DNA تعداد مونومرها برابر با ۴ است.
معرفی
k-تاییها زیر رشتههایی به طول k هستند. برای مثال در جدول زیر تمام k-تاییها برای یک رشته نمونه از DNA را مشاهده میکنید.
| k | k-تایی |
|---|---|
| 1 | G, T, A, G, A, G, C, T, G, T |
| 2 | GT, TA, AG, GA, AG, GC, CT, TG, GT |
| 3 | GTA, TAG, AGA, GAG, AGC, GCT, CTG, TGT |
| 4 | GTAG, TAGA, AGAG, GAGC, AGCT, GCTG, CTGT |
| 5 | GTAGA, TAGAG, AGAGC, GAGCT, AGCTG, GCTGT |
| 6 | GTAGAG, TAGAGC, AGAGCT, GAGCTG, AGCTGT |
| 7 | GTAGAGC, TAGAGCT, AGAGCTG, GAGCTGT |
| 8 | GTAGAGCT, TAGAGCTG, AGAGCTGT |
| 9 | GTAGAGCTG, TAGAGCTGT |
| 10 | GTAGAGCTGT |
یک روش برای نمایش دادن k-تاییها استفاده از طیف k-تایی است. طیف k-تایی یک نمایش گرافیکی از یک پایگاه داده است که نشان میدهد چند تا k-تایی به تعداد مشخصی تکرار شدهاند. بنابراین محور x آن فرکانس تکرار k-تایی را نشان میدهد و محور y تعداد k-تاییهایی که به آن اندازه تکرار شدهاند.[۶] شکل توزیع طیف k-تایی اطلاعات مفیدی از ویژگیهای نمونه بیولوژیکی را به ما میدهد.[۷] تعداد مدها در توزیع طیف k-تایی برای ژنوم یک گونه میتواند متفاوت باشد. در این بین تکمدیها بیشترین آمار تعداد گونه را دارند. در حالیکه تمام پستانداران توزیع طیف k-تایی چندمدی دارند. همچنین در یک طیف k-تایی، توزیع در مناطق مختلف ژنوم میتواند متفاوت باشد. برای مثال انسانها در مناطق ترجمه نشده '5 و اگزونها طیف تکمدی دارند و در مناطق ترجمه نشده '3 و اینترونها طیف چندمدی دارند.
منابع
- ↑ Compeau, Phillip E C; Pevzner, Pavel A; Tesler, Glenn (2011-11). "How to apply de Bruijn graphs to genome assembly". Nature Biotechnology (به انگلیسی). 29 (11): 987–991. doi:10.1038/nbt.2023. ISSN 1087-0156. PMC 5531759. PMID 22068540.
{{cite journal}}: Check date values in:|date=(help)نگهداری یادکرد:فرمت پارامتر PMC (link) - ↑ Gustafsson, Claes; Minshull, Jeremy; Gurney, Austin; Villalobos, Alan; Ness, Jon E.; Govindarajan, Sridhar; Welch, Mark (2009-09-14). "Design Parameters to Control Synthetic Gene Expression in Escherichia coli". PLOS ONE (به انگلیسی). 4 (9): e7002. doi:10.1371/journal.pone.0007002. ISSN 1932-6203. PMC 2736378. PMID 19759823.
{{cite journal}}: نگهداری یادکرد:فرمت پارامتر PMC (link) - ↑ Minshull, Jeremy; Govindarajan, Sridhar; Gustafsson, Claes (2004-07-01). "Codon bias and heterologous protein expression". Trends in Biotechnology (به انگلیسی). 22 (7): 346–353. doi:10.1016/j.tibtech.2004.04.006. ISSN 0167-7799. PMID 15245907.
- ↑ Beiko, Robert G.; Perry, Scott C. (2010-01-01). "Distinguishing Microbial Genome Fragments Based on Their Composition: Evolutionary and Comparative Genomic Perspectives". Genome Biology and Evolution (به انگلیسی). 2: 117–131. doi:10.1093/gbe/evq004. PMC 2839357. PMID 20333228.
{{cite journal}}: نگهداری یادکرد:فرمت پارامتر PMC (link) - ↑ Kunec, Dusan; Osterrieder, Nikolaus; Trimpert, Jakob; Eschke, Kathrin (2018-01-29). "Attenuation of a very virulent Marek's disease herpesvirus (MDV) by codon pair bias deoptimization". PLOS Pathogens (به انگلیسی). 14 (1): e1006857. doi:10.1371/journal.ppat.1006857. ISSN 1553-7374. PMC 5805365. PMID 29377958.
{{cite journal}}: نگهداری یادکرد:فرمت پارامتر PMC (link) - ↑ Clavijo, Bernardo J.; Wright, Jonathan; Kettleborough, George; Garcia Accinelli, Gonzalo; Mapleson, Daniel (2017-02-15). "KAT: a K-mer analysis toolkit to quality control NGS datasets and genome assemblies". Bioinformatics (به انگلیسی). 33 (4): 574–576. doi:10.1093/bioinformatics/btw663. ISSN 1367-4803. PMC 5408915. PMID 27797770.
{{cite journal}}: نگهداری یادکرد:فرمت پارامتر PMC (link) - ↑ Chor, Benny; Horn, David; Goldman, Nick; Levy, Yaron; Massingham, Tim (2009). "Genomic DNA k-mer spectra: models and modalities". Genome Biology. 10 (10): R108. doi:10.1186/gb-2009-10-10-r108. ISSN 1474-760X. PMC 2784323. PMID 19814784.
