مجموعه کلاهکبرداری
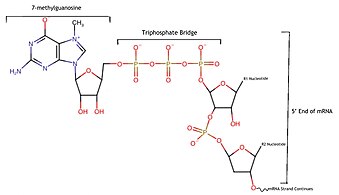
مجموعهٔ کلاهکبرداری آرانای پیامرسان یک مجموعهٔ پروتئینی در سلولهای یوکاریوتی است که مسئول حذف کلاهک ۵' است.[۱] آنزیم فعال مجموعهٔ کلاهکبرداری، آنزیمی از خانوادهٔ نودیکس (Nudix) با نام Dcp2 است که کلاهک ۵' را هیدرولیز میکند و 7mGDP و آرانای پیامرسان ۵'-مونوفسفریلهشده را آزاد میکند.[۱] این آرانای پیامرسان جداشده، ترجمه نشده و توسط اگزونوکلئازها تجزیه میشود.[۲][۳] همراه با بسیاری از پروتئینهای جانبی دیگر، مجموعهٔ کلاهکبرداری، در اجسام پی در سیتوپلاسم جمع میشود.
هدف از کلاهکبرداری[ویرایش]
آرانای پیامرسان پس از استفاده در سلولها باید تجزیه شود، در غیر این صورت در اطراف سلول شناور میشود و پروتئینهای ناخواسته را بهطور تصادفی ایجاد میکند. کلاهک ۵' بهطور خاصی طراحی شده تا از تجزیهٔ آرانای پیامرسان پیش از استفاده، جلوگیری کند؛ بنابراین، کلاهک باید برداشته شود تا مسیر فروپاشی آرانای پیامرسان بتواند آن را تجزیه و حذف کند.[۴]
سازوکار[ویرایش]
Dcp2 پروتئینی است که در واقع آرانای پیامرسان را کلاهکبرداری میکند و سایر پروتئینهای موجود در مجموعه، عملکرد آن را افزایش میدهند و به آن اجازه میدهند پیوند شیمیایی متصلکنندهٔ آرانای پیامرسان به کلاهک ۵' را هیدرولیز کند.[۵] دامنهٔ نودیکس در Dcp2 یکی از پیوندهای روی پُل تریفسفات را که آرانای پیامرسان و کلاهک ۵' را بههم متصل میکند، هیدرولیز میکند و باعث میشود کلاهک ۷-متیلگوانوزین جدا شده و آرانای پیامرسان برای تجزیه توسط اگزونوکلئازها در سلول، آماده شود.[۶]
ساختار[ویرایش]
هم جانداران تکسلولی و هم گونههای چندسلولی باید آرانای پیامرسان خود را پس از استفاده، کلاهکبرداری و در نهایت، حذف کنند اما جانداران مختلف، پروتئینهای کمی متفاوتی دارند که این فرایند را انجام میدهند. پروتئینهای زیادی در جانداران مختلف، مشترک هستند اما چندین تفاوت کلیدی نیز میان مجموعههای کلاهکبرداری در میان جانداران تکسلولی (مانند مخمر) و چندسلولی (مانند جانوران) وجود دارد.[۵]
منابع[ویرایش]
- ↑ ۱٫۰ ۱٫۱ Mugridge, Jeffrey S; Ziemniak, Marcin; Jemielity, Jacek; Gross, John D (November 2016). "Structural basis of mRNA cap recognition by Dcp1–Dcp2". Nature Structural & Molecular Biology. 23 (11): 987–994. doi:10.1038/nsmb.3301. ISSN 1545-9993. PMC 5113729. PMID 27694842.
- ↑ Chantarachot T, Bailey-Serres J (January 2018). "Polysomes, Stress Granules, and Processing Bodies: A Dynamic Triumvirate Controlling Cytoplasmic mRNA Fate and Function". Plant Physiology. 176 (1): 254–269. doi:10.1104/pp.17.01468. PMC 5761823. PMID 29158329.
- ↑ Sieburth LE, Vincent JN (2018-12-17). "Beyond transcription factors: roles of mRNA decay in regulating gene expression in plants". F1000Research. 7: 1940. doi:10.12688/f1000research.16203.1. PMC 6305221. PMID 30613385.
- ↑ Beelman, C. A.; Parker, R. (1995-04-21). "Degradation of mRNA in eukaryotes". Cell. 81 (2): 179–183. doi:10.1016/0092-8674(95)90326-7. ISSN 0092-8674. PMID 7736570.
- ↑ ۵٫۰ ۵٫۱ Charenton, Clément; Graille, Marc (2018-12-19). "mRNA decapping: finding the right structures". Philosophical Transactions of the Royal Society B: Biological Sciences. 373 (1762): 20180164. doi:10.1098/rstb.2018.0164. PMC 6232594. PMID 30397101.
- ↑ Beelman, C. A.; Parker, R. (1995-04-21). "Degradation of mRNA in eukaryotes". Cell. 81 (2): 179–183. doi:10.1016/0092-8674(95)90326-7. ISSN 0092-8674. PMID 7736570.
