تنوع تعداد کپی
تنوع تعداد کپی یا تکرار، یک زمینه جدید در علم ژنتیک است که به بررسی پدیدهای میپردازد که در طی آن قسمتی از ژنوم فرد در طول آن تکرار میشود و تعداد این تکرار برای اشخاص متفاوت با یکدیگر فرق میکند.[۱] تنوع تعداد تکرار یک نوع تنوع ساختاری در ژنوم موجودات محسوب میشود و به ویژه یک نوع مضاعف شدن و حذف در ژنوم است که تعداد زیادی از نوکلئوتیدها را درگیر میکند.[۲] اگرچه، امروزه مطالعات در ژنتیک مدرن بر روی ژنوم انسان تمرکز دارد، این پدیده در سایر موجودات از جمله باکتری اشریشیا کلی نیز دیده شدهاست.[۳] مطالعات اخیر نشان دادهاست که تقریباً دو سوم کل ژنوم انسان از نواحی تکرار تشکیل شدهاست.[۴] در پستانداران، تنوع تعداد تکرار نقش مهمی را در ایجاد تنوع ضروری در یک جمعیت از جمله فنوتایپهای یک بیماری ایفا میکند.[۱]
انواع تنوع تعداد تکرار را میتوان در دو گروه دستهبندی کرد: تکرارهای کوتاه و تکرارهای بلند. با این وجود، مرز مشخصی میان دو گروه وجود ندارد و دستهبندی بیشتر وابسته به ویژگیهای جایگاه کروموزومی مورد نظر دارد. تکرارهای کوتاه بیشتر شامل تکرارهای دوگانه (همانند A-C-A-C-A-CC) و سهگانه هستند. تکرارهای طولانی شامل تکرار یک ژن میشوند. این دستهبندی بر مبنای اندازه واضحترین نوع دستهبندی است زیرا اندازه یک عامل بسیار مهم در تعیین نوع مکانیزمی است که منجر به وقوع تکرار شدهاست.[۵]
گونهها و تغییرات کروموزومی[ویرایش]

یکی از مشهورترین تکرارهای کوتاه، تکرار قطعه سه نوکلئوتیدی CAG در ژن هانتینگتون است که مسئول بروز بیماری عصبی هانتینگتون است.[۶] هنگامی که تعداد تکرار CAG در طول کروموزوم از ۴۴–۴۶ بگذرد، بیماری هانتینگتون در فرد شروع به توسعه میکند.[۶] همچنین احتمال به ارث رسیدن این عارضه توسط یکی از فرزندان بالا میرود. به علاوه، تعداد تکرارهای قطعه سه نوکلئوتیدی CAG ارتباط زیادی به سن آغاز این بیماری در افراد دارد. اینگونه از تکرارهای کوتاه معمولاً در اثر خطای پلیمراز در طول فرایند همانندسازی صورت میگیرد که از جمله میتوان به لغزش پلیمراز، تعویض الگو و تعویض چنگال اشاره کرد. اندازه کوتاه این نوع از تنوع تعداد تکرارها، احتمال خطای پلیمراز را با افزایش احتمال اشتباه در الگوشناسی بالا میبرد و در نتیجه منجر به تکرار بیشتر این نواحی میشود.[۷] به علاوه، اگر این ناحیههایی سه نوکلئوتیدی در ناحیه کدینگ یک ژن باشد، این مسئله ممکن است منجر به زنجیره طولانی از آمینواسیدهای یکسان در سلول میشود و اگر در ناحیهٔ غیر کدینگ ژن باشد ممکن است در فرایند بیان و تنظیم ژنها مؤثر باشد. از سوی دیگر، تنوع در تعداد تکرار یک ژن در ژنوم کمتر شناخته شدهاست. یک مثال از تکرار ژنها مربوط به ژن AMY1 است که دارای تنوع معنادار میان تعداد تکرارهای ژن در جمعیت با رژیمهای مختلف غذایی است. به علاوه، مکانیزم خاصی که منجر به کاهش و افزایش تعداد تکرارهای این ژن میشود هنوز موضوع مطالعات است، برخی فرضیهها بیان میکنند که مسیر NHEJ یا MMEJ مسئول ایجاد این تکرارهای در طی کل ژنوم است. تکرارهای یک ژن کامل اثر زیادی بر روی بیان آن ژن میگذارد. تأثیر مستقیم تنوع در تعداد تکرار ژن AMY1 در نوع رژیم غذایی گواهی بر این ادعا است. اگرچه این دستهبندی، کلیترین دستهبندی است که میتوان تعداد تکرارها را در آن قرار داد و تعداد دقیق نوکلئوتیدهایی که دچار تکرار با تعداد متفاوت در گونههای مختلف میشوند وابسته مکانی از ژنوم است که در آن قرار گرفتند. در حال حاضر، با استفاده از دادهای که از کل تنوع در تعداد تکرارها موجود است، میانگین اندازه آنها 118kb و میانه آن 18kb به دست آمدهاست.[۸]
تشخیص و شناسایی[ویرایش]
در ابتدا، بنابر مشاهدات سیتوژنتیک تصور میشد که تنوع در تعداد تکرارها در نواحی کوچک و ناچیزی از ژنوم رخ میدهند. تنوع تعداد تکرارها اغلب وابسته به تکرارهای پشت سر هم یا اختلالات ژنتیکی به نظر میآمدند و در نتیجه تنوع تعداد کپی در یک مکان خاص مورد بررسی قرار میگرفت. با این وجود، موفقیتهای به دست آمده در دهههای گذشته منجر به افزایش تعداد راههای شناسایی با دقت بالا در زمینه تکرارهای در طول ژنوم شدهاست. تنوع تعداد تکرارها در ابتدا به کمک روشهای سیتوزنتیک مطالعه میشدند که اطلاعاتی را در مورد ساختار فیزیکی آنها در اختیار میگذاشتند. یکی از این تکنیکها، هیبریداسیون موضعی فلورسنت (FISH) است که شامل قرار دادن پروبهای فلورسنت است که برای اتصال به سطح بالایی از مکمل در ژنوم نیاز دارند. همبند سازی ژنوم مقایسه ای نیز برای شناسایی تنوع در تعداد تکرارها با تجسم فلوروفور و سپس مقایسه طول کروموزومها مورد استفاده قرار گرفت. یکی از نکات مهم این تکنیکهای اولیه این است که وضوح ژنوم کم است و تنها تکرارهای بزرگ مانند تکرار یک ژن کامل قابل شناسایی است.
مکانیزم ملکولی[ویرایش]
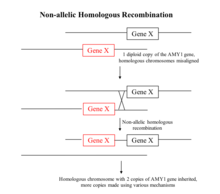
جالبتر از شناسایی تنوع تعداد تکرارها نحوه شکلگیری آنها در طول زنجیره ملکولی است. اولین نکتهای که در مواجهه با مکانیزمهای ارائه شده به نظر میآید این است که اغلب حدس و گمانهزنی هستند. به علاوه، هنوز هیچ گواه قطعی که یک تنوع در تعداد تکرار را به یک مانیزم خاص نسبت دهد وجود ندارد. بهطور کلی دو دسته مکانیزم ملکولی برای تشکیل تنوع تعداد تکرار مطرح است: همسان و غیر همسان.[۵]
یکی از شناختهشدهترین تئوریهایی که منجر به تنوع تعداد تکرار، درج و حذف میشود، نوترکیبهای همسان غیر آللی است.[۹] در طی بازسازی میتونی، کروموزمهای همسان یک سری کروموزومهای جفت با نقاط اتصال خاصی را تشکیل میدهند. با این حال، در طی یک مکانیزم ناپایدار، در طول شکلگیری این نقاط اتصال، گپ دو رشتهای به صورت منظم و ر موقعیتهای غیر آللی وجود دارد. سپس با امکانسازی عبور مواد شیمیایی میان دو کروموزوم یکسان قسمتی از ژنوم در این دو کروموزم همسان تکرار میشود. به همین دلیل، نواحی با تکرار بیشتر و مجزا، نواحی هستند که به صورت مستقل تکرار شدهاند. نوع دیگری از مکانیزم نوترکیبسازی همسان که میتواند منجر به تغییر در تعداد تکرار شود، تکرار ناشی از شکست نام دارد. هنگامی که یک شکست دوگانه در ژنوم اتفاق میافتد، سلول بهطور غیرمنتظره مسیرهایی را که بین این شکستها دچار اختلال شدهاند را فعال میکند. اشتباهات در تعمیر شکاف مانند نوترکیبسازی هلیوژنتیک غیر آللها، میتواند منجر به افزایش تعداد تکرارهای یک منطقه خاص از ژنوم شود. در طول تعمیر شکست دو رشته، انتهای شکسته میتواند به جای بازگشت به رشته اصلی به کروموزوم همولوگ آن حمله کند. همانند مکانیزم نوترکیبسازی همسان، یک نمونه اضافه به ناحیهٔ خاصی از کروموزوم انتقال مییابد که منجر به بروز تکرار میشود. به علاوه، پروتئینهای کوهسین به بازسازی شکستگیهای دوگانه با بستن دو طرف آن و جلوگیری از نفوذ به سایر نقاط کروموزوم، کمک میکنند. اگر به هر دلیلی مانند فعال شدن RNA ریبوزومی عملکرد کوهسین تحت تأثیر قرار بگیرد، یک افزایش در بازسازی خطا به صورت محلی ممکن است صورت گیرد.[۱۰]
ژنهای همخانواده و انتخاب طبیعی[ویرایش]

در سالهای اخیر، در رابطه با احتمال وجود ارتباط میان ژنهای همخانواده و تنوع تعداد تکرارها بحثهایی شدهاست. ژنهای همخانواده، مجموعهای از ژنهای مرتبط هستند که دارای عملکردهای مشابه و اختلافهای زمانی یا فضایی جزئی هستند و به احتمال زیاد از یک ژن اجدادی مشترک حال شدهاند. اصلیترین انگیزه برای وجود ارتباط میان تنوع تعداد تکرار و این مفهوم این است که به احتمال زیاد ژنهای یک خانواده از یک ژن اجدادی که در نسخههای مختلف کپی شدهاست، به وجود آمدهاند. جهشها در طول زمان بر روی ژنها ایجاد میشوند و آن دسته از ژنهایی که با طبیعت سازگار هستند در اثر انتخاب طبیعی باقیمانده و خانوادهای از ژنها را ایجاد میکنند. یک نمونه از خانواده ژنی که ممکن است به دلیل تنوع تعداد تکرار ایجاد شده باشد، خانواده گلوبین است. خانواده گلوبین یک شبکه دقیق از ژنهای متشکل از ژنهای آلفا و بتا گلوبین است که شامل ژنهایی هستند که در جنینها و بزرگسالان بیان میشوند. ژنهای گلوبین در خانواده گلوبین به خوبی ویژگیهای مشترک خود را حفظ کردهاند و تنها در بخش کوچکی متفاوت هستند که نشاندهندهٔ این نکته است که از یک جد مشترک به دست آمدهاند.
تحقیقات نشان دادهاست که تغییرات تعداد تکرارها در ژنهایی که منشأ پروتئینهایی هستند که با محیط در تعامل اند بیشتر از ژنهایی است که پروتئینهای آنها در فعالیتهای سلولی پایه هستند.[۱۱] علاوه بر این، پروتئینها با یکدیگر کار میکنند و با پروتئینهای مسیرهای دیگر همکاری میکنند، بنابراین بررسی اثر انتخاب طبیعی بر روی مسیرهای زیست مولکولی مهمتر از بررسی پروتئینهای فردی است. با این گفته میشود که پروتئینها در حاشیه مسیر در تغییرات تعداد نسخهها غنی شدهاند، در حالی که پروتئینها در مرکز مسیرها در تغییرات تعداد کپی هستند. تحقیقات نشان دادهاست که پروتئینهای در حاشیه مسیر ارتباط کمتری با پروتئینهای دیگر دارند و بنابراین تغییر در مقدار بیان آنها اثر کمتری بر روی نتیجه یک مسیر زیست ملکولی دارد.[۱۲]
در سالهای اخیر، محققان تمرکز خود را از شناسایی، مکانیابی و توالییابی تنوع تعداد تکرار به تجزیه و تحلیل نقش این تغییرات در ژنوم انسان و طبیعت تغییر دادهاند. همچنان، شواهدی برای اثبات ارتباط بین تنوع تعداد تکرار و خانوادههای ژنی و همچنین نقش انتخاب طبیعی در شکلدادن این روابط و تغییرات نیاز است. به علاوه، محققان همچنان به دنبال کشف مکانیزمهای مولکولی درگیر در ایجاد تنوع تعداد تکرار هستند که ممکن است اطلاعات ضروری دربارهٔ تغییرات ساختاری را دربرداشته باشند. این دادههای تحقیقاتی نه تنها شواهد اضافی برای تکامل و انتخاب طبیعی را فراهم میکنند، بلکه میتوانند برای درمان انواع مختلف بیماریهای ژنتیکی مورد استفاده قرار گیرند.
جستارهای وابسته[ویرایش]
منابع[ویرایش]
- ↑ ۱٫۰ ۱٫۱ Mccarroll, S. A. ; Altshuler, D. M. (۲۰۰۷). Copy-number variation and association studies of human diseases. Nature Genetics. صص. ۳۷–۴۲.
- ↑ Sharp, A. J. ; Locke, D. P. ; Mcgrath, S. D. ; Cheng, Z; Bailey, J. A. ; Vallente, R. U. ; Pertz, L. M. ; Clark, R. A. ; Schwartz, S. ; Segraves, R. (۲۰۰۵). Segmental Duplications and Copy-Number Variation in the Human Genome. The American Journal of Human Genetics. صص. ۷۸–۸۸.
- ↑ Taniguchi, Y. ; Choi, P. J. ; Li, J. W. ; Chen, H. ; Babu, M. ; Hearn, J. ; Emili, A. ; Xie, X. S. (۲۰۱۰). Quantifying E. coli Proteome and Transcriptome with Single-Molecule Sensitivity in Single Cells. Science. صص. ۵۳۳–۵۳۸.
- ↑ Koning, A. P. J. D. ; Gu, W. ; Castoe, T. A. ; Bazter, M. A. ; Pollock, D. D. (۲۰۱۱). Repetitive Elements May Comprise Over Two-Thirds of the Human Genome. PLOS Genetics. صص. ۱۲.
- ↑ ۵٫۰ ۵٫۱ Hastings, P. J. ; Lupski, J. R. ; Roseberg, S. M. ; Ira, G. (۲۰۰۹). Mechanisms of change in gene copy number. Nature Reviews Genetics. صص. ۵۵۱–۵۶۴.
- ↑ ۶٫۰ ۶٫۱ Macdonald, M. ; Ambrose, C. M. ; Duyao, M. P. ; Myers, R. H. ; Lin, C. ; Srinidhi, L. ; Barnes, G. ; Taylor, S. A. ; James, M. ; Groot, N. (۱۹۹۳). A novel gene containing a trinucleotide repeat that is expanded and unstable on Huntington's disease chromosomes. Cell. صص. ۹۷۱–۹۸۳.
- ↑ On the formation of spontaneous deletions: The importance of short sequence homologies in the generation of large deletions (۱۹۸۲). Albertini, A. M. ; Hofer, M. ; Calos, M. P. ; Miller, J. H. Cell. صص. ۳۱۹–۳۲۸.
- ↑ Freeman, J. L (۲۰۰۶). Copy number variation: New insights in genome diversity. Genome Research. صص. ۹۴۹–۹۶۱.
- ↑ Pagues, F; Haber, J. E. (۱۹۹۹). Multiple pathways of recombination induced by double-strand breaks in Saccharomyces Cerevisiae. Microbiology Review. صص. ۳۴۹–۴۰۴.
- ↑ Kobayashi, T. ; Ganley, A. R. (۲۰۰۵). Recombination Regulation by Transcription-Induced Cohesin Dissociation in rDNA Repeats. Science. صص. ۱۵۸۱–۱۵۸۴.
- ↑ Redon, R. ; Ishikawa, S. ; Fitch, K. R. ; Feuk, L. ; Perry, G. H. ; Andrew, T. D. ; Fiegler, H. ; Shapero, M. H. ; Carson, A. R. ; Chen, W. (۲۰۰۶). Global variation in copy number in the human genome. Nature. صص. ۴۴۴–۴۵۴.
- ↑ Kim, P. M. ; Korbel, J. O. ; Gerstein, M. B. (۲۰۰۷). Positive selection at the protein network periphery: Evaluation in terms of structural constraints and cellular context. Proceedings of the National Academy of Sciences. صص. ۲۰۲۷۴–۲۰۲۷۹.
